 A- и В- формы ДНК. Структура РНК
A- и В- формы ДНК. Структура РНК A- и В- формы ДНК. Структура РНК
A- и В- формы ДНК. Структура РНКПри помощи программы fiber пакета 3DNA были построены модели структур A-, B- и Z-формы ДНК. У A- и B- формы последовательность одной из нитей представляет собой 5 раз повторенную последовательность "gatc".
На рисунках 1, 2 и 3 показаны полученные структуры.

Рисунок 1. A-форма ДНК.

Рисунок 2. B-форма ДНК.
Рисунок 3. Z-форма ДНК.
На рисунке 4 показана А-форма ДНК, оранжевым выделен сахарофосфатный остов, синим - аденин, остальные азотистые основания - зелёным, красным показаны атомы N7 во всех гуанинах.
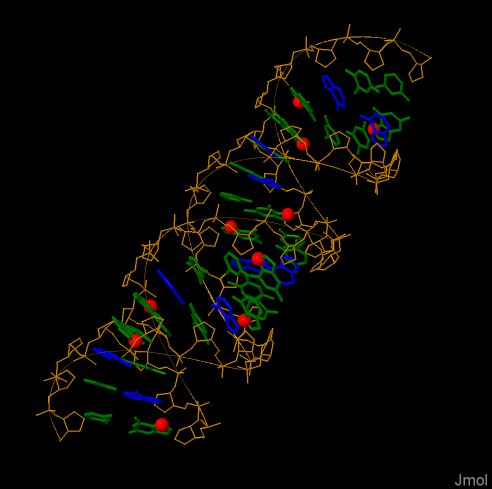
Рисунок 4. Структура ДНК.
Далее были скачаны PDB-файлы: 1D5Y и 1EHZ. В данных структурах отсутствуют разрывы цепи нуклеиновых кислот. Для дальнейшей работы с этими файлами в структурах были оставлены только цепи ДНК и РНК. Полученные файлы: 1D5Y-dna.pdb и 1EHZ-rna.pdb.
Далее нужно было выбрать аденин в любом месте цепи ДНК В-формы и определить, какие атомы основания обращены в сторону большой бороздки, а какие в сторону малой. Был выбран аденин под номером 30. На рис.5 показано изображение аденина, полученное при помощи ChemSketch, красным помечены атомы, обращённые в сторону большой бороздки (A30.C6, A30.N6, A30.C5 и A30.N7), синим - в сторону малой (A30.C2, A30.N3, A30.C4, A30.N9), оставшиеся два атома находятся посередине. На рис.6 показан аденин в структуре ДНК.
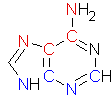
Рисунок 5. Аденин.
Рисунок 6.
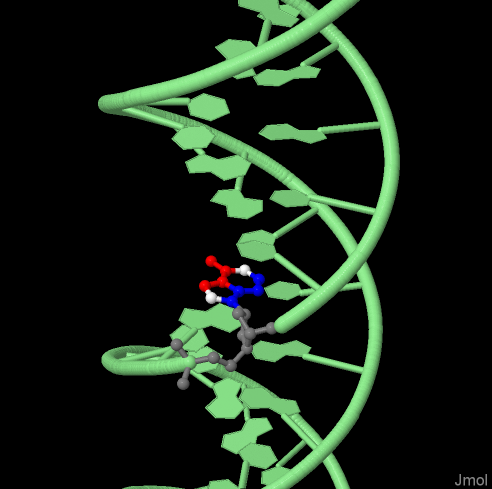
Рисунок 7. Аденин в структуре ДНК. Красным помечены атомы, обращённые в сторону большой бороздки, синим - в сторону малой, два атома, находящиеся посередине, помечены белым.
В структуре ДНК А-формы ориентация атомов аденина такая же. ДНК Z-формы не содержит аденинов.
Далее были сравнены спиральные параметры трёх разных форм ДНК. Полученные данные показаны в таблице 1.
|
A-форма |
B-форма |
Z-форма |
Тип спирали |
Правая |
Левая |
Левая |
Шаг спирали (A) |
28.05 |
33.80 |
43.56 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
7.96 |
17.21 |
7.20 |
Ширина малой бороздки |
16.61 |
11.69 |
17.60 |
Таблица 1.
В таблице 2 показаны значения торсионных углов аденина.
| α (O3'–P–O5'–C5') | β (O5'–C5') | γ (O5'–C5'–C4'–C3') | δ (C4'–C3') | ε (C4'–C3'–O3'–P) | ζ (O3–P) | χ (O4'–C1'–N9–C4) | |
| А-ДНК | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-ДНК | -60.0 | -163.4 | 31.1 | 143.4 | -140.8 | -160.6 | -98.0 |
Таблица 2.
С помощью программ find_pair и analyze были полученны файлы с информацией о нуклеиновых кислотах в структурах 1D5Y и 1EHZ.
В таблице 3 указаны средние значения торсионных углов для данных структур.
| α | β | γ | δ | ε | ζ | χ | |
| 1D5Y (ДНК) | -53,6 | 4,5 | 45 | 140,1 | -12,6 | -117,9 | -107,2 |
| 1EHZ (РНК) | -52,2 | 72,6 | 56,8 | 87,7 | -147,8 | -75 | -151,3 |
Таблица 3.
Наибольшие отклонения от среднего значения по углам имеет аденин 27 в РНК (γ, ε и ζ).
Далее нужно было определить структуру водородных связей в тРНК.
Участок 1-7 и комплементарный ему участок 66-72 убразуют акцепторный стебель:
(0.008) A:...1_:[..G]G-----C[..C]:..72_:A (0.006) (0.006) A:...2_:[..C]C-----G[..G]:..71_:A (0.013) (0.021) A:...3_:[..G]G-----C[..C]:..70_:A (0.018) (0.018) A:...4_:[..G]G-*---U[..U]:..69_:A (0.015) (0.018) A:...5_:[..A]A-----U[..U]:..68_:A (0.011) (0.011) A:...6_:[..U]U-----A[..A]:..67_:A (0.020) (0.008) A:...7_:[..U]Ux----A[..A]:..66_:A (0.016)
T-стебель состоит из участков 49-53 и 61-65:
(0.020) A:..49_:[5MC]c-----G[..G]:..65_:A (0.017) (0.024) A:..50_:[..U]U-----A[..A]:..64_:A (0.014) (0.030) A:..51_:[..G]G-----C[..C]:..63_:A (0.016) (0.020) A:..52_:[..U]U-----A[..A]:..62_:A (0.016) (0.027) A:..53_:[..G]G----xC[..C]:..61_:A (0.012)
D-стебель из 10-13 и 22-25:
(0.018) A:..10_:[2MG]g-----C[..C]:..25_:A (0.011) (0.005) A:..11_:[..C]C-----G[..G]:..24_:A (0.013) (0.012) A:..12_:[..U]U-----A[..A]:..23_:A (0.030) (0.013) A:..13_:[..C]C----xG[..G]:..22_:A (0.017)
Антикодоновый стебель из 39-44 и 26-31:
(0.004) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.007) (0.005) A:..40_:[5MC]c-----G[..G]:..30_:A (0.013) (0.013) A:..41_:[..U]U-----A[..A]:..29_:A (0.019) (0.010) A:..42_:[..G]G-----C[..C]:..28_:A (0.007) (0.025) A:..43_:[..G]G-----C[..C]:..27_:A (0.011) (0.015) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.013)
Остальные водородные связи стабилизируют третичную структуру тРНК:
(0.013) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.012) (0.024) A:..55_:[PSU]Px**+xG[..G]:..18_:A (0.028) (0.025) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.016) (0.021) A:..15_:[..G]G-**+xC[..C]:..48_:A (0.015) (0.188) A:..16_:[H2U]ux**+xU[..U]:..59_:A (0.029) (0.026) A:..19_:[..G]G-----C[..C]:..56_:A (0.014)
Присутвуют следующие неканонические пары оснований: 4G-69U, 39P-31A, 44A-26G.
В следующем задании было необходимо найти возможные стекинг-взаимодействия в тРНК.
Ниже представлены данные, полученные из файла 1EHZ.out:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GC/GC 5.19( 2.33) 0.00( 0.00) 0.00( 0.00) 6.59( 3.41) 11.78( 5.74) 2 CG/CG 0.51( 0.00) 0.00( 0.00) 2.98( 0.46) 0.71( 0.00) 4.20( 0.46) 3 GG/UC 2.74( 1.28) 0.00( 0.00) 0.01( 0.00) 0.30( 0.00) 3.05( 1.28) 4 GA/UU 4.55( 2.72) 0.00( 0.00) 0.00( 0.00) 2.78( 0.92) 7.34( 3.64) 5 AU/AU 4.86( 3.47) 0.00( 0.00) 0.00( 0.00) 3.00( 1.72) 7.86( 5.18) 6 UU/AA 0.50( 0.00) 0.00( 0.00) 0.00( 0.00) 3.45( 3.11) 3.94( 3.11) 7 Uc/GA 0.98( 0.00) 0.00( 0.00) 0.00( 0.00) 3.08( 1.54) 4.06( 1.54) 8 cU/AG 0.00( 0.00) 0.00( 0.00) 0.40( 0.00) 3.43( 2.87) 3.83( 2.87) 9 UG/CA 0.31( 0.00) 0.00( 0.00) 2.45( 1.12) 0.22( 0.00) 2.99( 1.12) 10 GU/AC 5.87( 3.10) 0.00( 0.00) 0.00( 0.00) 4.01( 2.38) 9.88( 5.48) 11 UG/CA 0.00( 0.00) 0.00( 0.00) 4.02( 1.93) 0.00( 0.00) 4.02( 1.93) 12 Gu/aC 8.58( 3.94) 0.00( 0.00) 0.00( 0.00) 3.56( 0.68) 12.14( 4.63) 13 uP/Ga 6.08( 1.82) 0.00( 0.00) 0.00( 0.00) 8.21( 3.27) 14.29( 5.10) 14 PA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 15 AA/cU 2.73( 0.95) 0.00( 0.00) 0.00( 0.00) 4.92( 2.25) 7.66( 3.20) 16 AP/Ac 5.04( 3.17) 0.00( 0.00) 0.00( 0.00) 6.30( 3.92) 11.34( 7.08) 17 Pc/GA 3.17( 0.17) 0.00( 0.00) 0.00( 0.00) 6.10( 4.17) 9.28( 4.34) 18 cU/AG 0.00( 0.00) 0.00( 0.00) 0.70( 0.00) 1.74( 1.71) 2.44( 1.71) 19 UG/CA 0.00( 0.00) 0.00( 0.00) 2.99( 1.72) 0.00( 0.00) 2.99( 1.72) 20 GG/CC 2.53( 1.04) 0.00( 0.00) 0.27( 0.00) 0.15( 0.00) 2.94( 1.04) 21 GA/gC 3.12( 0.91) 0.00( 0.00) 0.00( 0.00) 4.81( 2.13) 7.93( 3.04) 22 Ag/Cg 0.00( 0.00) 0.00( 0.00) 1.02( 0.00) 0.40( 0.07) 1.42( 0.07) 23 gC/GC 3.53( 0.73) 0.00( 0.00) 0.00( 0.00) 7.32( 4.35) 10.85( 5.08) 24 CU/AG 1.03( 0.01) 0.00( 0.00) 0.00( 0.00) 2.64( 2.33) 3.67( 2.35) 25 UC/GA 1.53( 0.43) 0.00( 0.00) 0.00( 0.00) 1.09( 0.09) 2.62( 0.52) 26 CA/UG 0.00( 0.00) 1.92( 0.00) 2.31( 0.19) 0.00( 0.00) 4.23( 0.19) 27 AG/CU 2.06( 0.54) 0.00( 0.00) 0.25( 0.00) 0.00( 0.00) 2.31( 0.54) 28 Gu/UC 3.09( 1.19) 1.91( 0.00) 0.00( 0.00) 0.00( 0.00) 5.00( 1.19) 29 uG/CU 0.00( 0.00) 0.00( 0.00) 1.39( 0.19) 0.16( 0.00) 1.55( 0.19)
Здесь указана величина площади "перекрываний" 2-х последовательных пар азотистых оснований. Наибольшее значение у тринадцатой пары.
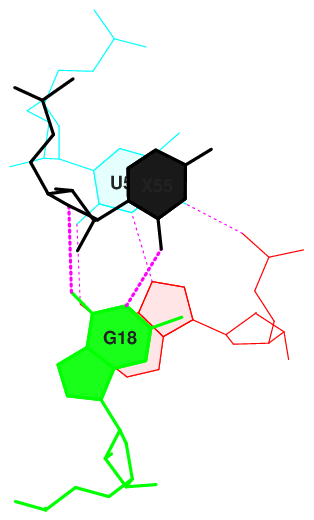
Рисунок 8. Стекинг - взаимодействия в структуре т-РНК, шаг 13..