Занятие 6.
Сегодня мы будем изучать как реализован контроль температуры в молекулярной динамике на примере GROMACS. Объект исследования это одна молекула этана.1.Начнем с того, что подготовим файл координат и файл топологии. В прошлом занятии нам был предоставлен gro файл с 38 молекулами этана. Создадим индекс файл котором будет группа из одной молекулы этана.
make_ndx -f box_38.gro -o 1.ndxПосле запуска команды у Вас появится приглашение к вводу. Сначала ознакомитесь с помощью нажав "h" + enter. Выберите остаток номер 1. Нажмите enter и вы увидите, что появилась новая группа. Теперь создадим gro файл с одной молекулой и зададим ячейку . При запуске ediconf выберем номер соответствующей группе из одной молекулы.
editconf -f box_38.gro -o et1.gro -n 1.ndx #зададим ячейку и расположим молекулу по центру ячейку editconf -f et1.gro -o et.gro -d 2 -cПолучили et.gro Исправим файл топологии et.top из прошлого задания. В разделе [ molecules ] измените количество молекул этана. et2.top
2.Нам даны 5 файлов с разными параметрами контроля температуры:
be.mdp - метод Берендсена для контроля температуры. vr.mdp - метод "Velocity rescale" для контроля температуры. nh.mdp - метод Нуза-Хувера для контроля температуры. an.mdp - метод Андерсена для контроля температуры. sd.mdp - метод стохастической молекулярной динамики.
3. Сначала надо построить входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd см. выше список mdp файлов
4.Получилось 5 tpr файлов. Теперь для каждого из них запустим mdrun.
mdrun -deffnm et_${i} -v -nt 1
5.Теперь переходим к анализу результатов. Начнем с визуального анализа. Для каждой из 5 систем проведите конвертацию в pdb и просмотрите в PyMol.
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
В методе Андерсена молекула практически неподвижна. В методе стохастической молекулярной динамики молекула
вращается очень-очень быстро. Методы Берендсена и "Velocity rescale" похожи: молекула постепенно раскручивается
все быстрее и быстрее. В методе Нуза-Хувера молекула вращается как волчок (атомы водорода), а атомы углерода
почти не двигаются.
6.Сравним потенциальную энергию связи и кинетическую энергию для каждой из 5 систем.
g_energy -f et_${i}.edr -o et_${i}_en.xvg
Построим графики изменения энергий. Рекомендуемый вид это dot-plot.
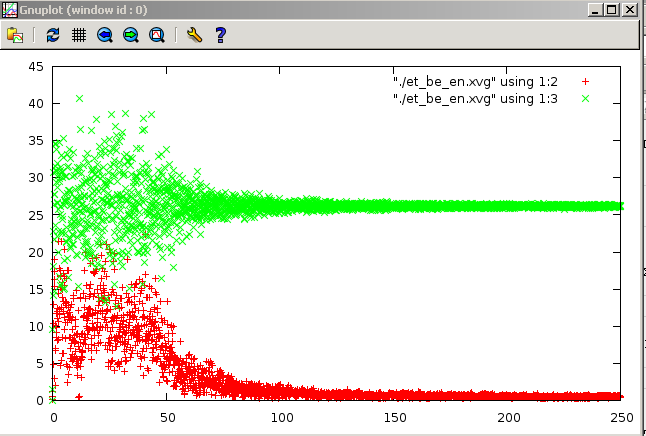
метод Берендсена:
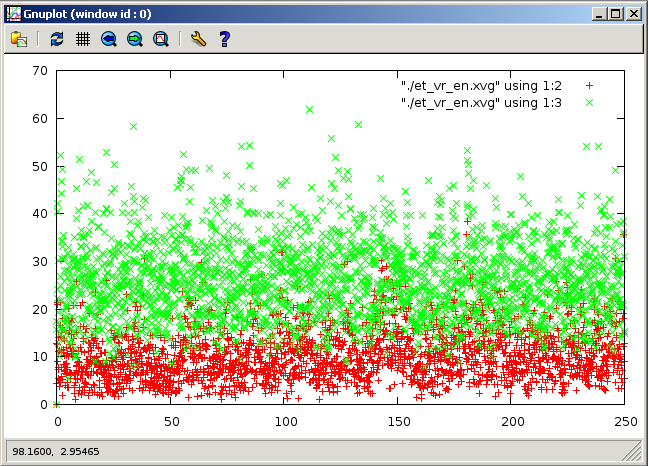
метод "Velocity rescale":
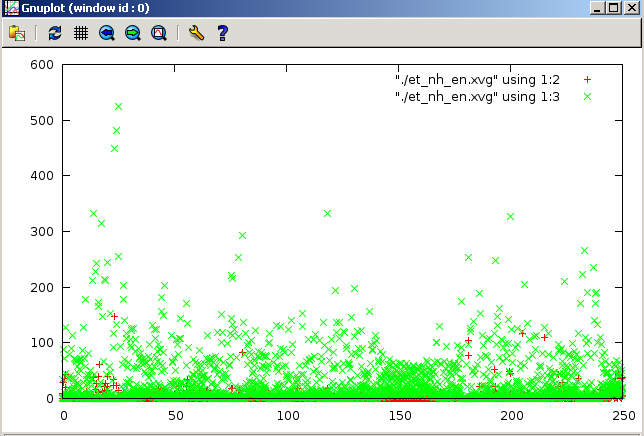
метод Нуза-Хувера:
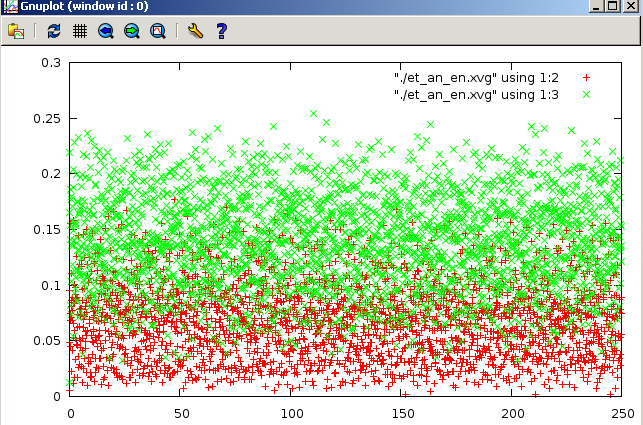
метод Андерсена:
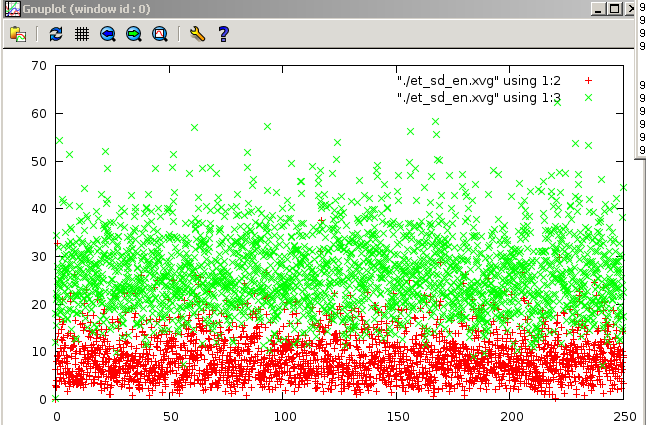
метод стохастической молекулярной динамики:
7.Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. В текстовом редакторе создайте файл b.ndx со следующим содержимым:
[ b ] 1 2И запустим утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
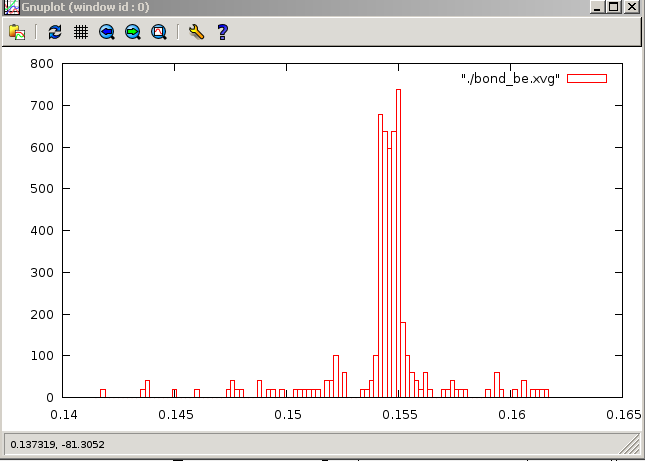
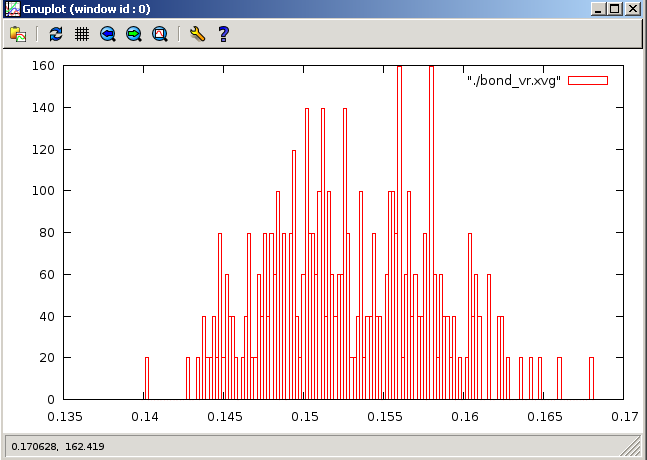
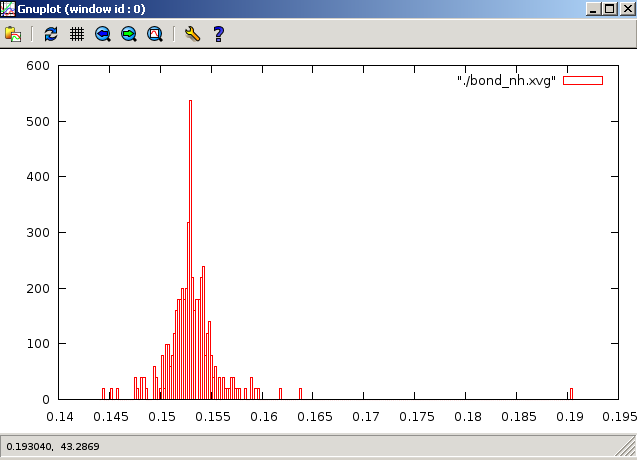
Постройте графики распределения длинн связей. Рекомендуемый вид это гистограмма или boxes в Gnuplot.
метод Берендсена:
метод "Velocity rescale":
метод Нуза-Хувера:
метод Андерсена:
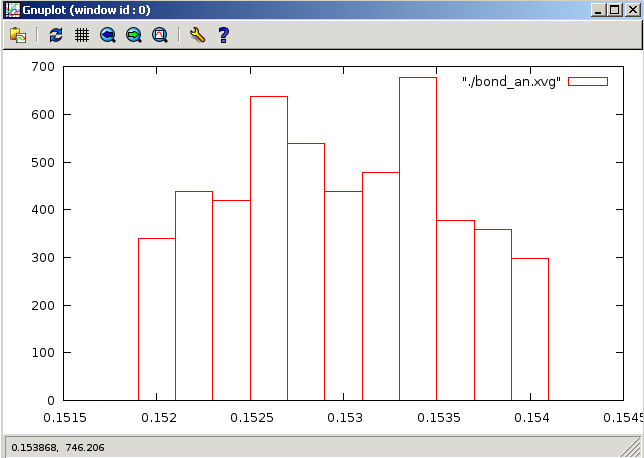
метод стохастической молекулярной динамики:
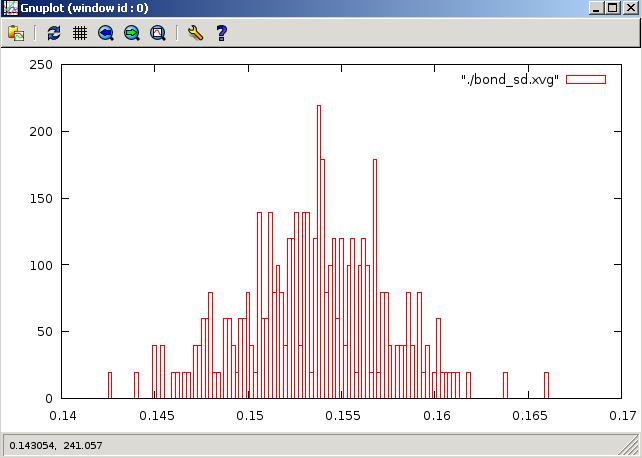
8.Форме распределения Больцмана вполне соответствуют графики методов Нуза-Хувера и стохостической молекулярной динамики. На рисунках зеленым цветом обозначена кинетическая энергия. Высокая энергия - это удобство термостата. Чем больше она по оси OY, тем лучше термостат, который дает эту энергию молекуле, чтобы "скатить" ее в минимум (больше всего у Нуза-Хувера). Из наблюдений можно сделать вывод о том, что метод Нуза-Хувера позволяет наиболее реалистично поддерживать температуру в системе. Методы "Velocity rescale", стохастической молекулярной динамики на втором месте. Метод Андерсена совсем плоховат.