Занятие 2. Программы DSSP и HBplus.
Получим файл с записью PDB (PDB-код 1DBP).
Определим вторичную структуру молекул белка, представленных в соответствующей
записи, программой DSSP(скачаем уже готовый результат по FTP с ftp://ftp.cmbi.ru.nl//pub/molbio/data/dssp/).
1DBP.dssp
Выходной файл (в данном случае 1DBP.dssp) импортируем в Excel.
1DPB.dssp в Excel
Для A цепи белка:12 бета-тяжей, 9 альфа-спиралей
Откроем PDB-файл в редакторе и изучим поля HELIX и SHEET.
helix_sheet.txt
Сравним два описания вторичной структуры (вычисленное DSSP и приведённое
авторами записи). Имеются несовпадения номеров остатков.
HELIX В PDB:105-120; 191-204; 237-252, а в DSSP: 105-119; 191-203; 237-254.
SHEET В PDB: 60-63;184-188; 231-235 ,а в DSSP: 58-63; 183-188; 231-234
Проанализируем колонку PHI выдачи DSSP. Перечислим остатки, имеющие
положительное значение угла φ
(лизин 1 цепи А не учитываем, т.к. его значение угла &phi=360, а &phi в пределах от -180 до +180)
глицин 31 цепи А
глутамин 40 цепи А
аспарагин 41 цепи А
глицин 57 цепи А
аспарагин 82 цепи А
аспарагиновая кислота 89 цепи А
глицин 95 цепи А
глицин 121 цепи А
глицин 123 цепи А
лизин 153 цепи А
аспарагиновая кислота 163 цепи А
фенилаланин 164 цепи А
глицин 205 цепи А
аспарагиновая кислота 215 цепи А
глицин 216 цепи А
глицин 227 цепи А
глицин 254 цепи А
глутамин 271 цепи А
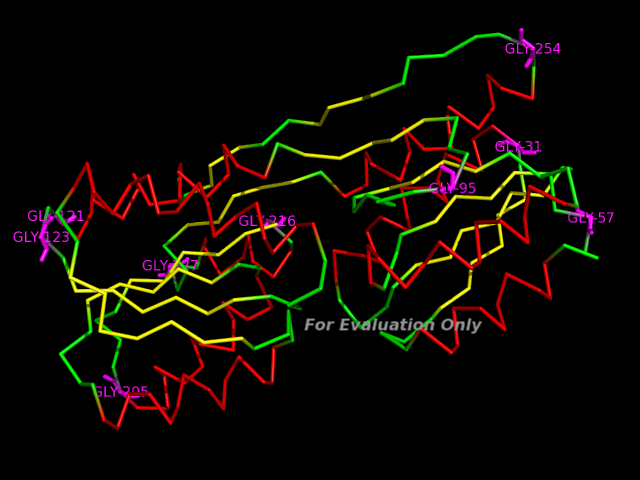
Остовная модель (ribbon) цепи А.
Красным цветом выделены альфа-спирали, желтым- бета-тяжи, розовым-глицины с положительным фи .
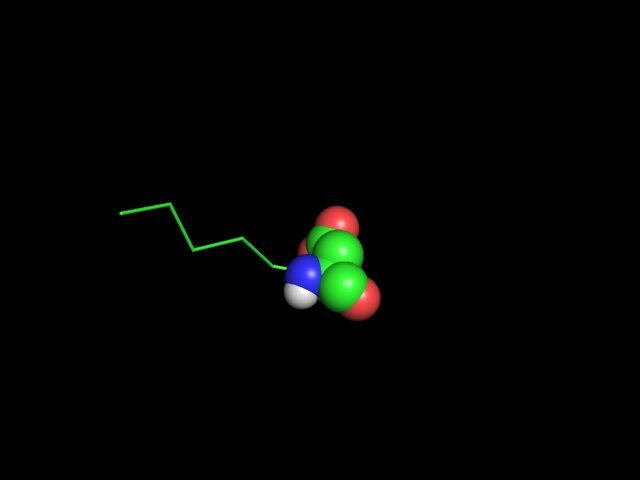
Изображен бета-тяж 210-215 и аспарагиновая кислота 215 с углом PHI >0 (125.4)
 Программой HBPlus определим водородные связи в записи (файл 1DBP.hb2 получим командой hbplus 1XYZ.pdb
).
Программой HBPlus определим водородные связи в записи (файл 1DBP.hb2 получим командой hbplus 1XYZ.pdb
).
Приведем примеры водородных связей
а) участвующих в стабилизации вторичной структуры;
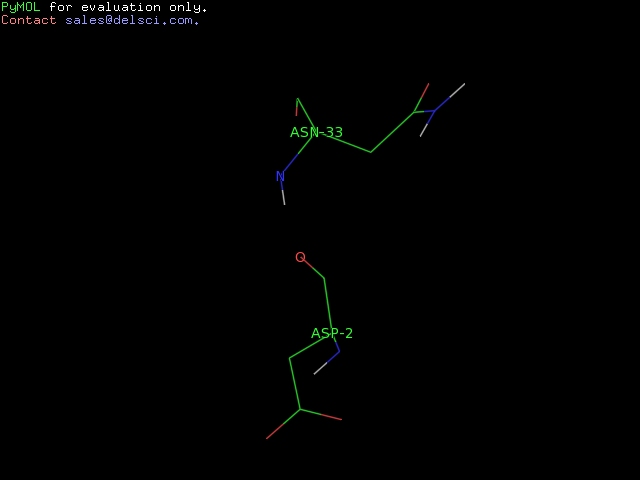 A0033-ASN N A0002-ASP O
A0033-ASN N A0002-ASP O
б) между боковыми цепями аминокислотных остатков;
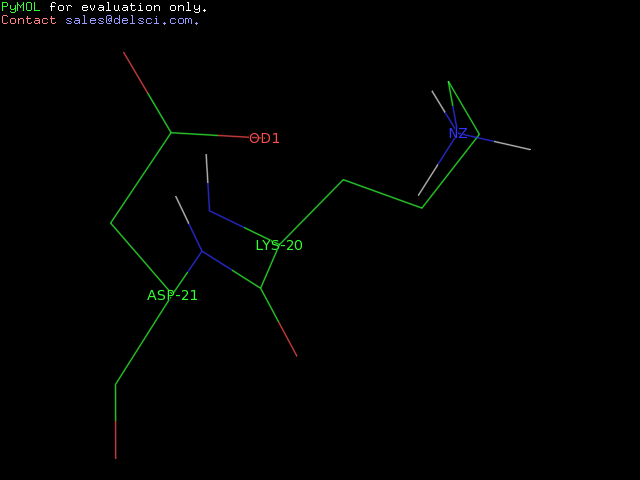 A0020-LYS NZ A0021-ASP OD1
A0020-LYS NZ A0021-ASP OD1
в) между боковой цепью одного остатка и остовным атомом другого.
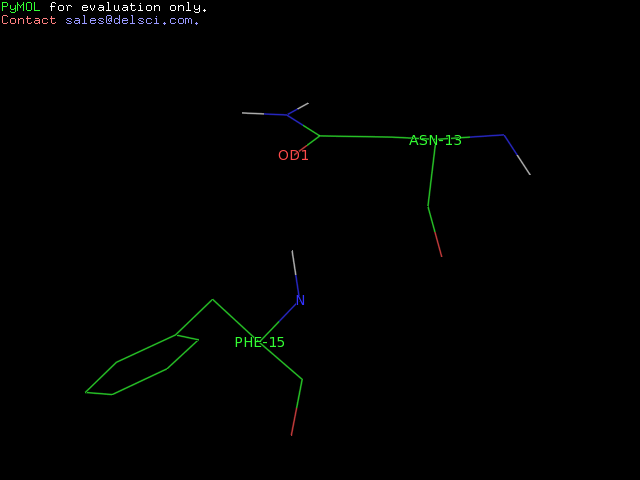 A0015-PHE N A0013-ASN OD1
A0015-PHE N A0013-ASN OD1
Приведем также один пример водяных мостиков
между различными остатками.
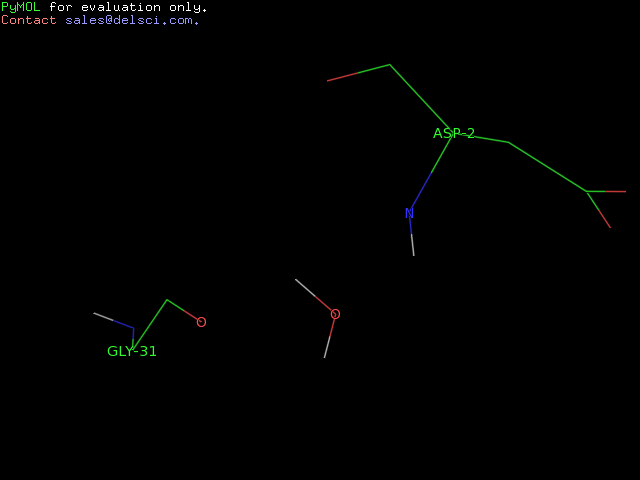 A0002-ASP N A0287-HOH O A0031-GLY O
См. указания.
A0002-ASP N A0287-HOH O A0031-GLY O
См. указания.


 A0033-ASN N A0002-ASP O
A0033-ASN N A0002-ASP O
 A0020-LYS NZ A0021-ASP OD1
A0020-LYS NZ A0021-ASP OD1
 A0015-PHE N A0013-ASN OD1
A0015-PHE N A0013-ASN OD1
 A0002-ASP N A0287-HOH O A0031-GLY O
См.
A0002-ASP N A0287-HOH O A0031-GLY O
См.