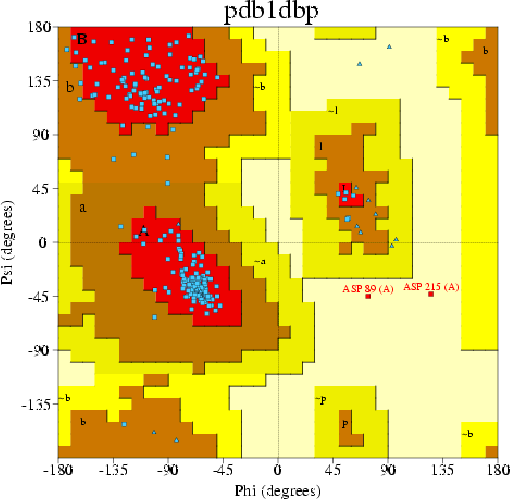
Скопируем в отчёт карту Рамачандрана своей структуры.
No. of
residues %-tage
------ ------
Most favoured regions [A,B,L] 219 92.4%
Additional allowed regions [a,b,l,p] 16 6.8%
Generously allowed regions [~a,~b,~l,~p] 0 0.0%
Disallowed regions [XX] 2 0.8%*
---- ------
Non-glycine and non-proline residues 237 100.0%
End-residues (excl. Gly and Pro) 2
Glycine residues 23
Proline residues 9
----
Total number of residues 271
Видим, что два остатка попали в запрещенную область(белую)- это ASP 89 (A) и ASP 215.
Приведем процент остатков (отличных от Gly и Pro, они обозначены голубими треугольничками), попавших в предпочитаемые области на карте (красная область). 16 остатков, отличных от Gly и Pro, попали в допустимую область, два - в запрещенную. В предпочитаемой - 219 остатков. Таким образом, процент попавших в предпочитаемую область остатков 92,4% Это значение больше 90%, что является хорошим показателем структуры.
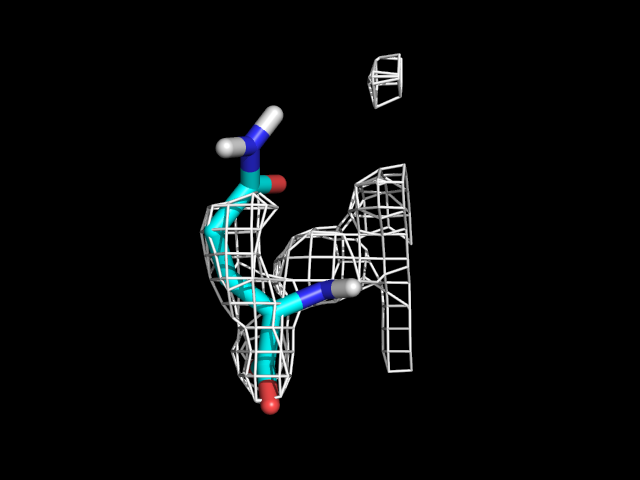
Значения:
пространственного R-фактора (RSR)=0,286,
коэффициента корреляции для электронной плотности ("real-space correlation coefficient") =0,650
Z-score пространственного R-фактора для этого остатка=2,574163.
Простр. R-фактор >10%, а Z-score >0. Это говорит о том, что действительно
расположение остатка плохое (он "выбился").Как видно из картинки, остаток очень плохо
вписался в электронную плотность
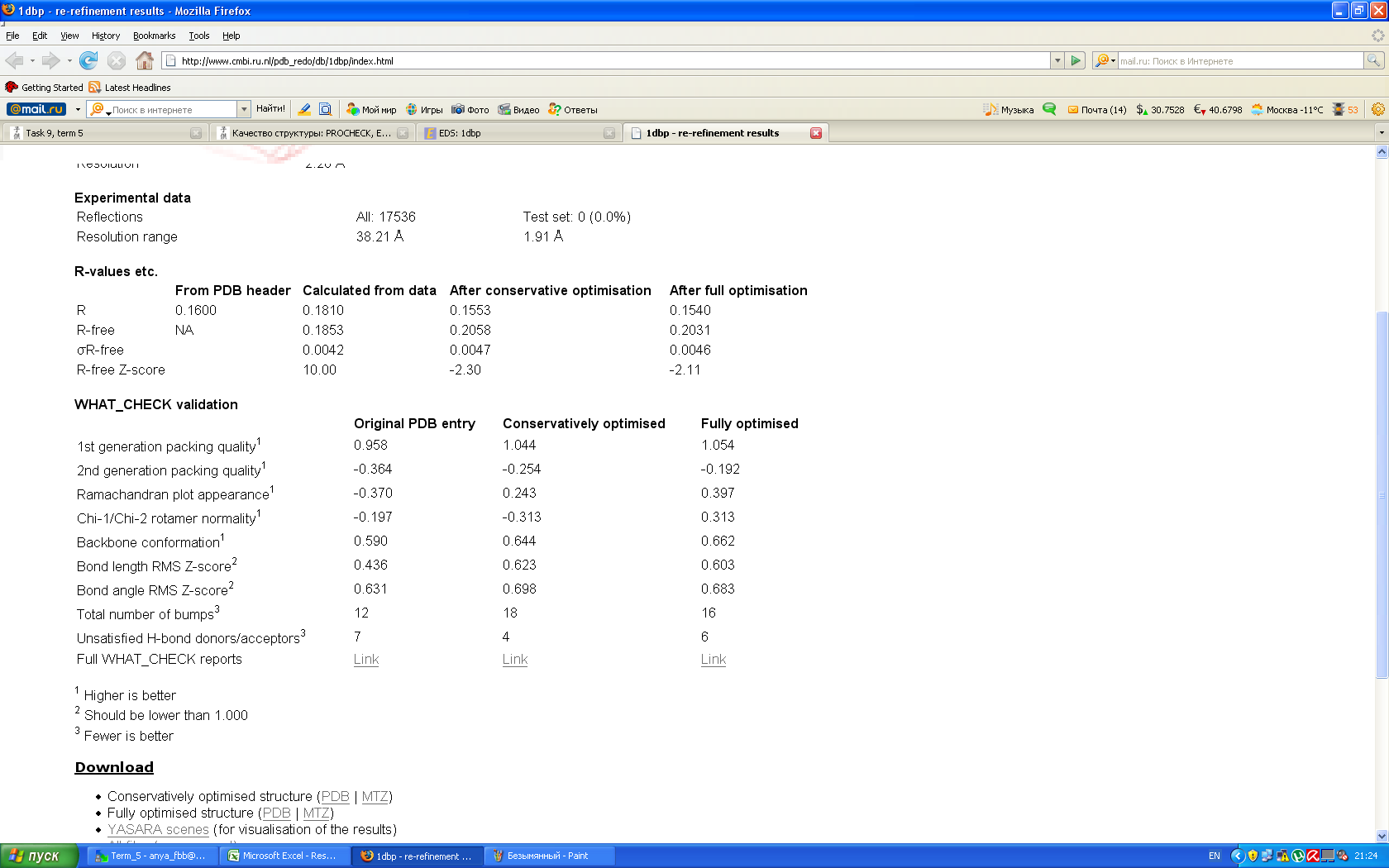
Улучшились показатели: R, R-free, 1st generation packing quality, 2nd generation packing quality, Ramachandran plot appearance, Chi-1/Chi-2 rotamer normality, Backbone conformation, Unsatisfied H-bond donors/acceptors Показатель Z-score ухудшился(он стал ближе к нулю по значению). Значения Bond length RMS Z-score,Bond angle RMS Z-score,Total number of bumps незначительно ухудшились. В Full WHAT_CHECK reports: Error: Matthews Coefficient (Vm) very high