Импортировала в Genedoc множественное выравнивание, полученное на прошлом занятии с помощью muscle : Картинка из GeneDoc myproteins.gif
Выбрала фрагмент выравнивания длиной 13 а.о. для дальнейшего исследования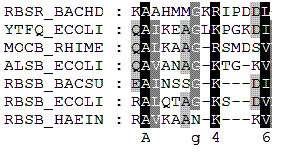
Рассмотрела выбранный мной фрагмент множественного выравнивания. Создала 3 паттерна, записанные в таблицу, см. ниже.
- Первый паттерн в точности является фрагментом последовательности белка RBSB_ECOLI (то есть только одной из последовательностей выравнивания)
- Второй ("сильный") паттерн надо постараться построить так, чтобы он распознавал все белки Вашей выборки, и только их (другой вопрос, что паттерн будет находить в действительности:)
- Третий ("слабый") паттерн надо создать на основе второго, сделав требования к последовательности более мягкими.
Провела поиск последовательностей банка Swiss-Prot, включающих мотивы, соответствующие каждому из полученных паттернов
По результатам упражнения заполнила табличку следующего вида:
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности белка RBSB_ECOLI | RALQTAGKSDV | 1 | из выравнивания найдена лишь последовательность моео белка RBSB_ECOLI |
| Сильный | [KQER]-A-[AILV]-[HKANQ]-[MEANST]-[MAS]-[GN]-X(0,1)-[RK]-X(0,3)-[DSK]-[LIV] | 133 | все |
| Слабый | {AG}-A-[AILV]-[HKANQ]-[MEANST]-[MAS]-[GN]-X(0,1)-[RK]-X(0,3)-[DSK] | 998 | все |