Salmonella typhimurium (SALTY ) - Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica
Escherichia coli (ECOLI) -Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia
Enterobacter sp. 638 (ENT38) - Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Enterobacter
Erwinia carotovora (ERWCT)- Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Pectobacterium
Pseudomonas aeruginosa (PSEAE) - Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group
Neisseria meningitidis (NEIMA) - Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria
Ralstonia pickettii (RALPJ) - Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia
Burkholderia cenocepacia (BURCA)- Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia cepacia complex
Выделим ветви, выделяющие таксоны: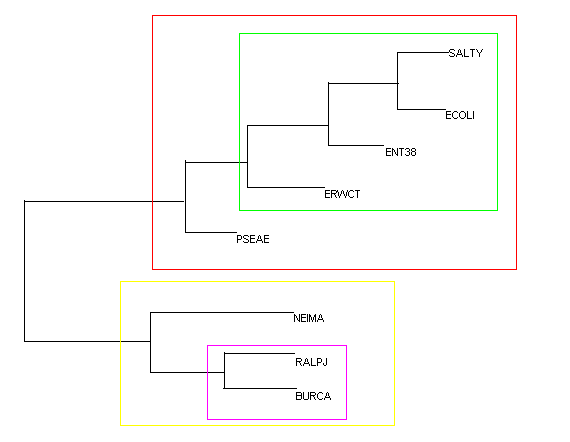
| Ветвь | Таксоны |
| {SALTY,ECOLI,ENT38,ERWCT,PSEAE} | (красный)Bacteria; Proteobacteria; Gammaproteobacteria |
| {SALTY,ECOLI,ENT38,ERWCT} | (зеленый)Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae |
| {RALPJ,BURCA} | (сиреневый)Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae |
| {RALPJ,BURCA,NEIMA} | (желтый)Bacteria; Proteobacteria; Betaproteobacteria |
Получила из Swiss-Prot последовательности белка RS12
с данной функцией из отобранных вами бактерий
seqret sw:RS12_X
где X - мнемоника вида бактерии.
последовательности
muscle -in sw.fasta -out sw_aligned.fasta
Экспортировала в GeneDoc:
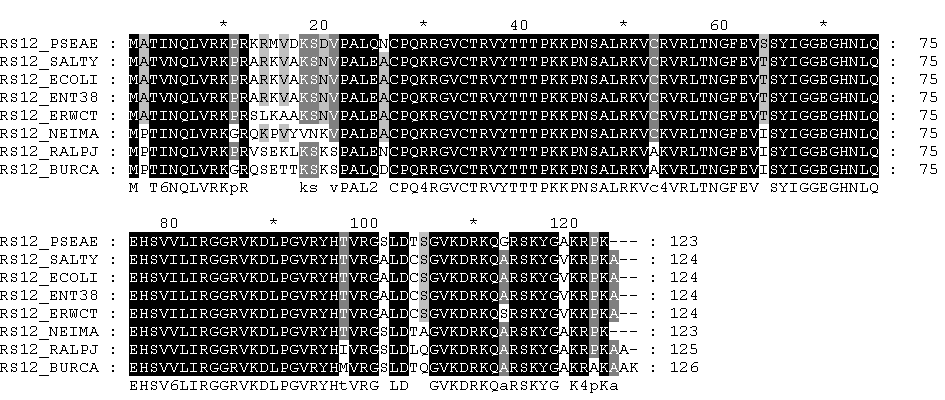
Подкрашены позиции, консервативные внутри таксона:

fprotpars -sequence sw_aligned1.msf -outfile sw_tree.fprotpars
Сравнила лучшее полученное дерево (неукорененное) из 5 с правильным. Оно совпало.
+--RS12_BURCA
+--7
+-----------6 +--RS12_RALPJ
! !
+--5 +-----RS12_NEIMA
! !
! ! +--------RS12_ERWCT
! +--------4
! ! +-----RS12_ENT38
1 +--3
! ! +--RS12_ECOLI
! +--2
! +--RS12_SALTY
!
+--------------------RS12_PSEAE
((((BURCA,RALPJ),NEIMA),(ERWCT,(ENT38,(ECOLI,SALTY)))),PSEAE)
Получила матрицу расстояний:
PSEAE SALTY ECOLI ENT38 ERWCT NEIMA RALPJ BURCA
PSEAE 0.000000 0.122280 0.122280 0.122280 0.141314 0.149348 0.155398 0.160124
SALTY 0.122280 0.000000 0.000010 0.000010 0.049010 0.154896 0.184116 0.210885
ECOLI 0.122280 0.000010 0.000000 0.000010 0.049010 0.154896 0.184116 0.210885
ENT38 0.122280 0.000010 0.000010 0.000000 0.049010 0.154896 0.184116 0.210885
ERWCT 0.141314 0.049010 0.049010 0.049010 0.000000 0.176529 0.195896 0.219681
NEIMA 0.149348 0.154896 0.154896 0.154896 0.176529 0.000000 0.147669 0.128858
RALPJ 0.155398 0.184116 0.184116 0.184116 0.195896 0.147669 0.000000 0.077149
BURCA 0.160124 0.210885 0.210885 0.210885 0.219681 0.128858 0.077149 0.000000
Аксиома ультраметричности: "из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
Оценим расстояния:
d(SALTY,ERWCT)=0.049010 d(ECOLI,ERWCT)=0.049010 d(SALTY,ECOLI)=0.000010 0.049010=0.049010 и больше 0.000010 тройка удовлетворяет аксиоме ультраметричности.
Аддитивность: если есть 4 последовательности: A, B, C, D, - то из трех сумм d(A,B) + d(C,D); d(A,C) + d(B,D); d(A,D) + d(B,C) две равны между собой и больше третьей. Оценим: d(SALTY,ERWCT)+d(ECOLI,NEIMA)= 0.049010+0.154896=0,203906 d(ECOLI,ERWCT+d(SALTY,NEIMA)= 0.049010+0.154896=0,203906 d(ERWCT,NEIMA)+d(SALTY,ECOLI)= 0.176529+0.000010=0,176539 Эти последовательности удовлетворяют аддитивности.
Neighbor-Joining:
fneighbor -datafile sw_tree.fprotdist -outfile sw.N-J.fneighbor
-outtreefile sw.N-J.fneighbor.tree
UPGMA:
fneighbor -datafile sw_tree.fprotdist -outfile sw.UPGMA.fneighbor
-outtreefile sw.UPGMA.fneighbor.tree -treetype u
Neighbor-Joining:(c неукорененным деревом и длинами ветвей) +--RS12_NEIMA +-2 ! ! +-RS12_RALPJ ! +--1 ! +--RS12_BURCA ! ! +-RS12_ERWCT 3--4 ! ! +RS12_SALTY ! +-5 ! ! +RS12_ECOLI ! +-6 ! +RS12_ENT38 ! +--RS12_PSEAE ((RS12_NEIMA:0.05238,(RS12_RALPJ:0.03107,RS12_BURCA:0.04608):0.04731):0.03422, (RS12_ERWCT:0.03325,(RS12_SALTY:0.00000,(RS12_ECOLI:0.00000, RS12_ENT38:0.00000):0.00000):0.01575):0.05671,RS12_PSEAE:0.05020);
UPGMA:(c укорененным деревом и длинами ветвей)
+--RS12_PSEAE
!
! +RS12_SALTY
+-5 +-1
! ! +-2 +RS12_ECOLI
! ! ! !
! +-3 +RS12_ENT38
--7 !
! +RS12_ERWCT
!
! +---RS12_NEIMA
+-6
! +-RS12_RALPJ
+-4
+-RS12_BURCA
((RS12_PSEAE:0.06352,(((RS12_SALTY:0.00000,RS12_ECOLI:0.00000):0.00000,
RS12_ENT38:0.00000):0.02450,RS12_ERWCT:0.02451):0.03901):0.02670,
(RS12_NEIMA:0.06913,(RS12_RALPJ:0.03857,RS12_BURCA:0.03857):0.03056):0.02109);
Топологии деревьев совпадают.
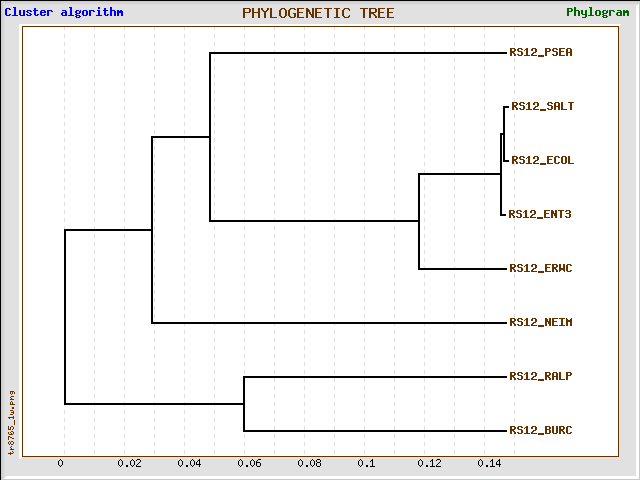 Топология этого дерева несколько не совпадает с предыдущими
Топология этого дерева несколько не совпадает с предыдущими