Гомологичное моделирование комплекса белка с лигандом
- Выравнивание белка 1lmp и лизоцима LYS_BOMMO
Выравнивание было произведено программой Clustal, файл выравнивания.
- Модификация файла со структурой 1lmp
- Создание управляющего скрипта lys_bommo.py
В скрипте указано:
- что нужно использовать стандартные валентные углы в полипептидной цепи (строчка 4)
- что дополнительно нужно сохранять взаимное расположение определенных пар атомов (3.5 ангстрема);
- В данном случае трех атомов белка, образующих водородные связи с тремя атомами лиганда - строчки 5-7 с ID пар атомов; параметры взаимного расположения атомов пары описаны в строчке 9-10. 3 точки могут однозначно расположить сложную структуру в пространстве, поэтому мы выбираем водородные связи как источник данных точек.
- что ковалентные связи в гетероатомах нужно вычислять по расстояниям между атомами (так же, как это делает Rasmol), строчка 12
- что имя файла с выравниванием и имена последовательностей образца и моделируемого белка, строчка 13 (а имя файла со структурой содержится в выравнивании)
- что число и номера моделей, которые нужно построить (в данном примере 5 моделей), строки 14-15
- что пора строить модель, строчка 16
- Анализ полученных структур
Мы видим, что несмотря на разные аминокислотные последовательности и то, что белок LYS_BOMMO на 10 а.к. короче 1lmp, элементы вторичной структуры у них совпадают.
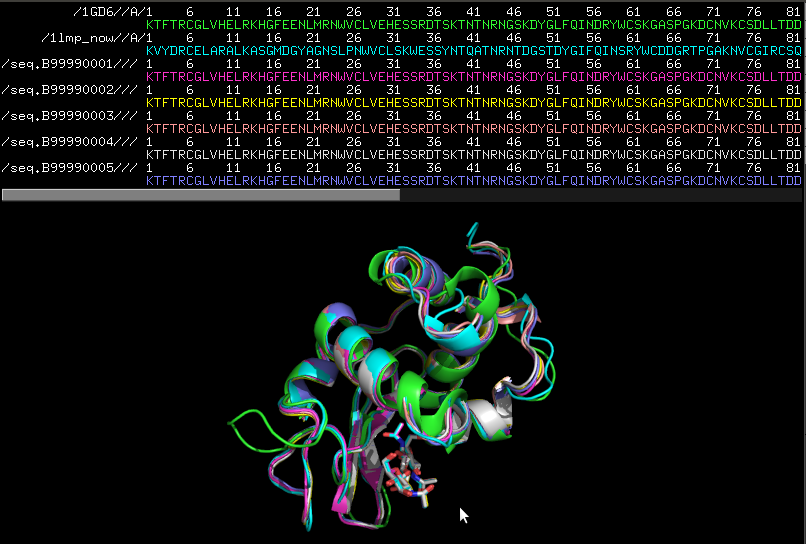
На рисунке представлены структуры 1lmp (бирюзовая) и 5 моделей гомолога, причем седьмая модель 1GD6 (зеленая) - белок LYS_BOMMO, структура которого взята была из pdb банка. Есть несоответствие между петлей и некоторых частей α-спиралей в структуре 1GD6 и 5 построенных моделей LYS_BOMMO.
Проверка на сервере WHATIF
В таблице голубым фоном выделены те значения Z-score пяти моделей, которые ближе всего к этому значению у 1lmp. Нельзя исходя из этого выбрать лучшую модель, притом что выравнивались они в PyMol почти одинаково. Поэтому для следущего практикума я возьму структуру под номером 1.Name Ramachandran Z-score RMS Z-score for bond lenghts RMS Z-score for bond angeles 1lmp -0.965 0.789 1.759 1 -1.333 1.034 1.535 2 -1.645 0.985 1.421 3 -1.684 0.983 1.490 4 -1.697 0.987 1.404 5 -1.967 1.003 1.547
Моделирование структур биополимеров
© Migur Anzhela 2012