Реконструкция филогении по нуклеотидным последовательнсотям. Анализ деревьев, содержащих паралоги
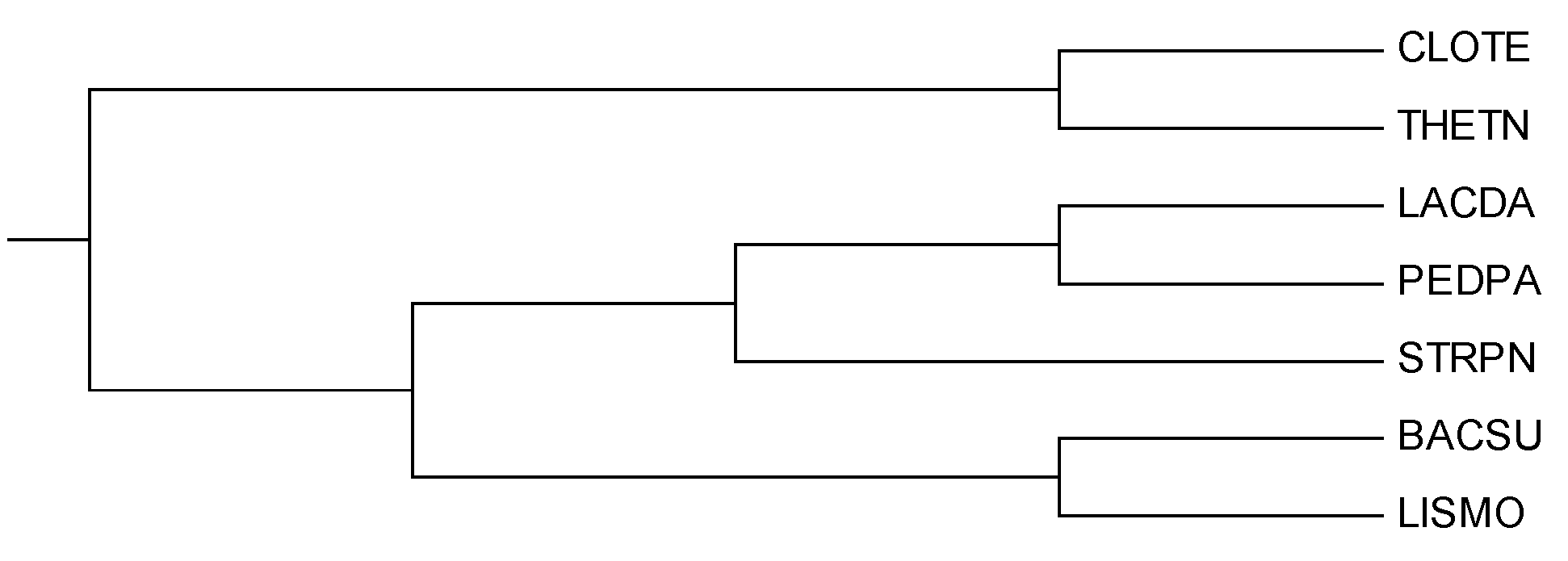
Правильное дерево
- Построение дерева по нуклеотидным последовательностям
- Построение и анализ дерева, содержащего паралоги
- CLPE_BACSU и CLPC_BACSU
- CLPY_BACSU и CLPX_BACSU
- HSLU_THETN и CLPX_THETN
- CLPX_CLOTE и CLPX_THETN
- CLPX_LISMO и CLPX_BACSU
- CLPX_PEDPA и CLPX_STRPN
Дерево построено при помощи программы fdnapars. Подробно в файле и выравнивание.
+-BACSU
|
+---4 +---PEDPA
| | +--3
| | | +----LACDA
| +--2
| | +-LISMO
| +--5
| +-----STRPN
|
1-----CLOTE
|
+------THETN
Правильно построены ветви:
(CLOTE,THETN), (PEDPA,LACDA)
Дерево, построенное программой fprotpars
+--THETN
+-----6
! +--CLOTE
+--5
! ! +--LISMO
+--3 +-----4
! ! +--BACSU
+--2 !
! ! +-----------PEDPA
1 !
! +--------------STRPN
!
+-----------------LACDA
Эти два дерева очень отличаются, они имеют только одну одинаковую тривиальную ветвь (CLOTE,THETN). Дерево, построенное программой fprotpars имеет 3 правильные ветви. Поэтому, возможно, построение деревьев по белкам точнее, чем по нуклеотидным последовательностям.
Дерево построено программой fprotpars
+-----HSLU_LISMO
+-11
! ! +--CLPY_BACSU
+----------------10 +-12
! ! +--HSLU_THETN
! !
! +--------HSLU_LACDA
!
+-----9 +--CLPX_CLOTE
! ! +-----8
! ! ! +--CLPX_THETN
! ! +-----7
! ! ! ! +--CLPX_LISMO
! ! ! +-----6
+--3 +-----------5 +--CLPX_BACSU
! ! !
! ! ! +--CLPX_PEDPA
! ! +-----------4
! ! +--CLPX_STRPN
1 !
! ! +--CLPE_BACSU
! +-----------------------------2
! +--CLPC_BACSU
!
+-----------------------------------FTSH_STRPN
- Паралоги
- Ортологи
Программа почти все белки с одинаковой функцией расположила на отдельные ветви
© Migur Anzhela 2010