Трансмембранные белки
- Сравнение трансмембранных белков с разной структурой
Альфа-спиральные трансмембранные белки 1uaz »
Archaerhodopsin-1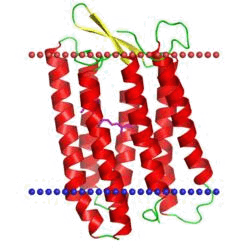
1m56 »
Bacterial cytochrome c oxidase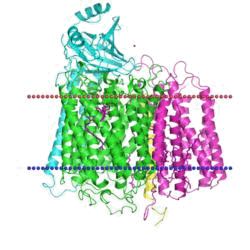
2k1k »
Receptor tyrosine kinase EphA1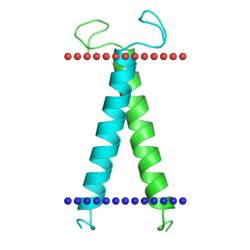
1afo »
Glycophorin A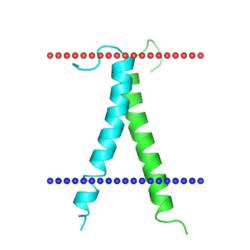
2kv5 »
Fst toxin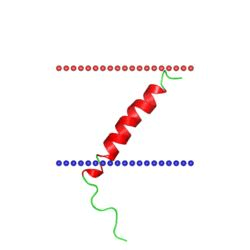
Трансмембранные бета-баррели 2jmm »
Transmembrane beta-barrel platform protein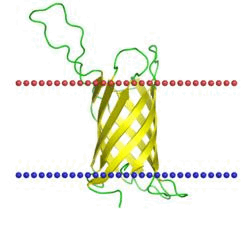
1qd5 »
Outer membrane phospholipase A, monomer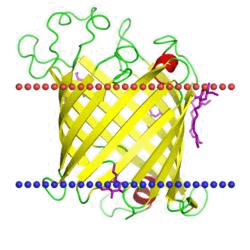
1af6 »
Maltoporin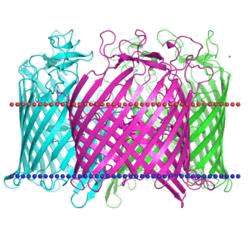
3fid »
Lipid A deacylase LpxR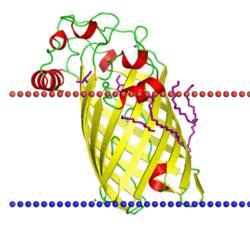
3gp6 »
Lipid A acylase PagP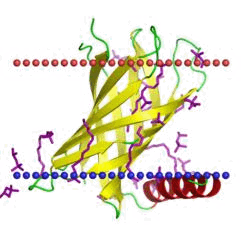
Сходство между двумя типами трансмембранных белков в том, что элементы спиралей или бета-листов соединяются друг с другом с помощью петель вне мембран. А в структуре бета-баррелей замечены элементы альфа-спиралей, но они лежат либо вне мембраны, либо внутри, но не доминируют в структуре белка.
Табл.1
PDB код Число цепей Тип
(спираль, баррель)Число трансмембранных участков в цепи Число остатков в одном трансмембранном участке
(типичное, минимальное, максимальное)(*) Толщина мембраны в ангстремах
(расстояние между атомами на границах трансмембранных участков перпендикулярно мембране1uaz 1 спираль 7 23, 19, 24 31.8 ± 1.3 Å 2jmm 1 баррель 8 10, 9, 11 23.9 ± 1.3 Å - Трансмембранный белок P21448
- По аннотациям записи Uniprot (координаты мембранных и цитоплазматических остатков)
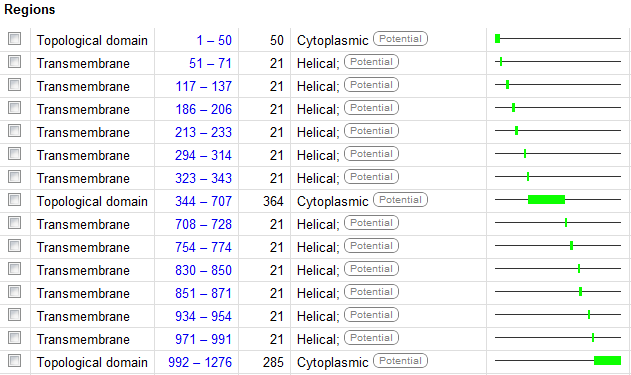
По аннотациям записи Uniprot - в мембране 12 спиралей - TMHMM
# sp_P21448_MDR1_CRIGR Length: 1276 # sp_P21448_MDR1_CRIGR Number of predicted TMHs: 11 # sp_P21448_MDR1_CRIGR Exp number of AAs in TMHs: 247.07675 # sp_P21448_MDR1_CRIGR Exp number, first 60 AAs: 12.77622 # sp_P21448_MDR1_CRIGR Total prob of N-in: 0.98442 # sp_P21448_MDR1_CRIGR POSSIBLE N-term signal sequence sp_P21448_MDR1_CRIGR TMHMM2.0 inside 1 47 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 48 70 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 71 112 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 113 135 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 136 186 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 187 209 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 210 213 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 214 236 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 237 291 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 292 314 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 315 323 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 324 346 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 347 706 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 707 729 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 730 751 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 752 774 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 775 847 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 848 870 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 871 931 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 932 954 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 955 966 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 967 989 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 990 1276

TMHMM показывает 11 трансмембранных спиралей - Сравнение с гомологом с известной пространственной структурой 3G5U
Пространственное изображение гомолога белка P21448
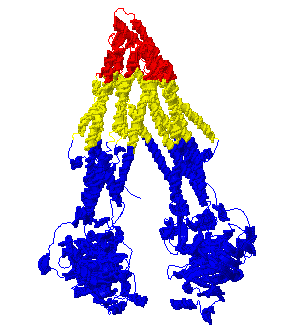
Желтым обозначен трансмембранный участок:
- Трансмембранные участки спиралей (координаты а.о.)
- 45-68
- 114-132
- 184-203
- 215-234
- 291-310
- 332-349
- 708-728
- 753-773
- 829-847
- 852-872
- 933-953
- 973-990
Всего 12 трансмембранных спиралейВыравнивание в файле. Белок P21448 и его гомолог, участки из TMHMM и UniProt. Зеленым отмечены трансмембранные участки спиралей (H). Всего 11 участков.
- По аннотациям записи Uniprot (координаты мембранных и цитоплазматических остатков)
- Сравнение предсказания TMHMM с предсказанием по структуре гомолога
Число а.к. остатков Всего а.к. остатков в последовательности 1276 Остатки, предсказанные TMHMM как локализованные в мембране (всего) 242 Правильно предсказали - совпадают с 3D предсказанием (true positives, TP) 214 Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным 3D таковыми не являются, false positives, FP) 28 Правильно не предсказали ( не предсказаны, и по данным 3D не находятся в мембране, true negatives, TN) 1010 Не предсказали то, что нужно (остатки по данным 3D находятся в мембране, false negatives, FN) 24 Чувствительность (sensivity) = TP / (TP+FN) 0,9 Специфичность (specificity) = TN / (TN+FP) 0,8 Точночть(precision) = TP /(TP+FP) 0,88 Сверхпредсказание = FP/ (FP+TP) 0,12 Недопредсказание = FN / (TN+FN) 0,18