В этом практикуме я научилась пользоваться программой BLAST и разобралась, что означает каждый из параметров запуска. Также я изучила критерии, по которым можно судить о гомологичности белковых последовательностей.
Описание параметров Blast
Построение множественного выравнивания
Среди находок я выбрала 7 белков. Было выбрано 6 предположительно гомологичных белков с разными названиями и отличающимися значениями E-value, но с большим процентом покрытия(>90%). Для сравнения был включён белок с большим значением E-value (2,8) и покрытием 50%. В программе Jalview было построено множественное выравнивание выбранных последовательностей (Рисунок 1). Затем я удалила часть последовательностей, участки выравнивания с которыми не свидетельствовали о гомологии. На Рисунке 2 можно увидеть выравнивание всех гомологичных последовательностей.
Нетрудно заметить, что представленный участок с 87 по 102 аминокислоту представлен 16 колонками без гэпов, начинается и завершается абсолютно консервативной позицией и имеет высокую плотность консервативных позиций (7 абсолютно консервативных колонок). Эти признаки могут свидетельствовать о гомологии выбранных последовательностей. Полное выравнивание можно увидеть в проекте (jvp).


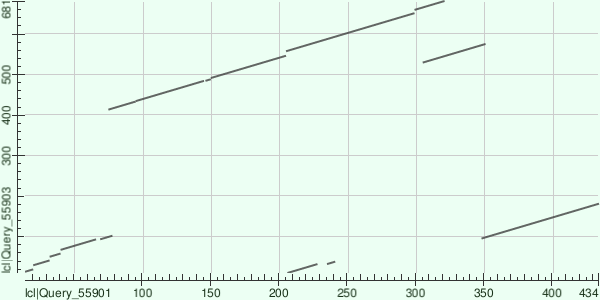
Из предложенных белков в файле было выбрано 2 белка из разных групп (идентификаторы U1LPR7_9MICO; F4Q4S4_CAVFA). Это белки из организмов Agrococcus pavilionensis RW1 и Cavenderia fasciculata (strain SH3) (Slime mold). Карта сходства (Dot Matrix) представлена на Рисунке 3. Для этих последовательностей программа BLAST привела 5 вариантов возможных локальных выравниваний (изображение). Рассмотрим первые три выравнивания с низким E-value. По карте видно, что первое выравнивание представляет собой достаточно длинный участок с мелкими делециями в первой последовательности (я насчитала 4 штуки). Второй участок выровнен без использования гэпов, поэтому линия не прерывается. Можно предположить, что произошла транслокация участка первой последовательности (участок в результате крупной перестройки мог переместится в конец). Думаю, эти белковые последовательности гомологичны.
Поиск по "случайной" последовательности
Для первого эксперимента я взяла последовательность, которая точно не кодирует белок - 'You can get addicted to a certain kind of sadness'. Для начала я запустила Blast с параметрами по умолчанию. Программа вывела один единственный результат с покрытием 67% и E-value, равным 2.6 (всё равно высокий). Очевидно, с повышением порога на E-value (параметр Expect threshold) количество находок возрастает. Поиск только по базе данных Swissprot дал 3 результата с E-value 21, остальные на порядки выше. Изменив параметр Word size на 3, добавилась интересная находка с E-value 0,25 (предсказанный белок) и покрытием 92% (Рисунок 4). То есть, действительно, при уменьшении длины слова поиск получается более полноценным, хоть и занимает больше времени.
Поиск по последовательности белка D-аланин-D-аланиновой лигазы с измененными параметрами
Отталкиваться буду от результатов задания 1. Следующие запуски были проведены с измененными параметрами Database: UniprotKB/Swiss-Prot, Max target sequences: 20000 и Word size = 3. Остальные параметры были выставлены по умолчанию.Word Size: 6. Число находок: 468
Word Size: 3. Число находок: 544
Word Size: 2. Число находок: 547
Можно сделать вывод, что с уменьшением длины слова возрастает число находок. Новые результаты не изменили 'топ-100' (судя по графикам).
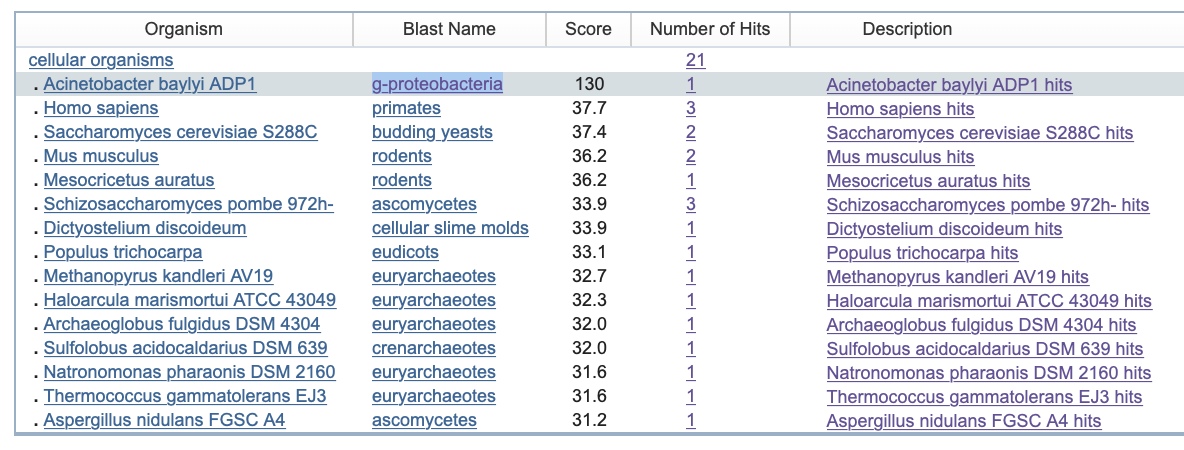
Я исключила из результатов поиска бактерий, поставив галочку рядом с exclude. Не смогла объяснить, почему из результатов поиска не исключилась гамма-протеобактерия (тогда я провела поиск только по бактериям и среди них оказалось много других видов Acinetobacter). Таксономический состав представлен на Рисунке 5. Число находок: 21.
С этим параметром неинтересно играть. Чем выше порог - тем больше результатов поиска. Число находок для порога 100: 619.
При использовании новой матрицы автоматически меняется значение по умолчанию параметра Gap Costs. При переходе от BLOSUM 62 к BLOSUM 90 увеличилось число находок (стало 548) и общий вес каждого из выравниваний. К тому же E-value существенно возрос. При переходе от BLOSUM 62 К BLOSUM 45 - всё наоборот.