В этом практикуме я рассматривала возможности AlphaFold2 для предсказания структуры последовательностей, имеющих спирали в качестве вторичной структуры. Для этого я совершила 3 запуска в ColabFold с параметрами по умолчанию для следующих последовательностей:
GSHMASLDMAEIKEKICDYLFNVSDSSALNLAKNIGLTKARDINAVLIDMERQGDVYRQGTTPPIWHLTDKKRERMQIK
GSHMASLDMAEIPPPPCDYLFNVSDSSALNLAKNIGLTKARDINAVLIDMERQGDVYRQGTTPPIWHLTDKKRERMQIK
GSHMAPPPPPPPPPPPPPPPPPPSDSSALNLAKNIGLTKARDINAVLIDMERQGDVYRQGTTPPIWHLTDKKRERMQIKПараметр use_template был выключен, то есть предсказание строилось без опоры на белки из PDB с похожим сиквенсом. Чтобы оценить предсказание программы, я сравнила предложенные модели со структурой, полученной методом рентгенотруктурного анализа.
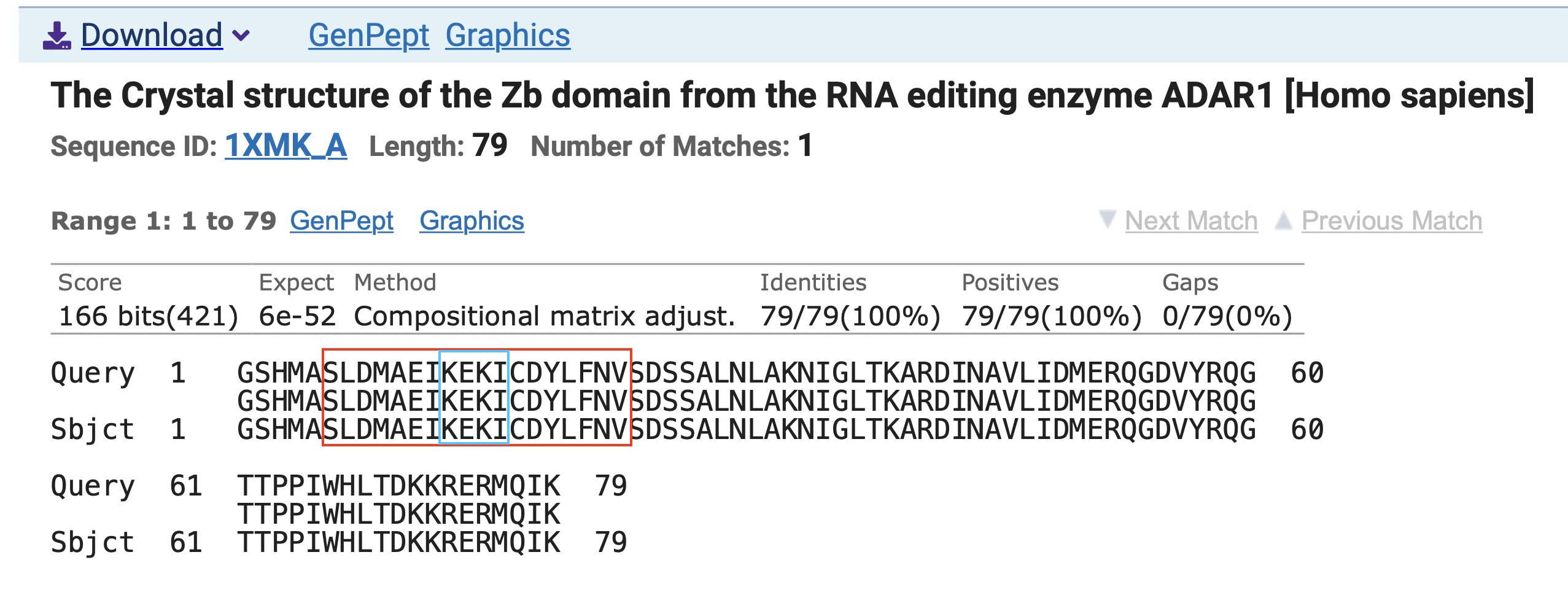
С помощью Protein Blast были получены последовательности, схожие с анализируемыми. Для первой анализируемой последовательности было обнаружено полное совпадение c последовательностью домена Zb фермента ADAR1. Выравнивание можно увидеть на Рисунке 1. Для этого белка есть структура 1XMK , разрешенная X-ray с отличным разрешением 0.97 Å.
Аденозин деаминазы (ADARs) - это ферменты, которые превращают аденозин (A) в инозин (I) в двухцепочечной РНК. [1] I структурно напоминает гуанин, поэтому нарушается нормальное спаривание A:T, и молекула РНК может стать нестабильной. ADAR1 важен для регуляции и процессинга РНК (сплайсинг, редактирование и тд), играет важную роль в иммунитете. Двухцепочечные РНК, в первую очередь ассоциируются с вирусами, но могут иметь и эндогенное происхождение (например, дцРНК транспозонов или образованные в митохондриальном матриксе). ADAR1 позволяет отличать "своих" от "чужих", ингибируя антивирусный ответ на собственные дцРНК. [2] У ADAR1 есть Z-a домен, который с высокой афинностью связывает Z-формы РНК/ДНК. Гомологичный ему домен, Z-бета, при этом связывать ДНК не может. Оба домена принадлежат к семейству доменов спираль-поворот-спираль (HTH укладка). [3]
Остальные две последовательности были получены из первой путем замещения остатков N-конца на цепочки пролинов разной длины - из 4 и 16 остатков пролинов соотвестсвенно. Подряд идущие пролины известны тем, что сами могут образовывать вторичную структуру - полипролиновую спираль. Обычно в белках встречается не более 5 идущих подряд остатков пролина, эти участки плохо кристаллизуются.[4] Видимо, таких структур с большим числом подряд идущих пролинов немного, я нашла одну - 4OR9. Посмотрим, как с этой задачей справится AlphaFold2.

После каждого запуска я получала 5 предсказаний. Они покрашены от N к C концу, а также по метрике pLDDT, которая отражает "уверенность" в предсказании для каждой позиции.
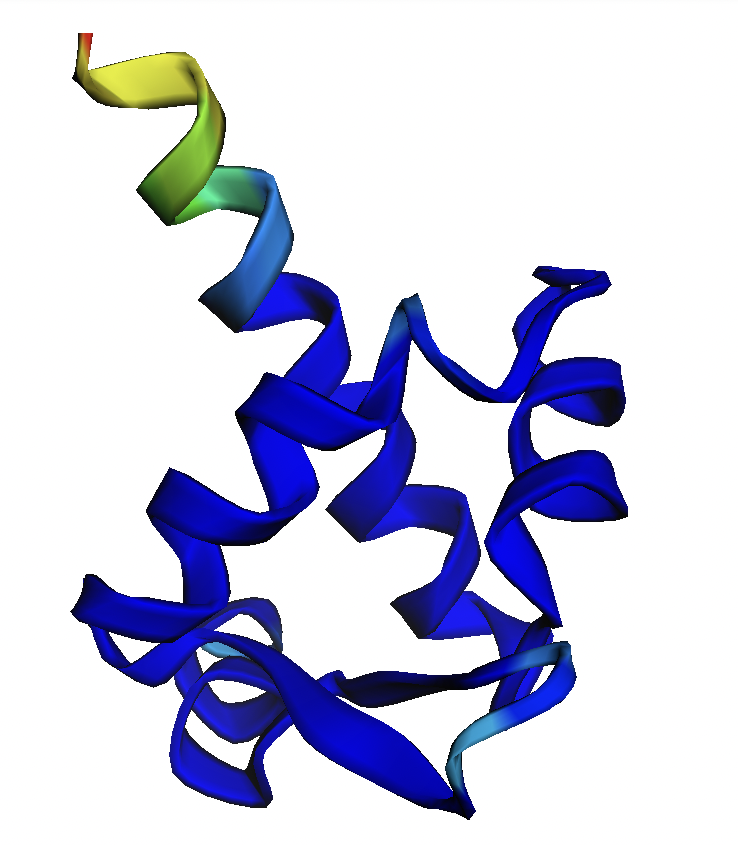
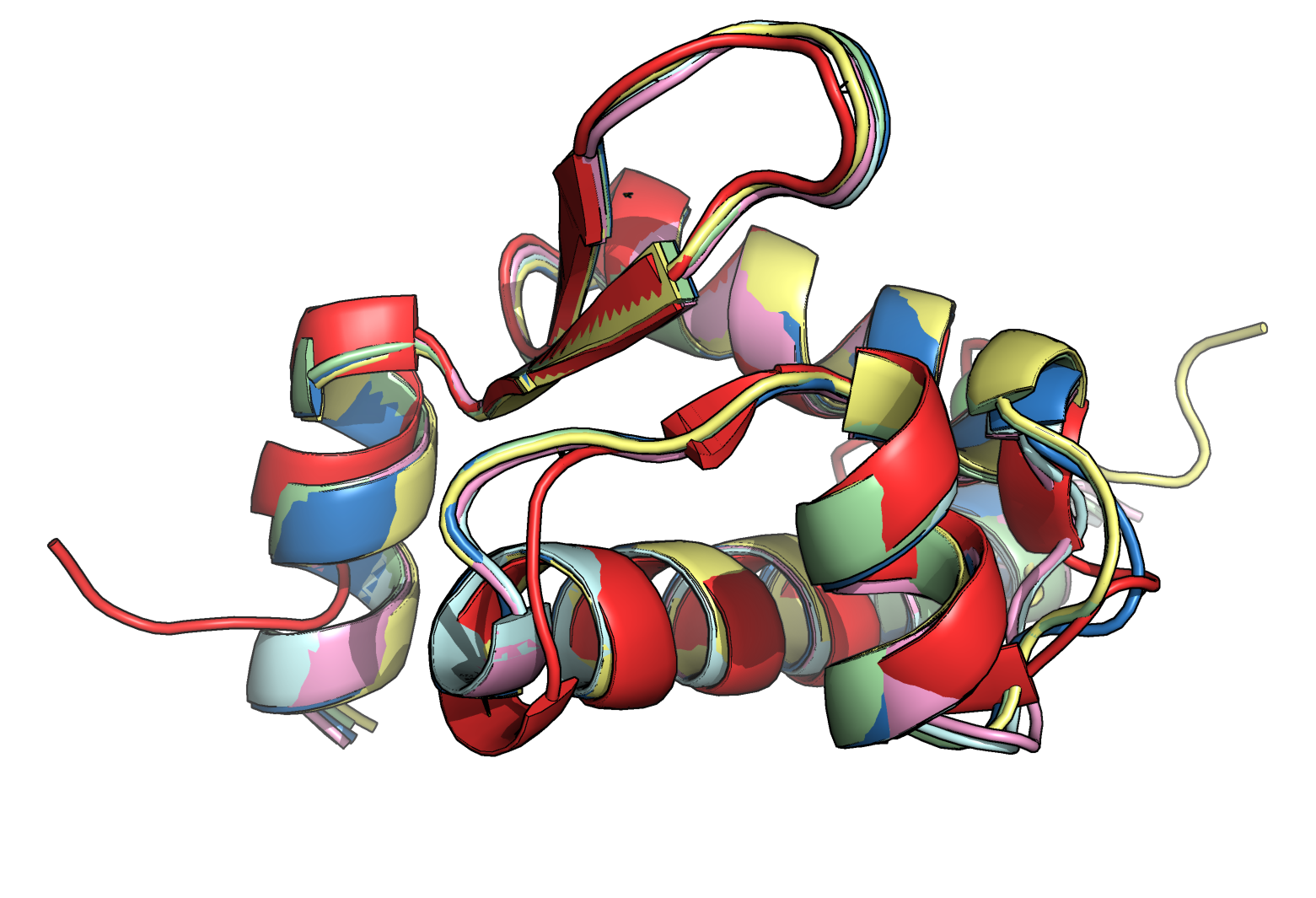
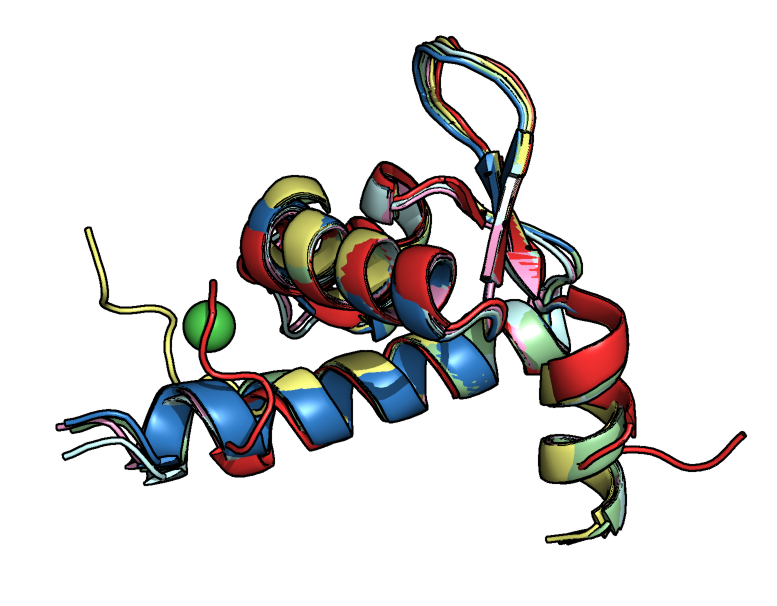
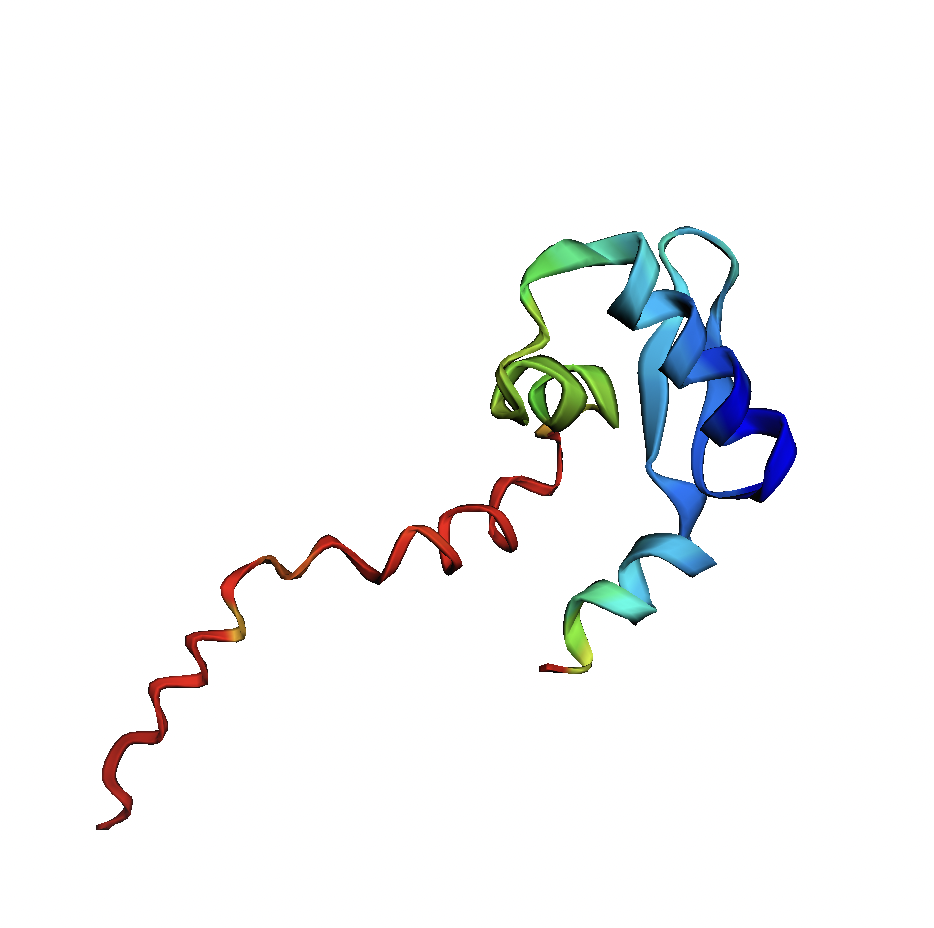
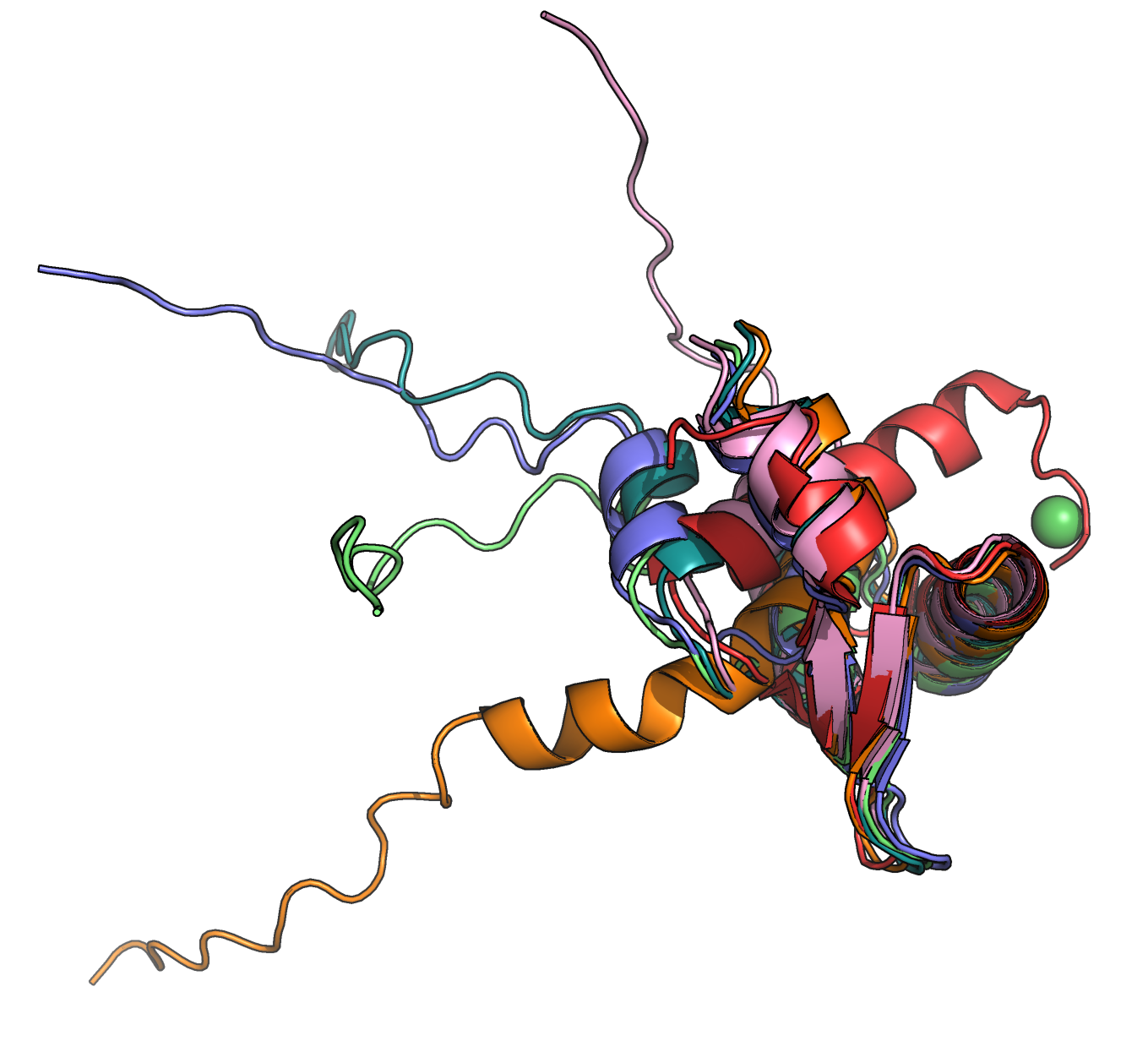
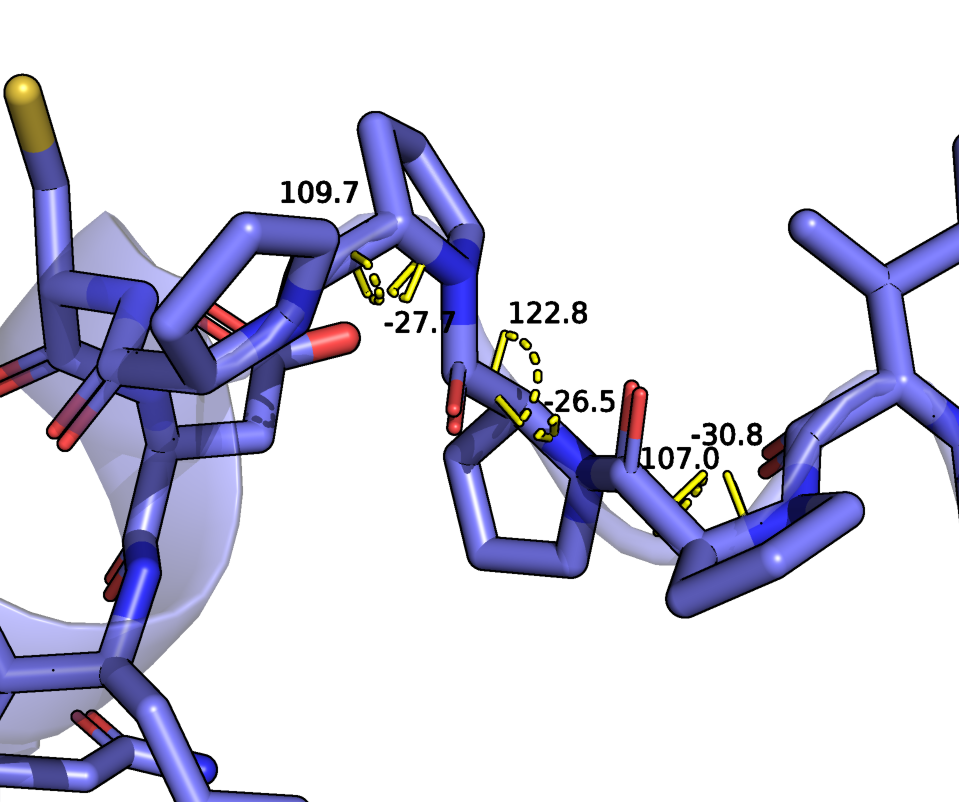
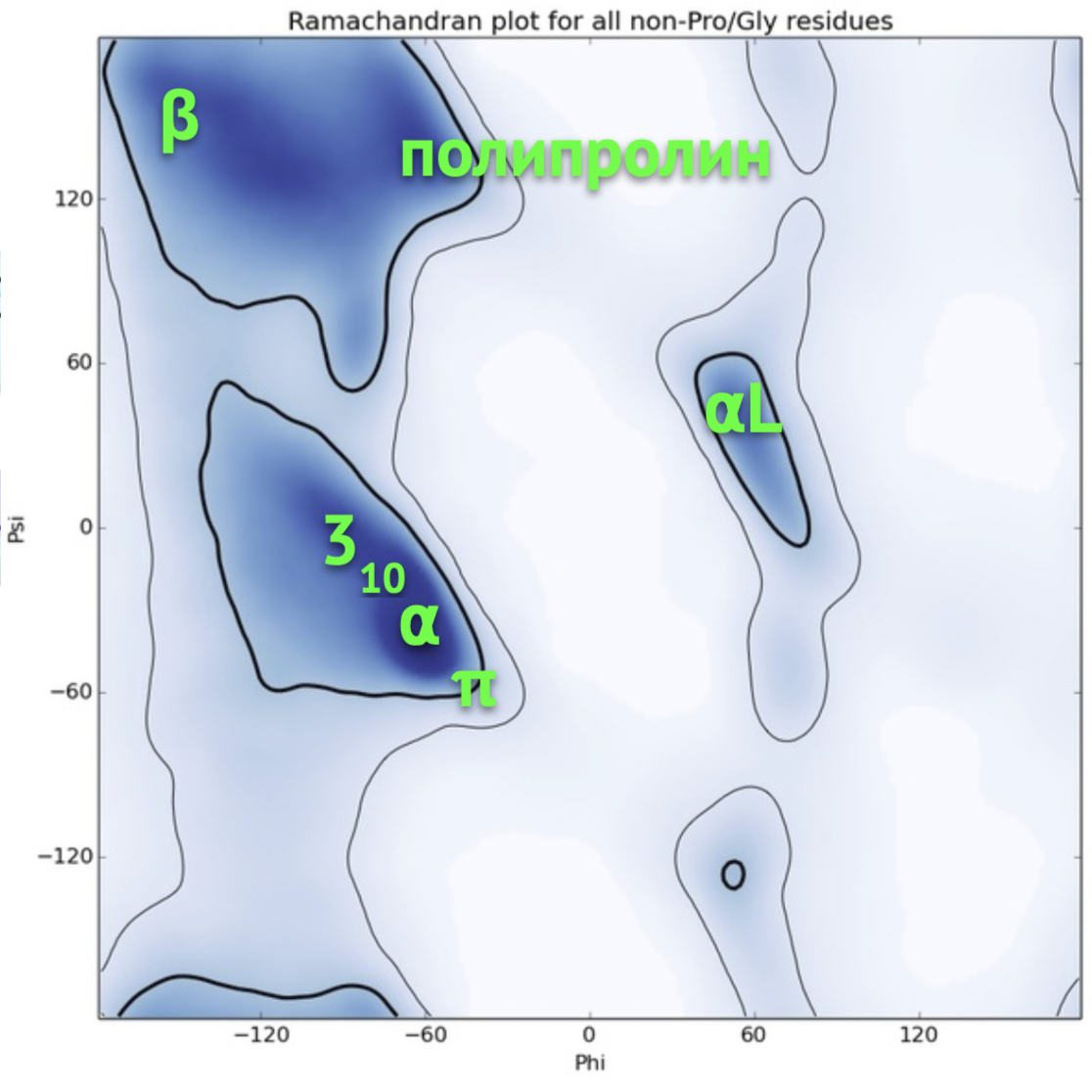
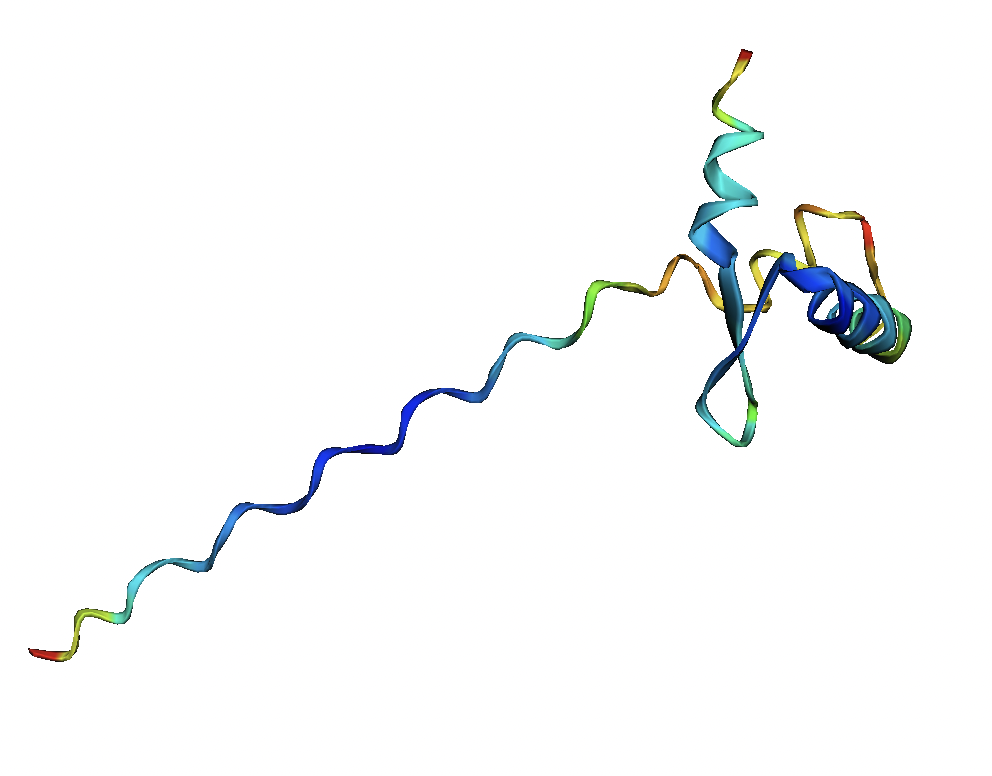
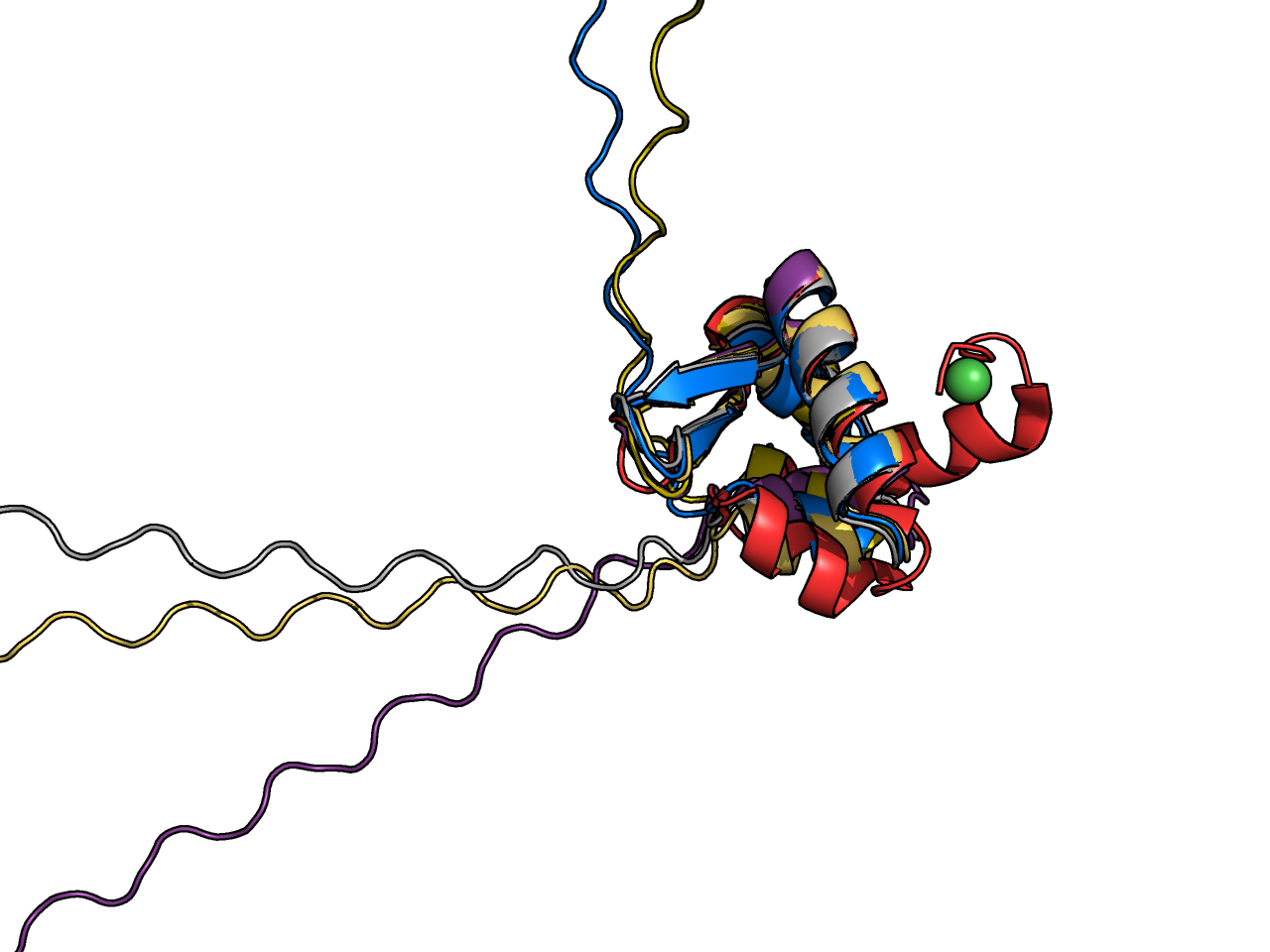
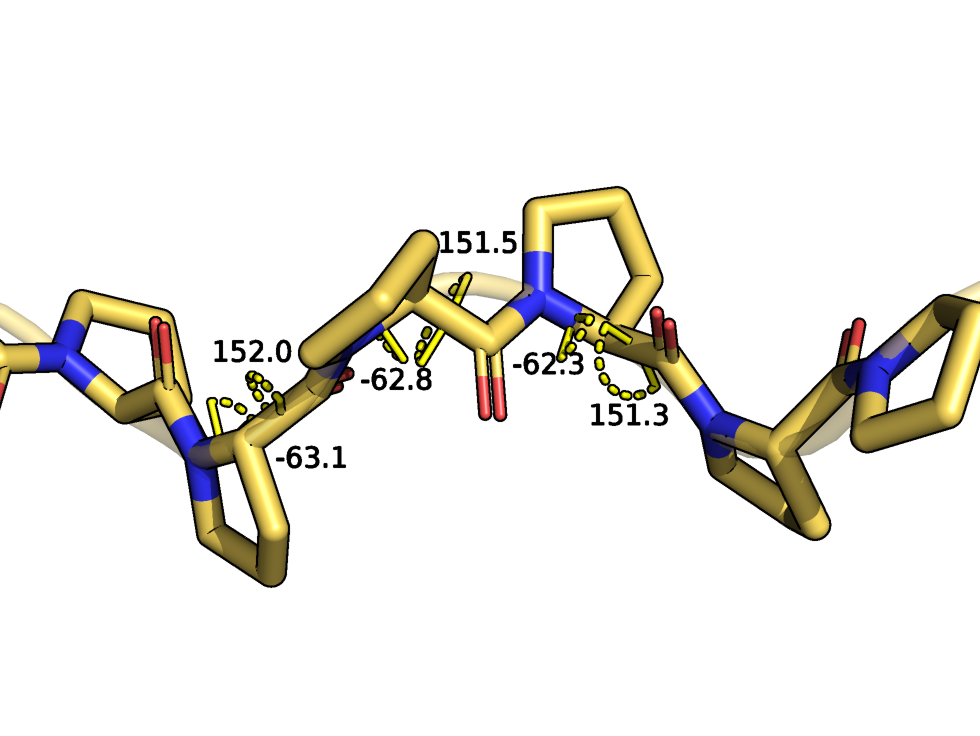

Я рассмотрела работу AlphaFold2 на частном примере. Он хорошо справился с предсказанием исходной последовательности, структура была близка к разрешенной X-Ray. При добавлении пролинов начались трудности, и я бы не стала здесь доверять прогнозам AlphaFold. Наверное, он пока не умеет надежно работать в "нестандартных" ситуациях. Анализ работы усложнен тем, что я нашла мало структур с паттернами из пролинов, идущих подряд. Следует отдельно рассматривать ситуации с другим числом пролинов, и пролинами,идущими не подряд. Также отдельно должны быть рассмотрены ситуации, когда полипролиновая спираль попадает в середину последовательности.
[1] Functions of the RNA Editing Enzyme ADAR1 and Their Relevance to Human Diseases
[2] ADAR1: “Editor-in-Chief” of Cytoplasmic Innate Immunity
[4] The structure and function of proline-rich regions in proteins