Для выравнивания двух последовательностей я выбрала две разные сборки геномов ВИЧа, а именно M30895.1 и KP890355.1
Геномы искались при помощи "Browse by Organism" в NCBI с параметрами: сборка complete. Взяла первые две находки.
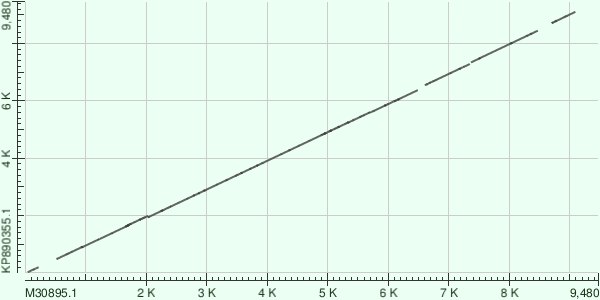
Было проведено 2 выравнивания: blastn и megablast. Результаты выравнивания можно увидеть на DotPlot (рис. 1 и рис. 2):
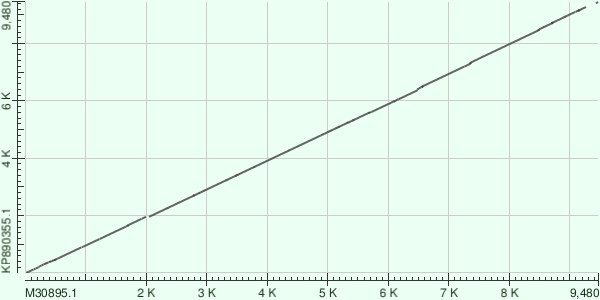
Основываясь на рисунках, можно сделать вывод, что blastn-ом последовательности выровнялись лучше: больше гомологичных кусков найдено. Blastn указывает на участок делеции или инсерции около 2к позиции. Megablastом получилась весьма странная картина, а именно отсутствие чего-либо по углам (в начале и конце последовательности): вообще не попали в выравнивание 0-2к и 6.5-10к. Такая ситуация может возникнуть из-за неудачно подобранных длинны слова и штрафов.
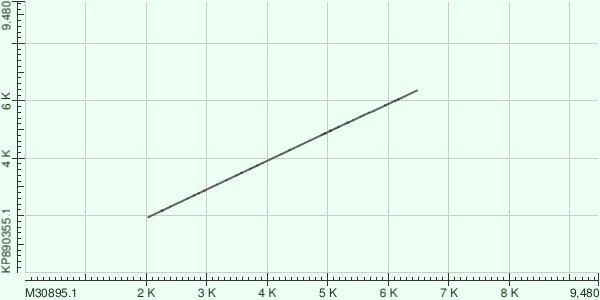
Чтобы в этом убедиться я запустила megablast ещё раз, но уже с word size 16 (рисунок 3). Как и следовало ожидать на ранее пустых местах детектировалось сходство. При этом также наблюдается участок делеции или инсерции около 2к позиции, правда меньших чем при blastn. На данной карте видны и другие крупные участки делеции/инсерции: 6,5к; 7,3к и 8,5к.