

Для выполнения данного практикума был выдан идентификатор трансмембранного белка: GERQB_BACCE. В Uniprot в разделе Structure было найдено предсказание трехмерной структуры белка.
В базе данных OPM (Orientations of Proteins in Membranes database) была найдена структура с β-листами в трансмембранной части FadL (FADL_ECOLI, 3pgu) (рис. 1). Это транспортер длинных жирных кислот внешней мембраны E.coli, данный белок также служит рецептором.
С помощью OPM были найдены следующие характеристики:
Толщина гидрофобной части белка в мембране: 25.2 Å;
Координаты трансмембранных участков: 1( 43- 52), 2( 78- 87), 3( 94- 101), 4( 126- 135), 5( 142- 149), 6( 207- 216), 7( 223- 229), 8( 274- 281), 9( 289- 296),10( 325- 333),11( 341- 348),12( 367- 376),13( 383- 390),14( 411- 421)
Среднее количество остатков в одном β-тяже белка: 8;
Белок находится на различных мембранах клетки хозяина.
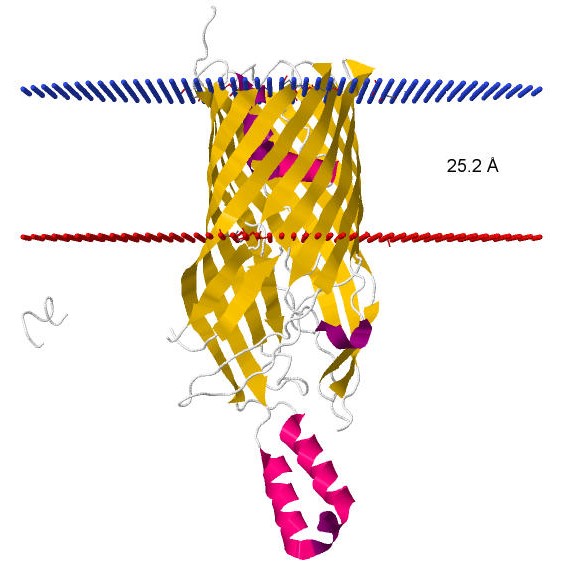
Рисунок 1. Вторичная структура белка FadL. Синим показана n-сторона, красным - p-сторона.
Сервис DeepTMHMM помогает предсказывать трансмембранные участки по первичной структуре белка. В графической выдаче по горизонтали отложены координаты остатков белка, а по вертикали показаны предсказание положение определенных участков (см верхние рисунки) и вероятность принадлежности конкретного остатка к той или иной топологии (см нижние рисунки). Каждый цвет означает определенную локализацию участка: Membrane - трансмембранный, Inside - внутриклеточный, Outside - внеклеточный.
Для выданного белка было предсказано (рис.2) 10 трансмембранных участков (α-спиралей). N- и C-концы находятся по внутреннюю сторону мембраны.
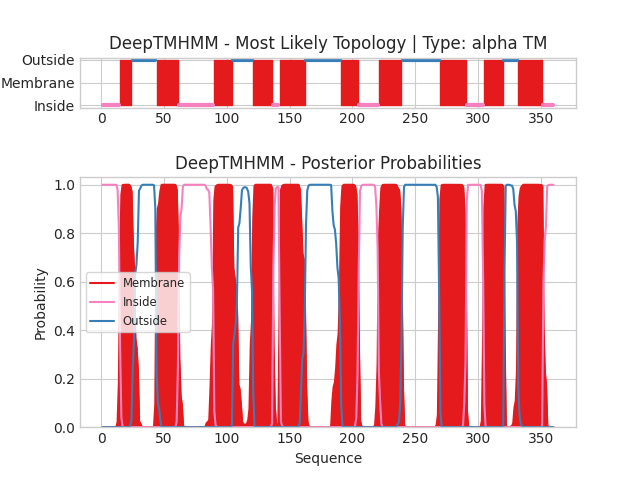
Рисунок 2. Выдача для альфа-спирального GERQB_BACCE
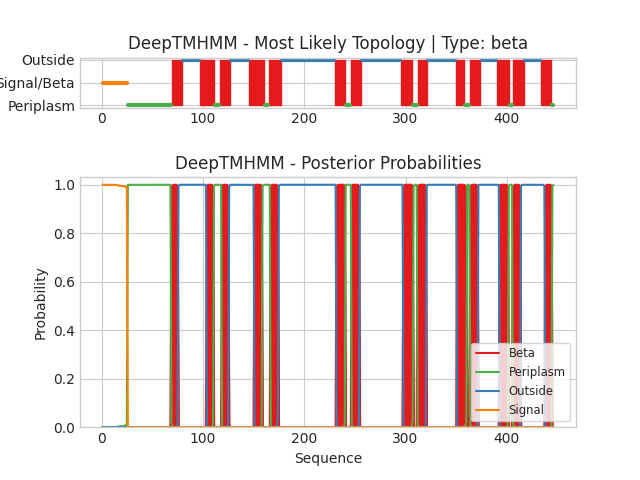
Рисунок 3. Выдача для "бета-бочонкового" FADL_ECOLI
Для рисунка 3 нужно дополнительное описание легенды. Каждый цвет означает определенную локализацию участка: Beta - бета-слой (мембранный), Periplasm - периплазматический (аналог внутриклеточной части), Outside - внеклеточный, Signal - сигнальный пептид.
Для этого белка было предсказано 14 трансмембранных участков (β-слоев). N-конец белка является сигнальным пептидом, а С- внутриклеточным (periplasm)
Предсказания не совсем совпадают с OPM. Но в данной выдаче учитывается сигнальный пептид, поэтому координаты всех предсказанных β-слоев сдвигаются на его длину.
С помощью PPM я анализиловала "свой" альфа-спиральный белок GERQB_BACCE. Для него не существует достоверной структуры, однако существует ее модель, которая предсказана с помощью AlphaFold. Средняя оценка достоверности (pLDDT) у этой модели довольно высокая (> 90), но концевые области оставляют желать лучшего (< 50). С помощью сервиса PPM было предсказано положение этого белка относительно мембраны (рис.4).
Критерии для анализа:
Number of Membranes: 1
Type of membrane: Gram-positive bacteria inner membrane
Allow curvature: no (чтобы не усложнять анализ выдачи)
Topology (N-ter): out (по предсказанию DeepTMHMM)
Рисунок 4. Результат работы PMM с белком GERQB_BACCE. Синим показана n-сторона, красным - p-сторона.
Толщина гидрофобной части белка в мембране: 28.0 ± 1.5 Å; Угол изгиба: 21 ± 0°; Координаты трансмембранных участков: 1( 14- 32), 2( 42- 62), 3( 82- 111), 4( 119- 138), 5( 142- 166), 6( 190- 204), 7( 217- 236), 8( 267- 294), 9( 302- 320),10( 326- 351); Среднее количество остатков в одном β-тяже белка: 28.
Оба сервиса показали довольно схожие результаты. Ключевое, что они находят одинаковое количество трансмемебранных участнов и N-/C-концы локализованы одинаково в двух моделях (внутри). Ниже предствлено более детальное сравнение белковых участков.
DeepTMHMM: 1(15-24), 2(44-61), 3(90-104), 4(121-136), 5(142-162), 6(191-204), 7(221-239), 8(270-290), 9(305-320), 10(332-351)
PMM: 1(14-32), 2(42-62), 3(82-111), 4(119-138), 5(142-166), 6(190-204), 7(217-236), 8(267-294), 9(302-320), 10(326-351)
Как можно заметить, участки отличаются совсем немного своей длинной (несколько аминокислот), что приятно, также это подтверждает модель AlphaFold (рис.5) с высокой степенью достоверности в данных позициях. В целом, можно считать, что структура предсказана успешно.
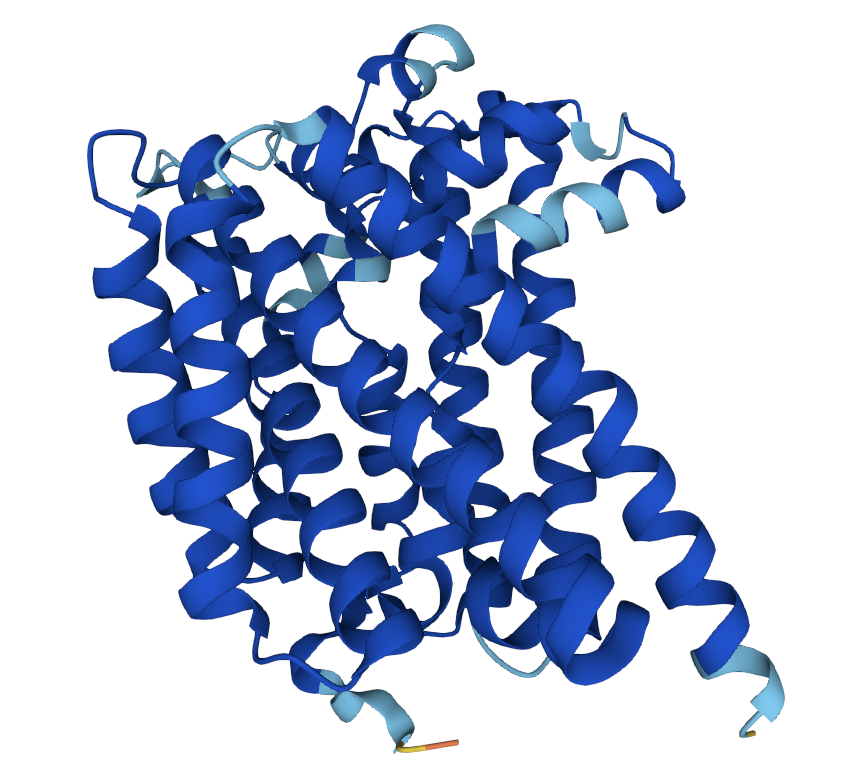
Рисунок 5. Структурная модель AlphaFold для GERQB_BACCE. Синий - pLDDT > 90; голубой - 90 > pLDDT > 70; желтый - 70 > pLDDT > 50; оранжевый - pLDDT < 50.