

Сайт транс-сплайсинга является сигналом к вырезанию части мРНК (аутрону), предшествующей экзону, и замене его на 22-звенную последовательность, вырезаемую из РНК-предшественника, который формирует SL (spliced leader) мяРНП (малый ядерный рибонуклеопротеин).
Сигнал транс-сплайсинга адресован к SL мяРНП, а также к компонентам сплайсосомы, включая мяРНП U2, U4, U5 и U6. С их помощью через промежуточные лассо-подобные структуры аутрон замещается частью SL мяРНП (Рис.1). Транс-сплайсинг крайне эффективный механизм: практически невозможно выделить пре-мРНК, содержащие аутрон (так что последовательности аутронов малоизвестны).
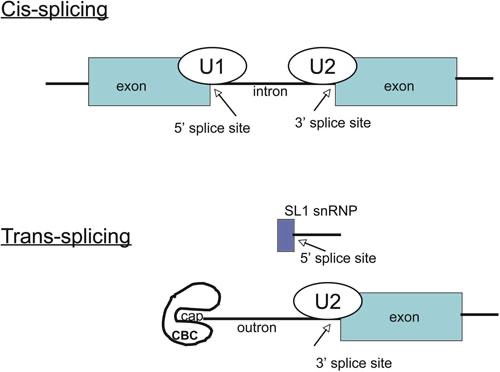
Рис.1 Сравнение цис- и транс-сплайсинга. ( DOI: 10.1895/wormbook.1.5.2 )
*всё представленное ниже было полученно с помощью скрипта, который был создан при помощи скрипта Петра Милейко.
Последовательность Козак – это крестность ATG кодона, являющаяся сигналом старта транскрипции.
В результате работы вышеупомянутого скрипта была получена позиционная весовая матрица (табл.1)
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.389 | -0.166 | -0.293 | -0.293 | 0.507 | 0.142 | -0.340 | 1.218 | -5.691 | -5.691 | -0.166 | -0.206 | -0.551 |
| T | 0.506 | 0.158 | 0.158 | 0.741 | -0.312 | 0.476 | 0.590 | -5.327 | -5.327 | -5.327 | -0.076 | 0.535 | -0.187 |
| G | 0.024 | 0.535 | 0.506 | -0.025 | 0.275 | -0.076 | 0.506 | -5.327 | -5.327 | 1.582 | 0.688 | -0.455 | 0.808 |
| C | -0.166 | -0.612 | -0.389 | -0.676 | -1.187 | -0.745 | -1.305 | -5.691 | 1.218 | -5.961 | -0.612 | -0.052 | -0.389 |
| обучение | положительный контроль | отрицательный контроль | |
|---|---|---|---|
| сигнал + | 53 | 108 | 41 |
| сигнал - | 47 | 92 | 159 |
Таблица 2. Матрица результатов проверки
Далее была получена матрица информационного содержания (таблица 3) и с использованием сервиса WebLOGO 3 построено LOGO(рисунок 3). Анализируя LOGO, можно заметить,что последовательности длиной 7 нуклеотидов до ATG и 3 нуклеотида после имеют значимый информационный вес, однако они не так значимы, как старт-кодон.
| letter | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | -0.112 | -0.060 | -0.093 | 0.359 | 0.070 | -0.103 | 1.761 | 0.000 | 0.000 | 0.000 | -0.060 | -0.071 | -0.135 |
| T | -0.060 | -0.141 | -0.112 | -0.146 | -0.154 | -0.151 | -0.151 | 0.000 | 1.761 | 0.000 | -0.141 | -0.021 | -0.112 |
| G | 0.007 | 0.270 | 0.248 | -0.007 | 0.107 | -0.021 | 0.248 | 0.000 | 0.000 | 2.286 | 0.386 | -0.085 | 0.536 |
| C | 0.248 | 0.055 | 0.055 | 0.460 | -0.068 | 0.227 | 0.315 | 0.000 | 0.000 | 0.000 | -0.021 | 0.270 | -0.046 |
Таблица 3. Матрица IC для последовательности Козак Homo sapiens
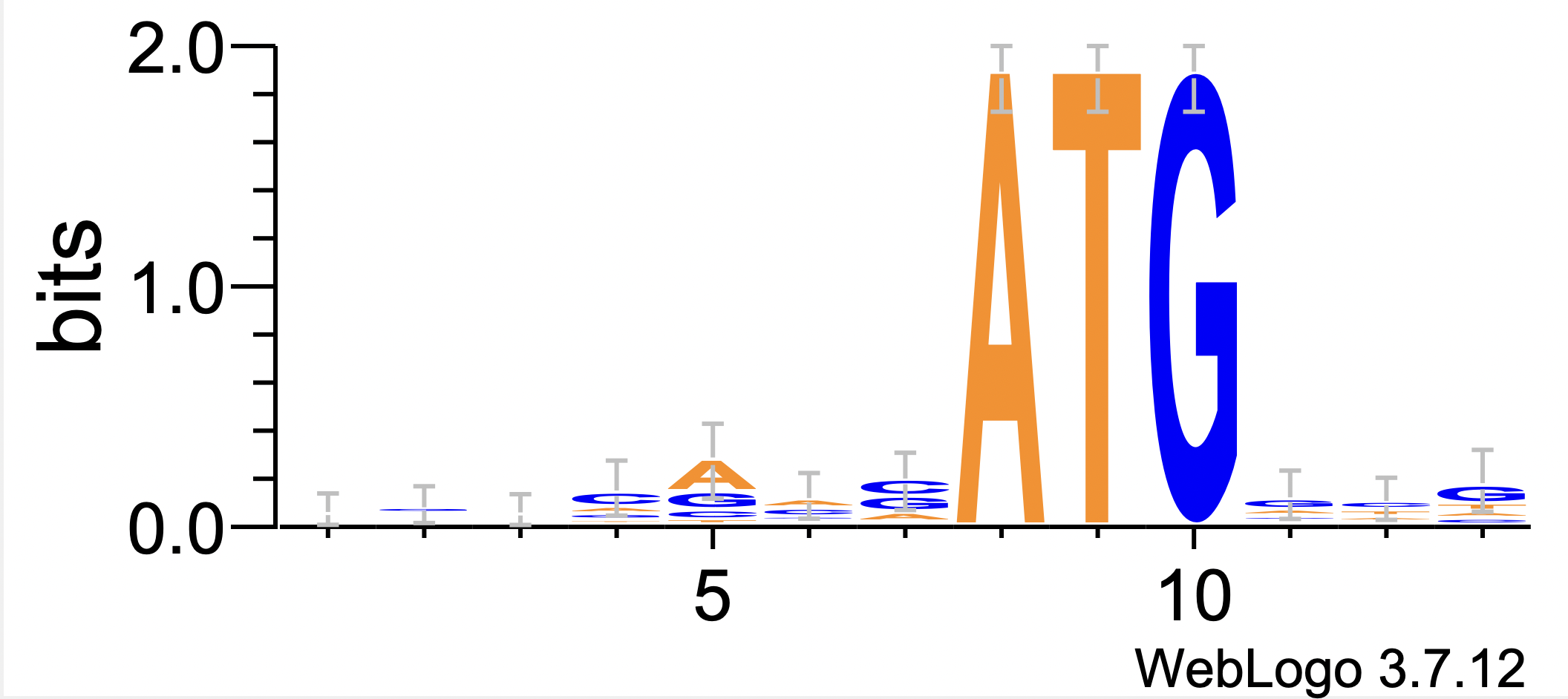
Рисунок 3. Визуализация ИС последовательности Козак Homo sapiens
Для выполнения этого задания я выбрала штамм Escherichia coli str. K-12 substr. MG1655. В геноме получен 646 сайт последовательности GAATTC. Ожидаемое число этих сайтов составило 1141. Для оценки различий использовалось биномиальное распределение. Различие оказалось статичстически значимым ( p-value = 1.77e-49).