BLASTТекстовое описание:В работе изучается поиск гомологичных последовательностей и их попарное выравнивание.
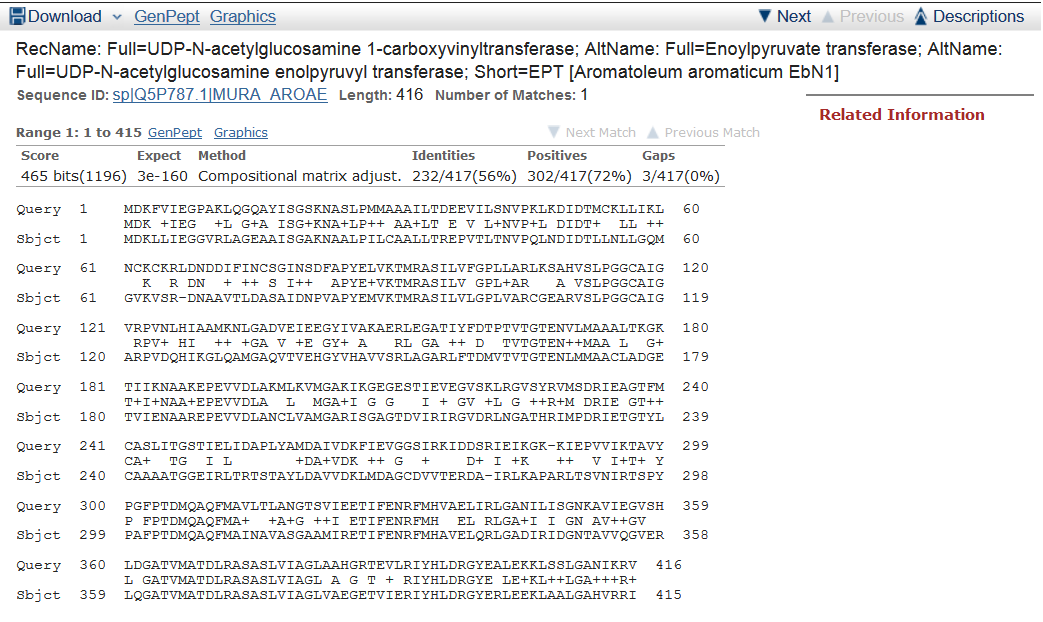
1.На сетевом ресурсе NCBI в разделе BLAST был произведён поиск гомологов белка UDP-N-ацетилглюкозамин-карбоксивинилтрансферазы, вырабатываемого бактерией Hippea Maritima (ссылка на параметры поиска). Найдено 654 последовательности, среди которых 14 организмов принадлежат к таксону "Археи", а все остальные - к таксону "Бактерии". Сравнительная таблица:
Выравнивания:
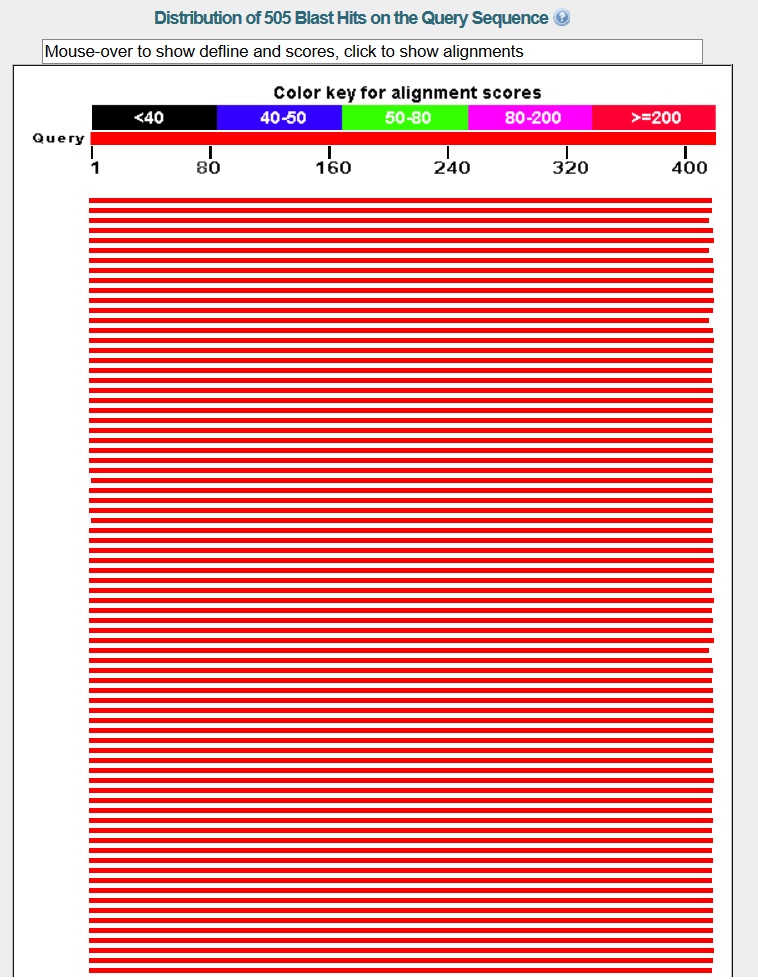
По критерию E-value < 1e-3 и Query cover > 70% проходят 491 последовательность из 654 найденных, графическое представление:
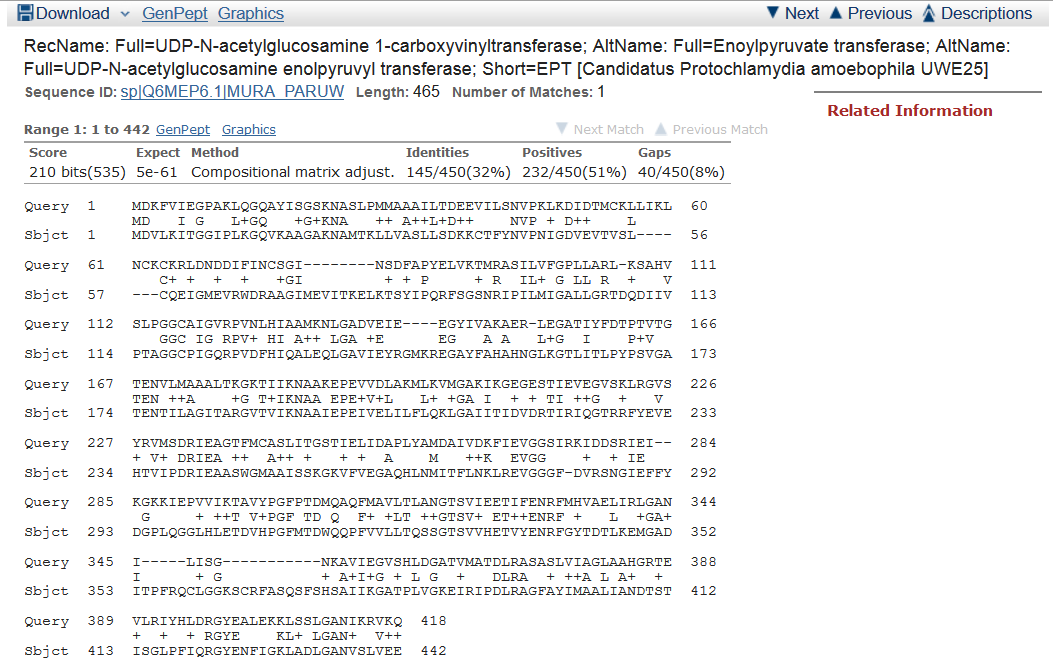
2.Для поиска выбран таксон "green sulfur bacteria" - "зелёные сульфобактерии" (ссылка на параметры поиска). Найдено 10 гомологичных последовательностей, среди которых Chlorobaculum parvum NCIB 8327 c e-value = 2e-126 встречается также в п.1 c e-value = 4e-124. В пункте 1 объём выборки больше, следовательно, e-value будет также больше.Поиск среди зелёных сульфобактерий: 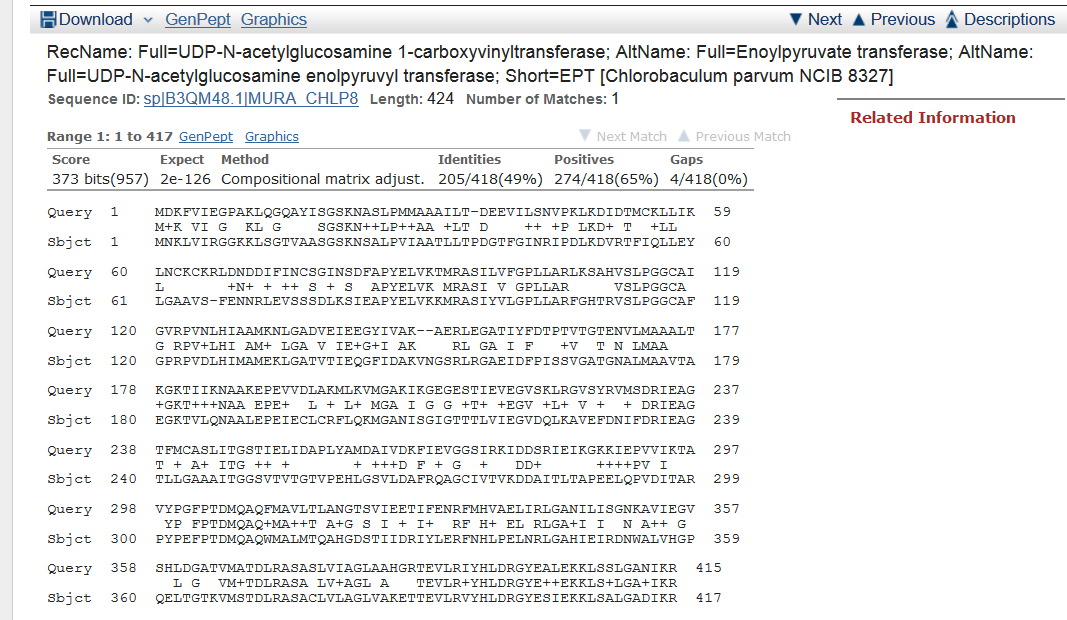 Поиск по всем таксонам: 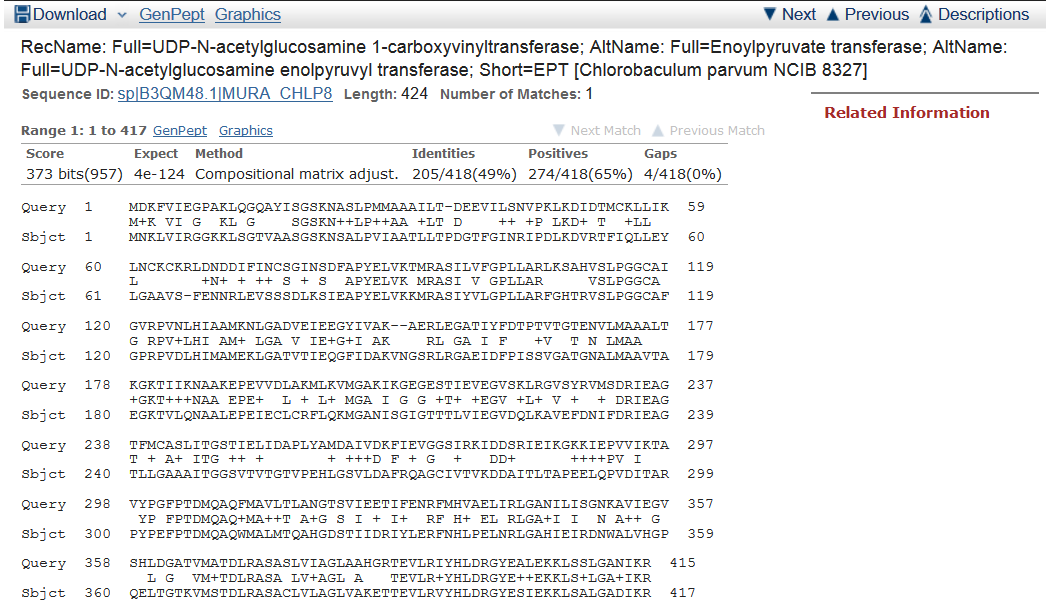
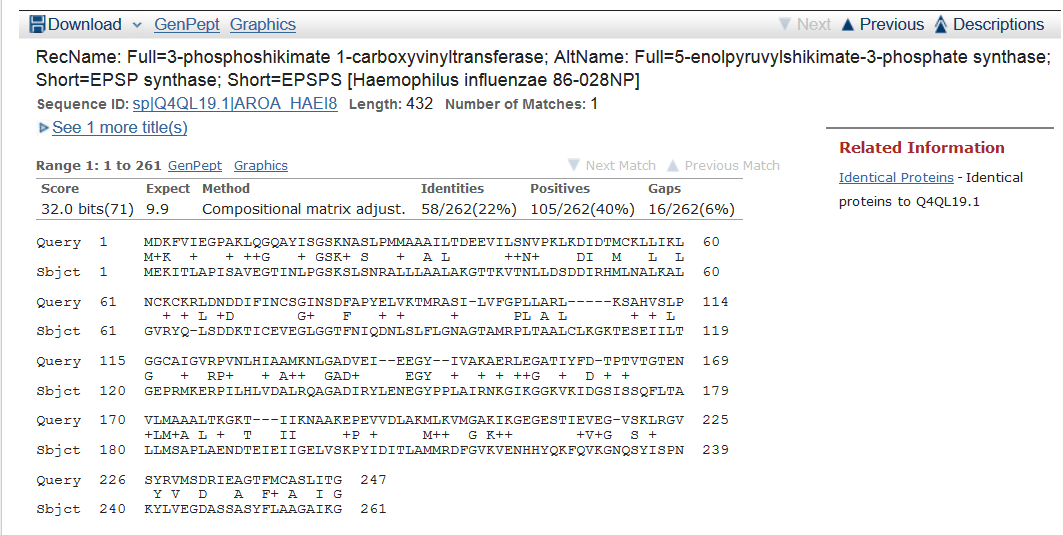
3.Для выравнивания выбраны последоватеьность белка-образца в пункте 1 и худшего по результатам поиска его гомологов белка EPSP-синтазы, вырабатываемого Haemophilus influenzae 86-028NP. В результате построено 4 выравнивания длинами в:262 позиции: 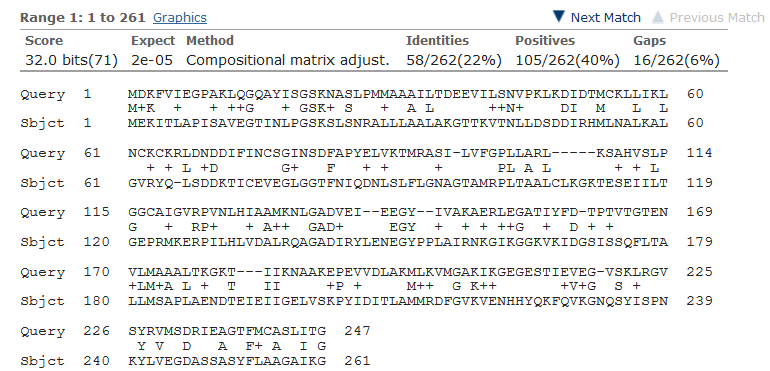 139 позиций: 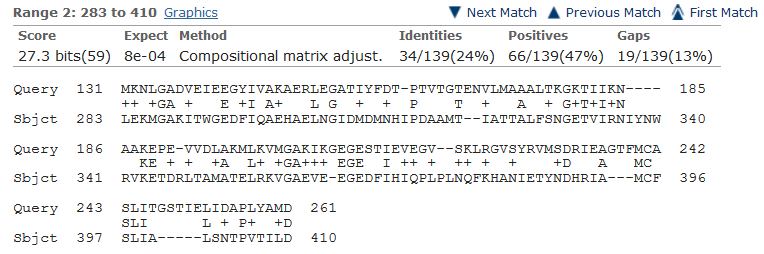 114 позиций: 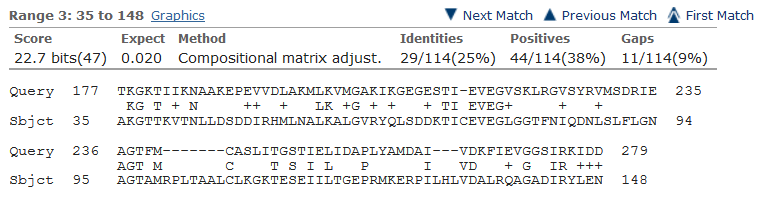 48 позиций: 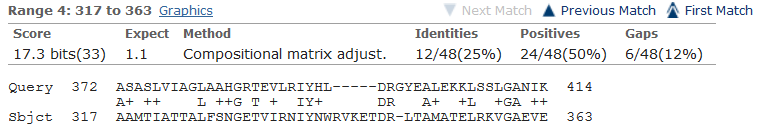 Карта локального сходства для полученного выравнивания (4 графика, каждый из которых представляет собой одно из четырёх выравниваний, ось абсцисс представляет последовательность белка Hippea Maritima, ординат - Haemophilus influenzae): 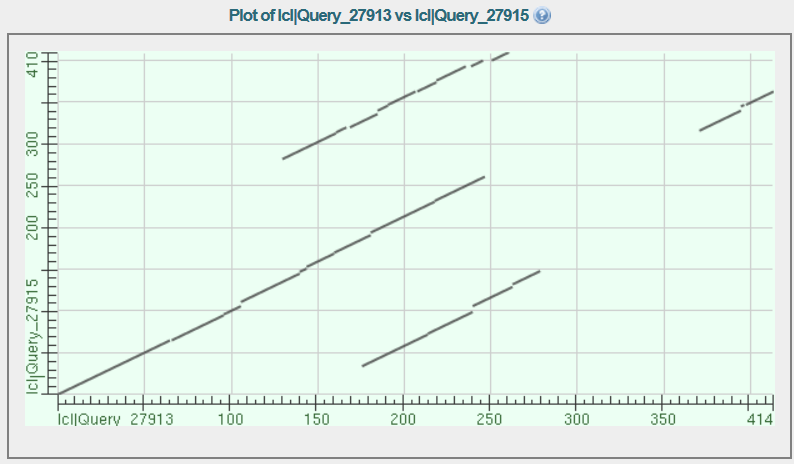
4.Для создания банка последовательностей взято выравнивание из практикума №8, в котором посредством jalview удалены все гэпы. Cервис blastp сумел построить два выравнивания, среди которых лучшее в силу их экстремально малой длины можно выбрать только по Bit-score. Информация о выравниваниях:
|