Структуры нуклеиновых кислот: X3DNA, JMol
1. X3DNA
Для работы с пакетом подключаем его в переменные окружения:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Построение А-спирали:
fiber -a gatc-a.pdb //(повторяющийся модуль: gatc, число повторов: 5)
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb //(создаст структуру из последовательности по умолчанию)
Полученные файлы:
gatc-a.pdb
gatc-b.pdb
gatc-z.pdb
2. Отображение структуры в JMol
Упражнение 1
Сценарий для отображения: ex2.spt
Скриншоты:
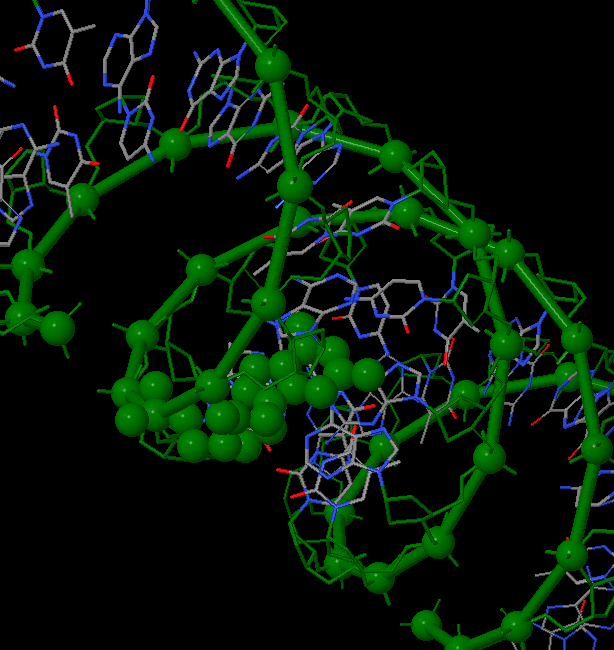
Сахарофосфатный остов ДНК:
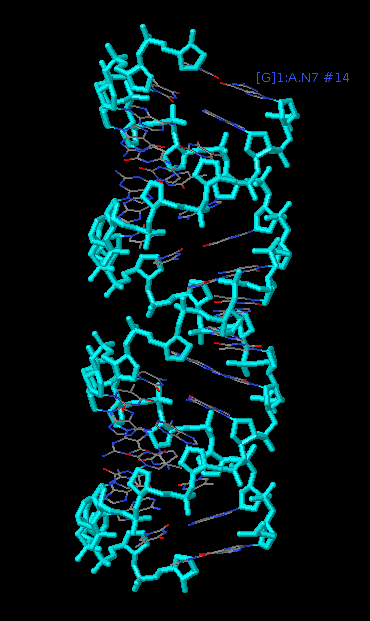
|
Все нуклеотиды:
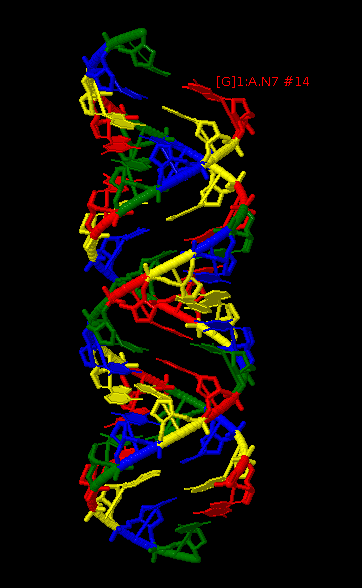
|
Нуклеотиды с аденином:
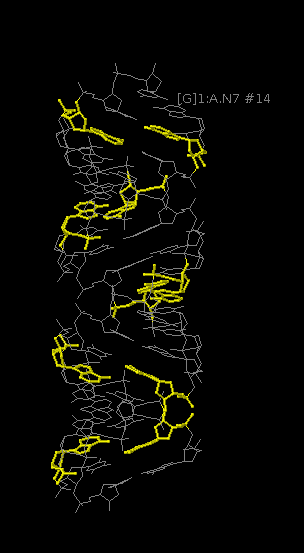
|
Атом N7 в гуанинах:
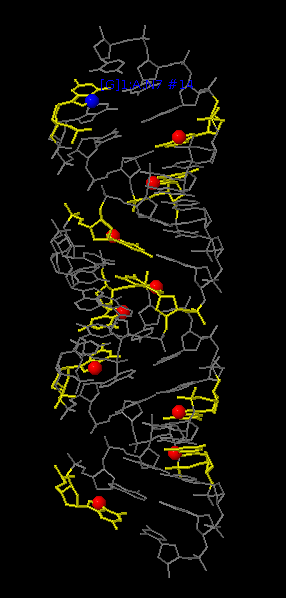
|
Упражнение 2
Полученные файлы:
1N77.pdb(Асимметричная единица)
1A02.pdb
Упражнение 3
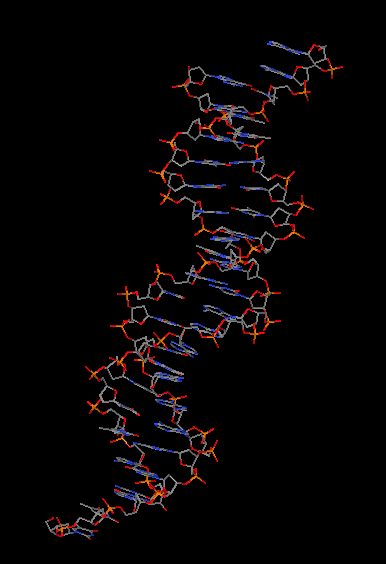
Структура ДНК разрывов не содержит:
Структура РНК также не содержит разрывов:
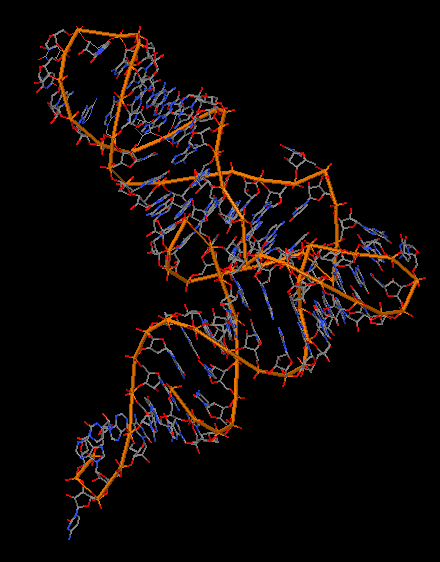
Сравнение 3х форм ДНК
Упражнение 1:
Азотистое основание - гуанин.
Ориентация атомов (красным - к большой бороздке, синим - к малой):
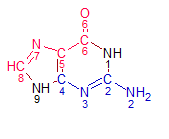
| Форма спирали | К большой бороздке | К малой бороздке | Не ориентированы |
|---|
| A |
[G]33:B.C8; [G]33:B.N7; [G]33:B.C5; [G]33:B.C6; [G]33:B.O6; [G]33:B.N1 |
[G]33:B.C4; [G]33:B.N3; [G]33:B.C2; [G]33:B.N2; |
[G]33:B.N1; [G]33:B.N9 |
 |
| B |
[G]33:B.C8; [G]33:B.N7; [G]33:B.C5; [G]33:B.C6; [G]33:B.O6; [G]33:B.N1 |
[G]33:B.C4; [G]33:B.N3; [G]33:B.C2; [G]33:B.N2 |
[G]33:B.N1; [G]33:B.N9 |
 |
| Z |
[G]33:B.C8; [G]33:B.N7; [G]33:B.C5; [G]33:B.C6; [G]33:B.O6; [G]33:B.N1 |
[G]33:B.C4; [G]33:B.N3; [G]33:B.C2; [G]33:B.N2 |
[G]33:B.N1; [G]33:B.N9 |
 |
Упражнение 2
Для определения характеристик спирали измерялись расстояния между атомами фосфора. Результаты:
| Характеристика | А-форма | В-форма | C-форма |
|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали | 28,03 | 33,75 | 43,50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,98 ([G]25:B.P-[A]10:A.P) |
17,21 ([A]26:B.P-[C]12:A.P) | 18,3 ([C]14:B.P-[C]4:A.P) |
| Ширина малой бороздки | 16,8 ([T]11:A.P-[C]32:B.P) |
11,69 ([A]26:B.P-[T]19:A.P | 9,87([C]10:A.P-[G]15:B.P) |
Упражнение 3
Измерение углов производилось в JMol (settings->torsion). Результаты:
| Угол | А-форма | А-форма (презентация) | В-форма | В-форма (презентация) |
| Α | -51,7 | 62 | -29,9 | 63 |
| β | 174,8 | 173 | 136,3 | 171 |
| γ | 41,7 | 52 | 31,2 | 54 |
| δ | 79,1 | 88/3 | 143,3 | 123/131 |
| ε | -147,8 | 178 | -140,8 | 155 |
| ζ | -75,1 | -50 | -160,5 | -90 |
| χ | -157,2 | -160 | -98 | -117 |
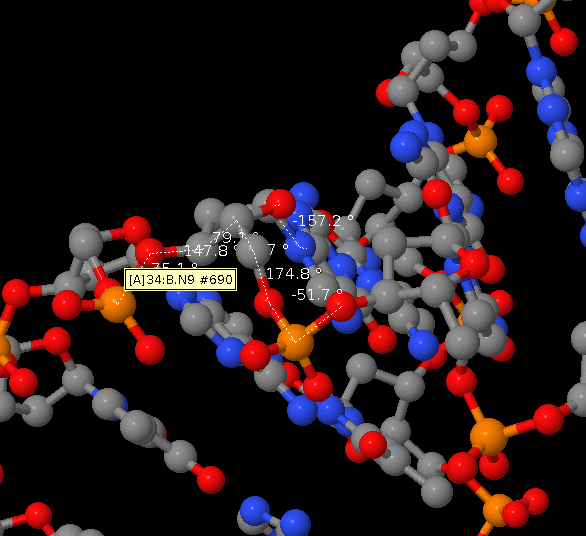
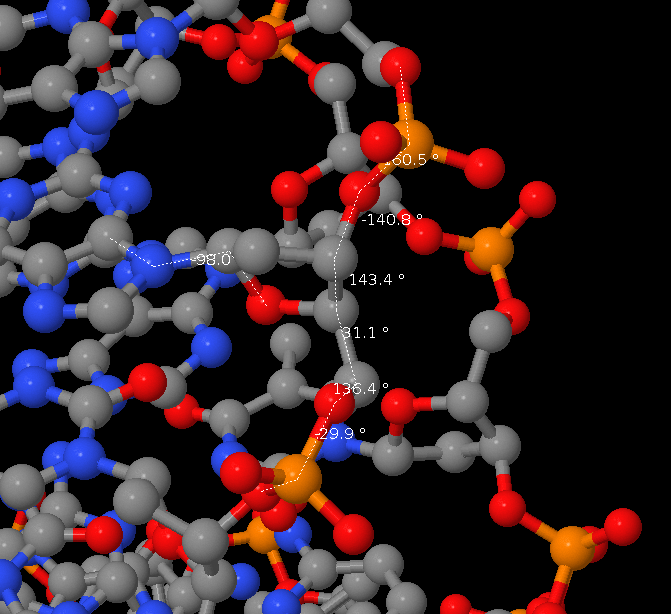
Определение параметров нуклеиновых структур
Получение данных:
remediator --old '1N77.pdb' > 1N77_old.pdb #переводим в старый формат для X3DNA
remediator --old '1A02.pdb' > 1A02_old.pdb
find_pair -t 1N77_old.pdb stdout | analyze
find_pair -t 1A02_old.pdb stdout | analyze
ex_str -2 stacking.pdb step2.pdb #получаем пару с самым большим перекрытием
stack2img -cdolt step2.pdb step2.ps #изображение этой пары
Данные-результаты выполнения этого пункта собраны в Tors.xls
Упражнение 1
В tRNA наиболее деформированный 11 G, сразу по трем углам: γ, ε и ζ.
Нужно отметить, что почти все нуклеотиды, на которых сменятся направление цепи, немного деформированы.
В DNA наиболее деформированный 4 A первой цепи, по χ, ε и ζ. В целом значения углов очень разбросаны и сильно отличаются друг от друга.
Упражнение 2
Средние значения торсионных углов:

Интерпретация структуры тРНК:
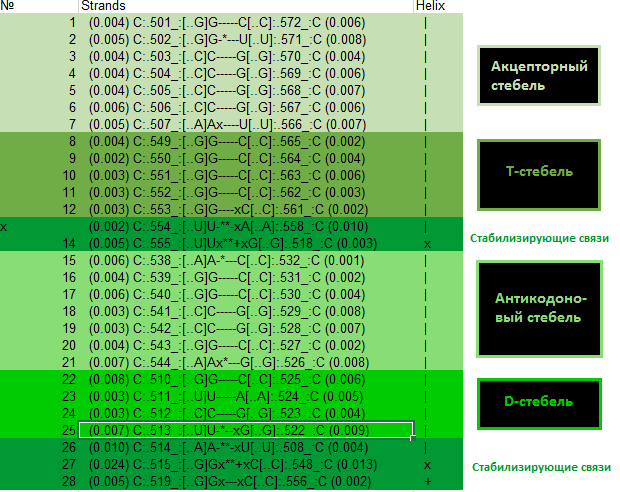
Неканонические взаимодействия: 2, 14, 15, 21, 25
Упражнение 3
Стекинг-взаимодействие:
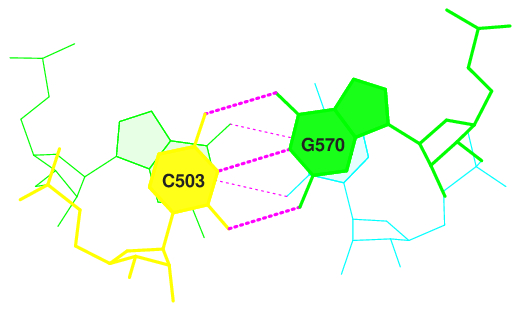
|














