Нуклеотидные банки данных
Текстовое описание:
1.Геном собаки
(Они ненавидят меня, ведь вместо весёлых прогулок мы мучительно делаем биоинформатику)


В качестве организма выбран Canis lupus familiaris: отличается огромной вариативностью фенотипа. Поиск в базе Genomes выдал две сборки, одна - вплоть до хромосом (боксёр, сука), вторая - на уровне контигов (пудель, сука), однако поиск в genome assembly выдаёт ещё и третью сборку, также на уровне контигов (бигль, сука).
Среди ссылок на ресурсы по геному собаки были найдены три "genome project", на один из которых шла переадресация (видимо, в связи с отсутствием страницы), остальные доступны по ссылкам: The NHGRI dog genome project и dog genome project at BROAD institute/
Проекты по секвенированию генома собаки, найденные в базе NCBI: (условия поиска - ("Canis lupus familiaris"[Organism] OR dog[All Fields]) AND "genome sequencing"[All Fields])
- Canis lupus familiaris
- A collection of grey wolves and dogs Genome sequencing
- Mizzou Comparative Canine Resequencing
- Canis lupus familiaris strain:Chinese Crested Genome sequencing
- Whole Genome Sequencing of Korean Jindo Dog
- Canis lupus familiaris Genome sequencing
- Canis lupus familiaris strain:Village Dogs Genome sequencing
- Canis lupus familiaris
- Canis lupus familiaris strain:Norwegian Elkhound
- Canis lupus familiaris Genome sequencing
- Canis lupus familiaris
- Canis lupus familiaris
- Whole genome sequencing of 12 breeds of Canis lupus familiaris
- Canis lupus familiaris strain:Norwegian Lundehund
- Canis lupus familiaris beagle Genome Sequencing
- Boxer 7.6x whole genome shotgun assembly by the Dog Genome Sequencing Consortium
- Canis lupus familiaris (dog) Reference genome sequence
В качестве описываемой сборки взята наиболее полная. Организм, из которого получен образец: Собака домашняя породы Боксёр, сука. Геном собаки состоит из 38 аутосом и 39-ой половой хромосомы. Образец один, предоставлен Agencourt Bioscience Corporation, 2014-08-06 и использовался в двух проектах(две последние ссылки списка). Метод секвенирования - по Сэнгеру, уровень сборки - хромосомы, геном представлен полностью. Детальная информация о сборке на скриншоте:
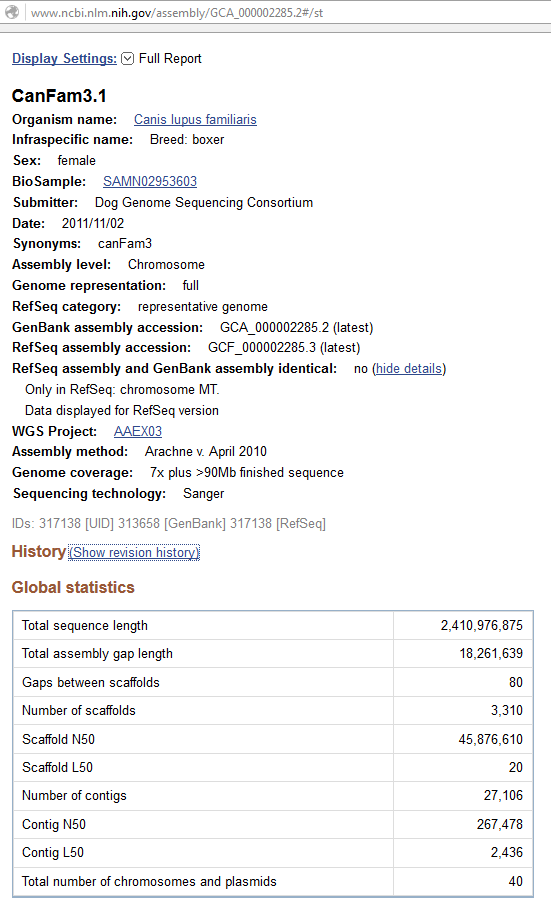
Информация о длинах скэффолдов и покрытии на каждой хромосоме:
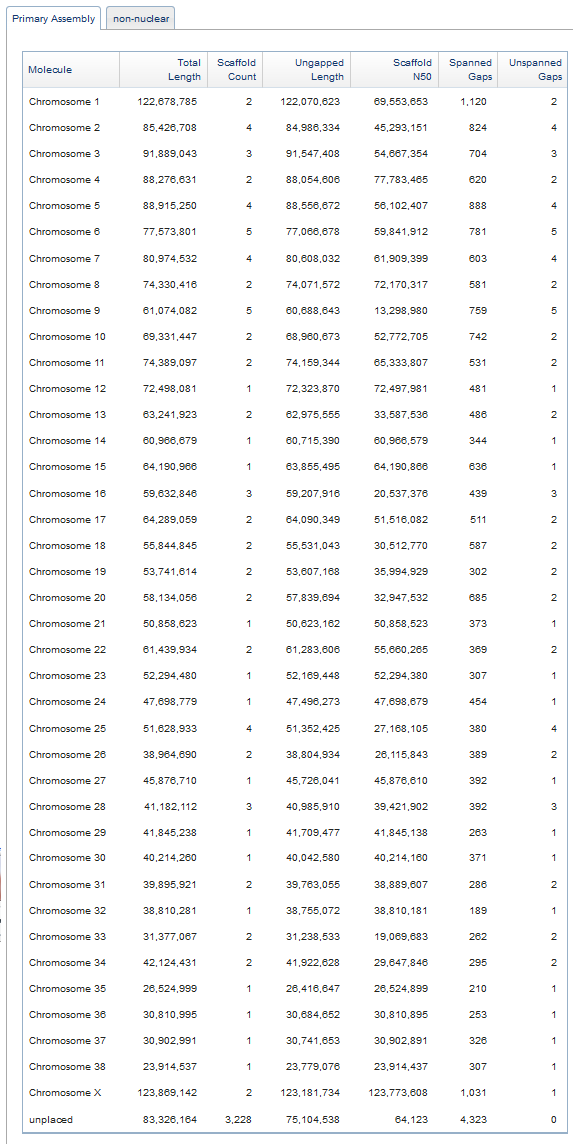
Для получения генома было получено чуть более 27 тысяч контигов (Таблица контигов ). Упорядочив вывод по длине, увидим, что максимальная длина контига составила 2428071, минимальная - 136 баз. ( самый короткий контиг )
2.Мох Physcomitrella patens

Отдел Бриофитовые, используется в качестве модельного организма благодаря быстрой смене поколений и преимущественно гаплоидному жизненному циклу.
По запросу "Physcomitrella patens"[Organism] AND "complete genome"[All Fields] AND "mitochondrial DNA"[All Fields] выходим на митохондриальный геном, далее в Related information следуем по ссылке Genome к описанию организма, где в таблице Replicon info в строке MT приведена сводная информация по митохондриону мха. Митохондрион размером 105,34Кб кодирует 42 белка, 3 рРНК, 24тРНК - суммарно 69 генов.
Пройдя от митохондриального генома по другой ссылке - Gene - в Related info и применив сортировку по хромосоме, получим таблицу из 69 генов митохондриона, упорядоченных по местоположению в нём. (список генов)
3. Ключи раздела "особенности"
Перейдя в раздел "NCBI help manual" и выполнив поиск по "features table keys", выйдем через несколько ссылок на определения таблицы особенностей, где в разделе 7.2 Appendix II: Feature keys reference описаны искомые ключи. Копипаста! перевод части раздела:
-
assembly_gap - гэп между двумя компонентами сборки транскриптома или генома.
Забавно, ключ описан в стандарте, но не находится в базе по запросу assembly_gap[Feature key]
-
attenuator - регуляторная область, контролирующая экспрессию некоторых бактериальных оперонов либо участок последовательности между промотором и первым структурным(?) геном, вызывающий частичную отмену транскрипции.
Был отменён 15 декабря 2014 года, взамен используется ключ regulatory в следующем виде:
regulatory 1041893..1042065
/regulatory_class="attenuator"
/note="Lys leader"
-
centromere - область биологического интереса, определённая как центромера и охарактеризованная экспериментально.
centromere 555957..556073
/note="CEN16; Chromosome XVI centromere"
/db_xref="SGD:S000006477"
-
D-loop - петля смещения, область внутри митохондриальной ДНК, в которой короткий участок спарен с одной цепочкой ДНК, вытесняя комплементарную ей цепь. Также используется для описания вытеснения одной из цепей дуплекса ДНК другой одноцепочечной последовательностью в реакции, катализируемой белком RecA
D-loop 14887..15205
/note="putative control region; A+T rich; contains origin
of replication"
-
exon - область генома, кодирующая участки остающиеся при сращивании мРНК, рРНК и тРНК. Может содержать 5' и 3'-нетранслируемые области и все кодирующие последовательности.
exon 101..1411
/gene="SOX30P1"
/inference="alignment:Splign::RefSeq|NM_001308165.1"
/pseudo
-
iDNA - промежуточная ДНК, ликвидируется при любой рекомбинации.
iDNA 1..2377
/gene="J-beta-2.1"
-
intron - Участок ДНК, удаляющийся после транскрипции посредством сращивания между собой других участков, экзонов, расположенных по обеим сторонам от него
intron <1..>688
/gene="Timm44"
/number=2
-
LTR - длинный концевой повтор, последовательность, прямо повторяющаяся на обоих концах определённого участка, вроде той, что обычно находят в ретровирусах
LTR <1..545
/note="5'LTR"
-
misc_binding - сайт нуклеиновой кислоты, ковалентно или нековалентно связывающий другой фрагмент, который нельзя описать другим ключом связывания (primer_bind или protein_bind)
misc_binding complement(41465..41660)
/note="cobalamin riboswitch; Derived by automated
computational analysis using gene prediction method:
cmsearch."
/bound_moiety="adenosylcobalamin"
-
ncRNA - ген, кодирующий не белок и не какую-либо из обычных РНК - рибосомную или транспортную,но функциональная молекула которого является РНК-транскриптом.
ncRNA 72629..72982
/ncRNA_class="RNase_P_RNA"
/locus_tag="TU73_RS16255"
/product="bacterial RNase P"
/note="Derived by automated computational analysis using
gene prediction method: cmsearch."
4. Сервис BLASTn для последовательности из №6
В
standart nucleotide BLAST задаём сначала поиск по прямой последовательности и упорядочиваем выдачу по идентичности, т.к. покрытие с учётом того, как получена последовательность, играет меньшую роль в доказательстве сходства. Первым в списке идёт ген гистона H3 организма Brada Inhabilis voucher WS1017 с идентичностью 99% и практически нулевым (exp(-178)) e-value:
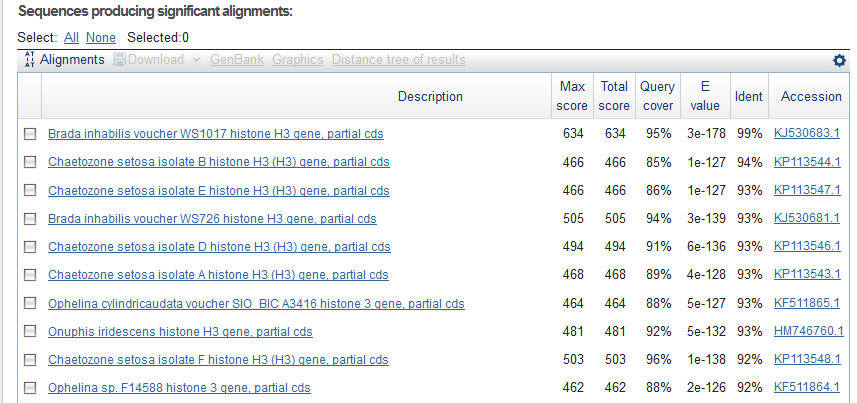
Поиск по обратной цепи выдаёт первым его же, но с идентичностью 100%:
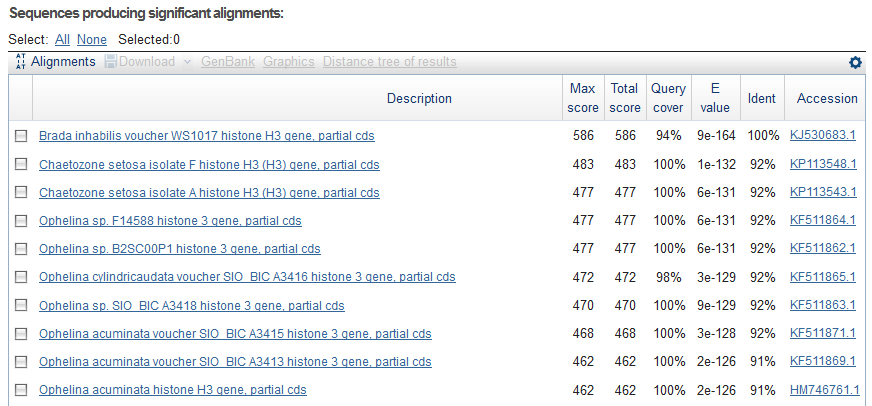
Древо сходства гена:
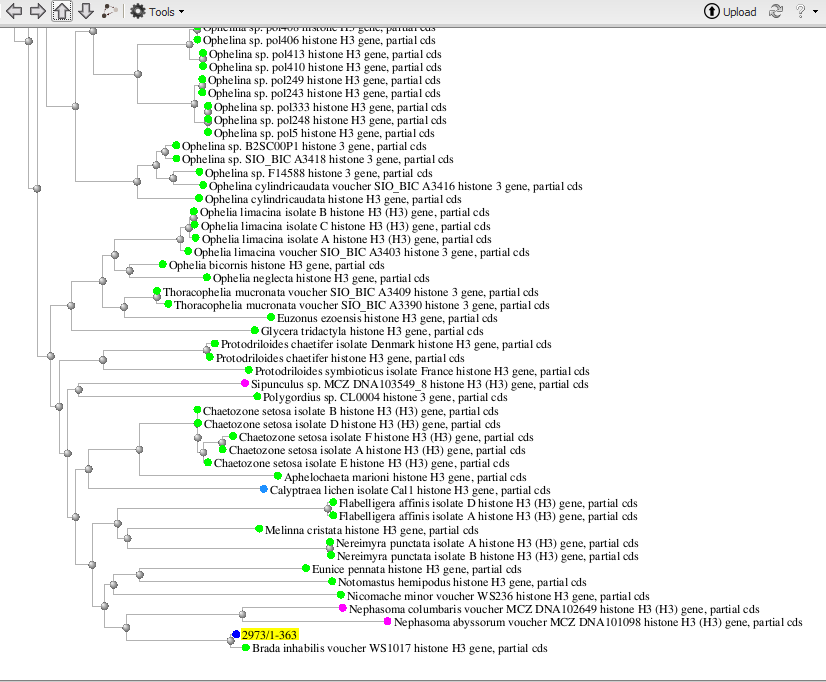
Возьмём соседей по выдаче из разных таксонов внутри полихет (cellular organisms; Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Annelida; Polychaeta): Brada inhabilis, Chaetozone setosa и Protodriloides chaetifer, у всех найдённые гены кодируют гистон H3.
| Организм |
Таксономия |
CDS (fasta) |
AAC sequence (fasta) |
| Brada inhabilis |
Palpata; Canalipalpata; Flabelligerida; Flabelligeridae; Brada |
H3_gene |
H3_proteine |
| Chaetozone setosa |
Scolecida; Spionida; Cirratulidae; Chaetozone |
H3_gene |
H3_proteine |
| Protodriloides chaetifer |
Polychaeta incertae sedis; Protodrilidae; Protodriloides |
H3_gene |
H3_proteine |
Результаты выравнивания CDS с помощью muscle в JalView (сверху вниз: исходная, inhabilis, setosa, chaetifer):
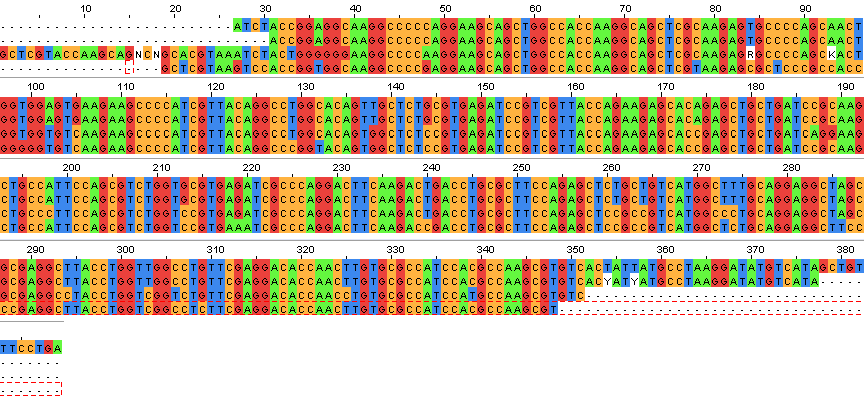
С 31-го по 377-ой нуклеотид у последовательности из №6 и Brada inhabilis несовпали только 2 неопознанных секвенатором нуклеотида (354 и 356), два других содержат около 25 нуклеотидных замен на ~320 нуклеотидов, то есь заменяется 1 из 13 в среднем.
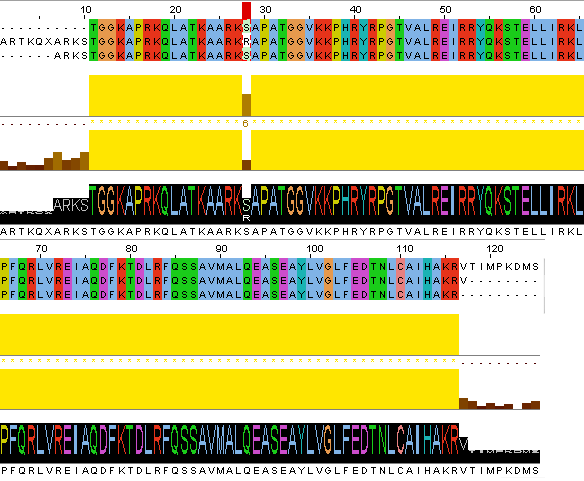
Выравнивание аминокислотных последовательностей белков-продуктов этих генов (сверху вниз: inhabilis, setosa, chaetifer):
Древо сходства по результатам выравнивания:
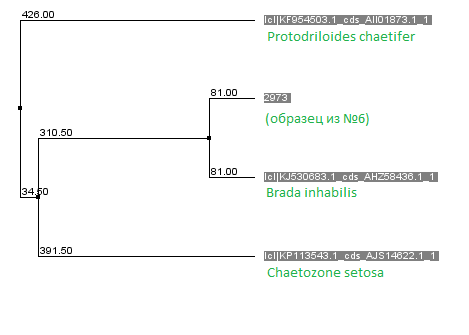
Вывод: ген, определённый по хроматограмме в №6, кодирует гистон H3 в организме многощетинкового червя, это Brada inhabilis. (с хорошей степенью достоверности, хотя исследование (стр. 61) по данному виду отмечает большую вариативность образцов гена Н3, чем допускается в рамках одного вида)

|











