Предсказание генов эукариот
1. Предсказание генов X5 с помощью AUGUSTUS
Выбор контига
Мне показалось утомительным логиниться на сервер, запускать инфосек или секрет, поэтому я просто открыла X5.fasta в notepad++, вышла к середине файла и выделением, копированием и вставкой сделала себе скэффолд 489 (выделение текста показало 26929 символов) в качестве исследуемой последовательности. Длина последовательности составила 26929(символов всего)-441(символов конца строки)=26488 нк. scaf489.fasta
Выбор образца для подражания
Образец должен подходить по составу кодируемых белков с учётом того, что организм у нас эукариотический, а, значит, исходная последовательность содержит интроны. BLASTX умеет производить поиск с учётом интронных вставок, поэтому используем его. Результаты выравнивания:
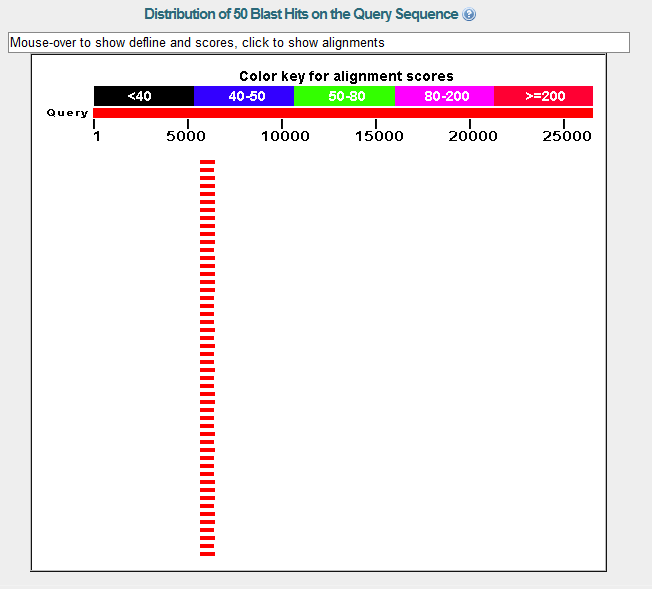
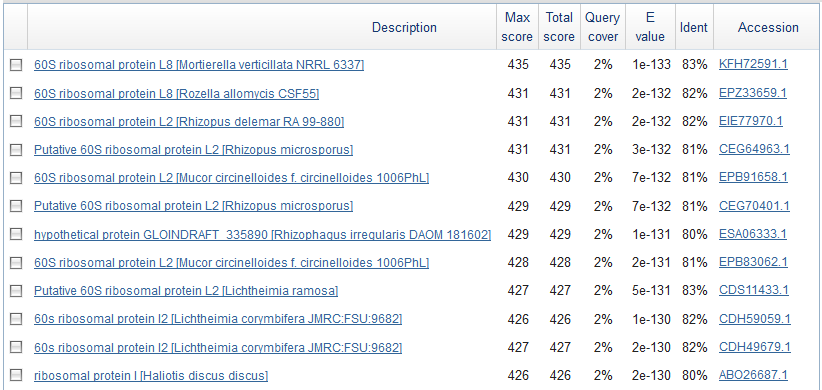
Лучшие места заняли представители рода Rhyzopus, по счастью, представитель этого же рода - Rhizopus oryzae (в хелпе сайта AUGUSTUS очепятка, он там Phizopus oryzae) - нашёлся в качестве эталона на AUGUSTUS.
Работа с AUGUSTUS
Заявка на предсказание подана со следующими параметрами:
-
AUGUSTUS species parameters: в выпадающем списке задан Phizopus oryzae
-
Report genes on: both strands
-
Alternative transcripts: only predict complete genes
-
Allowed gene structure: only predict complete genes, с галочкой в ignore conflicts with other strand
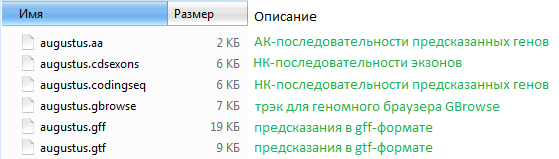
Заявка удовлетворена файлом predictions.tar.gz - архивом со следующей структурой:
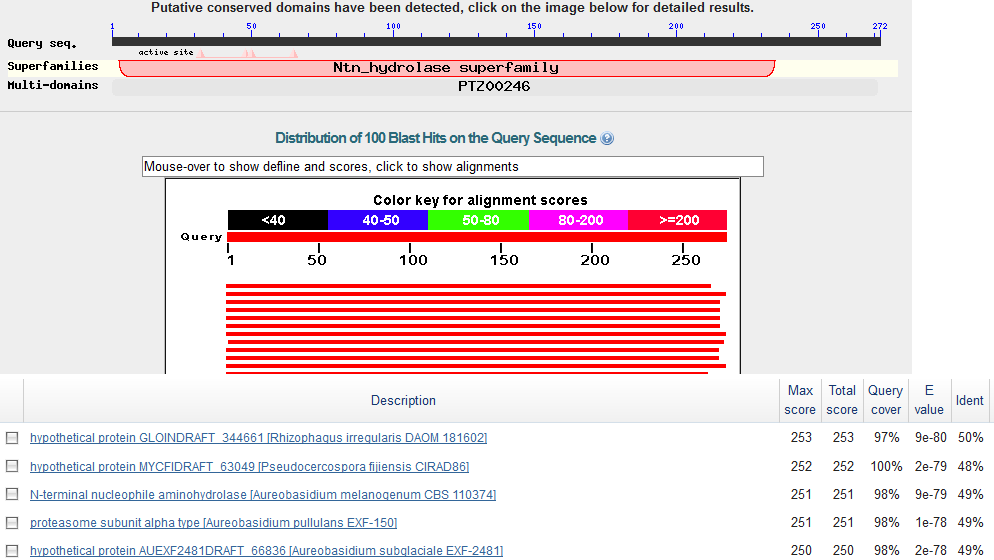
Проверка 5 предсказаний с помощью blastp
-
g1.t1: 4 экзона, кодирует N-концевую нуклеофильную аминогидролазу.
-
g2.t1: 2 экзона, кодирует фактор инициации трансляции SUI1
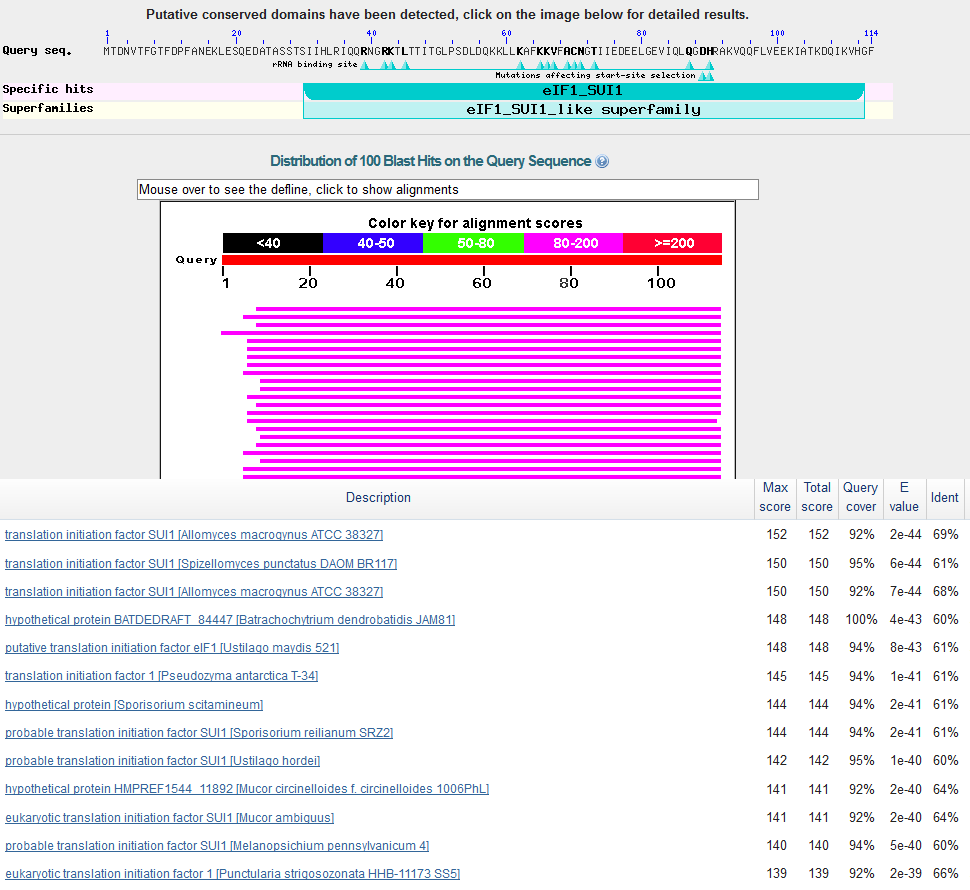
-
g3.t1: 2 экзона, кодирует белок 60s-рибосомы
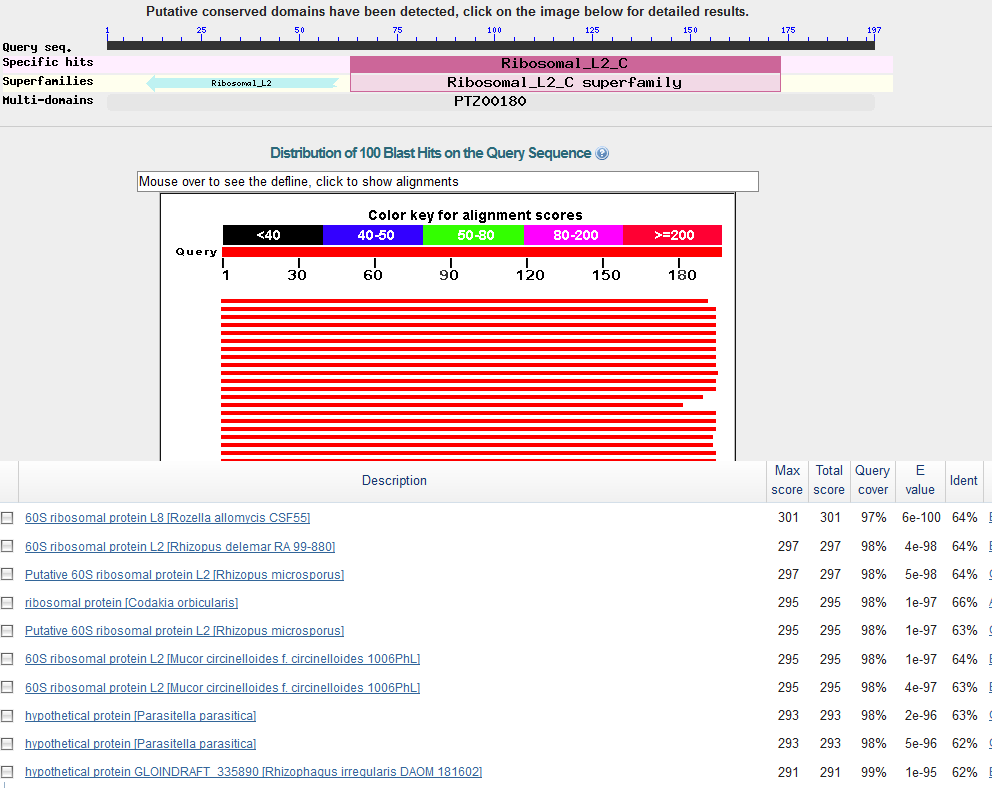
-
g4.t1: 2 экзона, однако гомологов найдено не было, следовательно, либо белка не существует, либо предсказание неверное.
-
g5.t1: 1 экзон, кодирует N-ацетилглюкозамин 1-фосфат-трансферазу, однако у большинства найденных гомологов в статусе PREDICTED, хотя попадаются и проверенные.
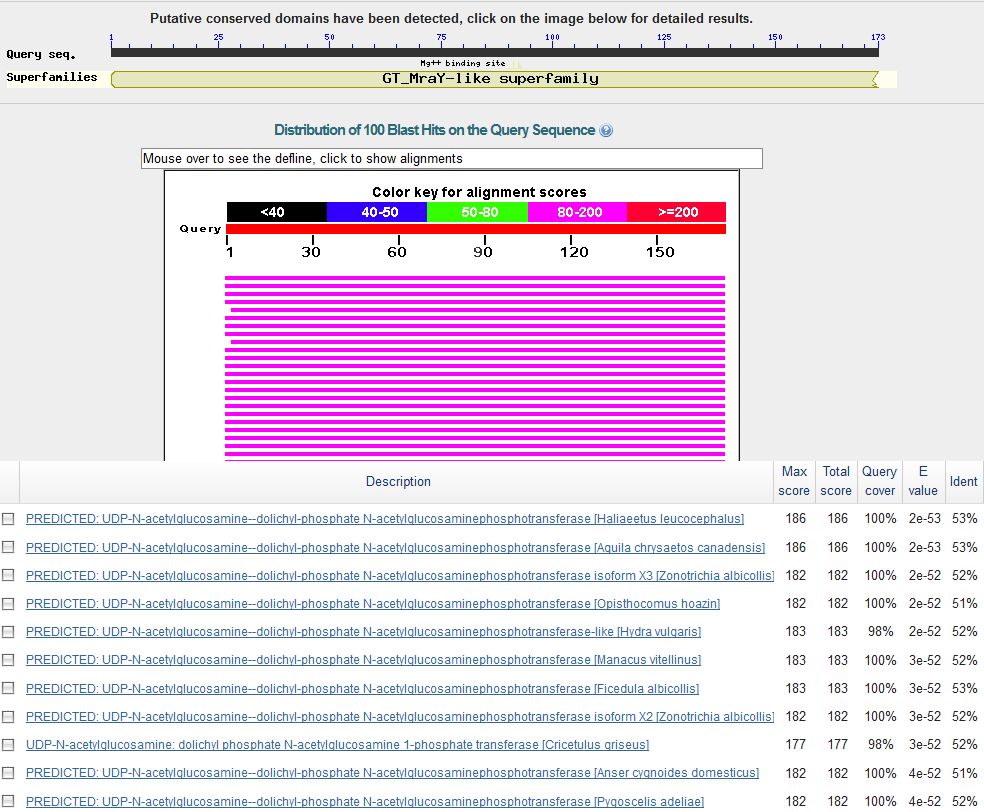
2. Сравнение аннотаций RefSeq и Augustus
Для сравнения был выбран ген tet1, отвечающий за деметилирование 5-цитозин-метилированной ДНК. Роль белка в организме человека множественная: помимо очевидного участия в опухолевых процессах (засчёт деметилирования онкогенов или деметилирования их супрессоров) также замечен в формировании памяти (замещения старых воспоминаний новыми, болезнь Альцгеймера и т.п.), и дедифференциации клеток (вплоть до превращения в стволовые) (статья Hydroxylation of 5-methylcytosine by TET1 promotes active DNA demethylation in the adult brain . Tet1 расположен на 10ой хромосоме, но локус в зависимости от сборки разный: предыдущая сборка генома человека, №37, содержит его с 70320116 по 70454238 нуклеотид:результат поиска на GenAtlas. Текущая сборка, №38 - с 68560360 по 68694487 нуклеотид:
результат поиска в NCBI
.
В геномном браузере на сайте UCSC после выбора hide во всех выпадающих списках, кроме base position (секция Mapping and Sequencing),RefSeq Genes и augustus (секция Genes and Gene Predictions) ген tet1 выглядит так:
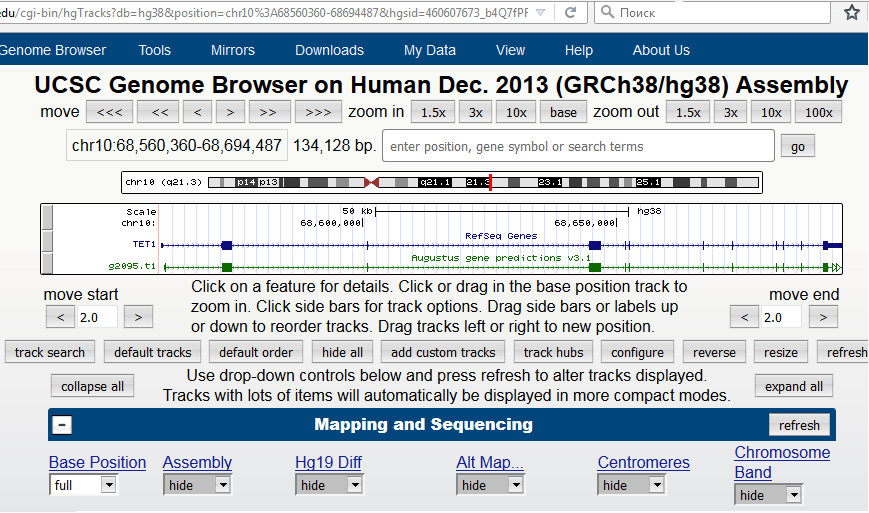
Для получения информации об аннотациях в RefSeq и AUGUSTUS воспользовалась Tools -> Table browser (полоска меню сверху сайта), где указала вручную (остальное - по умолчанию) параметры:
- position: chr10:68560360-68694487
- track: RefSeg genes (или augustus для augustus-аннотации соответственно)
- output format: selected fields from primary and related table
и скопировала выданные по кнопке get output результаты в текстовый файл с дальнейшим размещением и обработкой в excel-файле (лежит здесь)
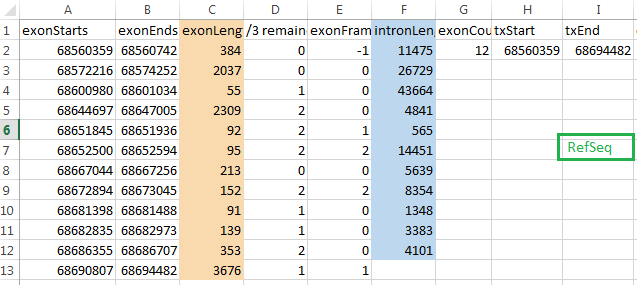
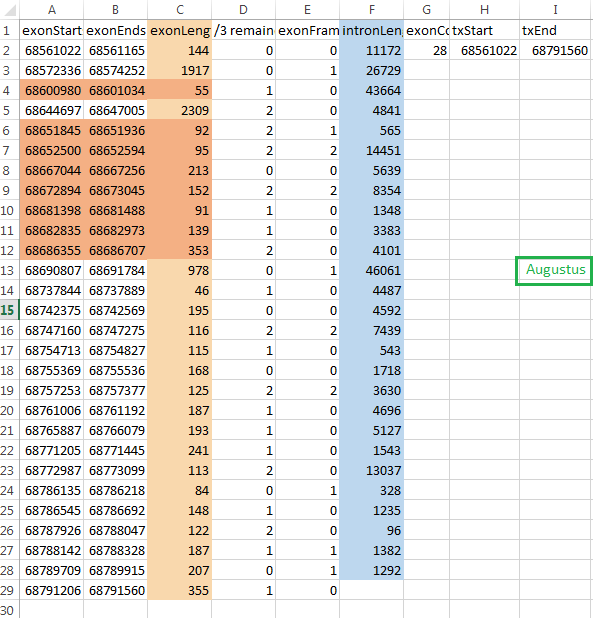
После обработки (разнесения данных по столбцам, транспонирования,вычисления длин экзонов и интронов) получилась следующая картина: Augustus выделил 28 экзонов, из них только 8 совпали с выделенными RefSeq'ом (помечены тёмно-рыжим). RefSeq нашёл 12 экзонов. В обеих аннотациях разные координаты начала и конца гена. Суммарные длины интронов и экзонов относятся как 221453:9140 у augustus и 124550:9596 у refseq, то есть ген у Августуса вышел в 1,72 раза длиннее и с вдвое большим соотношением длин интронов к длинам экзонов (24,23 против ~12)
Картинка в геномном браузере с границами видимости, установленными по augustus (ранее были по RefSeq):

|










