Филогенетические деревья, занятие 4Задание 1Нуклеотидные последовательности 16S рРНК организмов из занятия 2 (4семестр) взяты по ссылке ftp://ftp.ncbi.nlm.nih.gov/genomes/archive/old_refseq/Bacteria/ из файлов с расширением .frn и скопированы в единый fasta-файл.После выравнивания в MEGA с помощью MUSCLE и построения дерева по алгоритму Maximum likehood можно сравнить полученное по рибосомальным генам дерево: 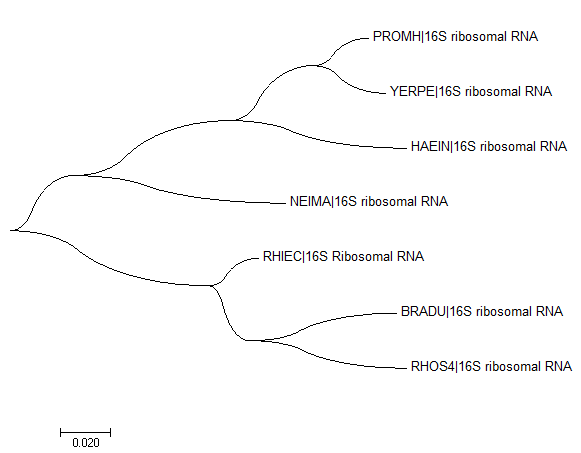 с деревом из задания 2: 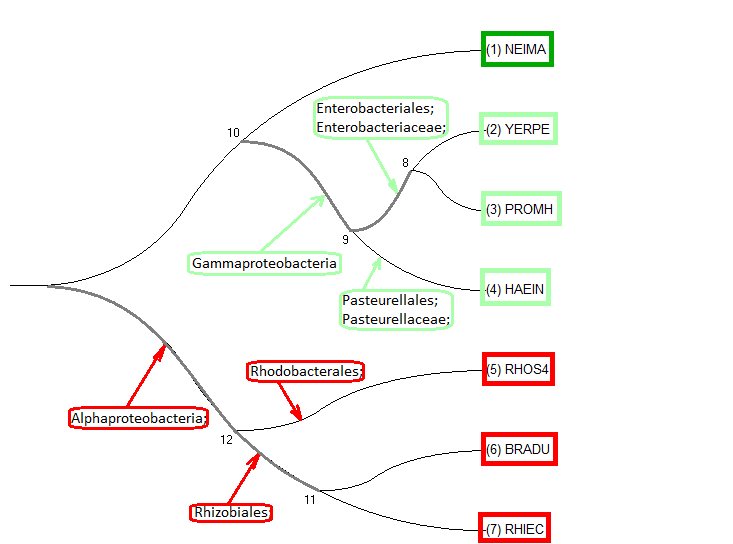 Топология ветвей для NEIMA, YERPE, PROMH и HAEIN осталась той же, однако виды BRADU и RHOS4 оказались в рРНК-версии дерева более схожими, чем BRADU и RHIEC. Что не соответствует существующей таксономии, поскольку и BRADU, и RHIEC в ней отнесены к одному порядку (Rhizobiales), а RHOS4 - к другому (Rhodobacterales). Задание 2Поиск гомологов АТФ-связывающего домена АТФ-зависимой Clp-протеазы E.Coli был выполнен с помощью обединения протеомов бактерий в один файл, создания на его основе базы для Blast ( makeblastdb -in All_proteoms.fasta -dbtype prot -out db.fasta) и последующего её использования для blastp (blastp -query clpx.fa -evalue 0.001 -db db.fasta -out results.txt). В результате было получено 24 гомолога.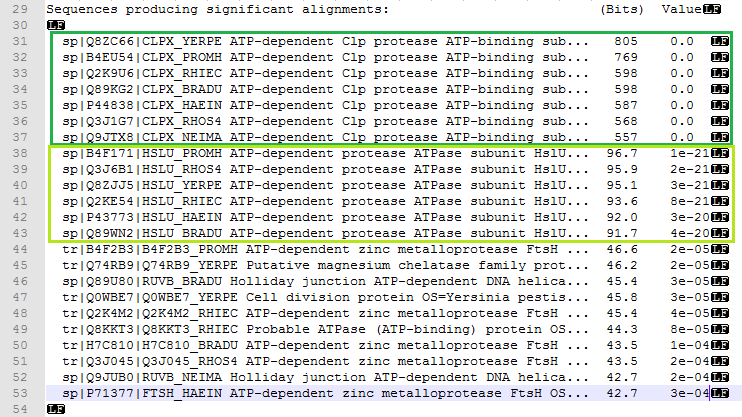 Все виды содержат ортологи clp-протеазы E.Coli, имея максимальное сходство с эталоном поиска: 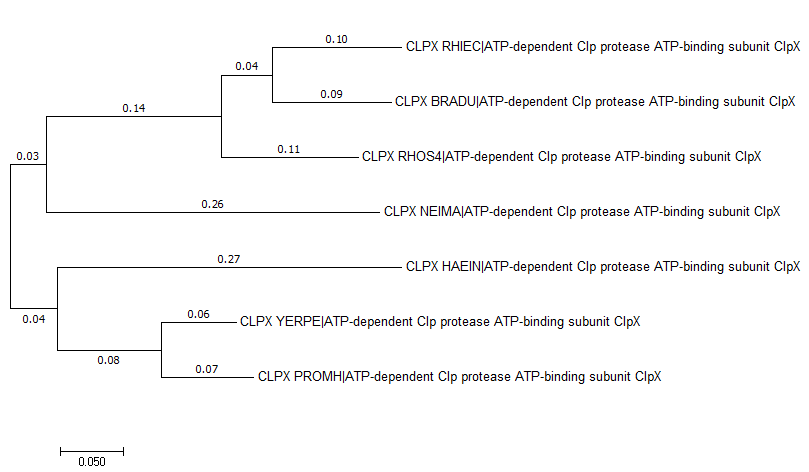 Также был выявлен паралог, АТФ-связывающий домен другой протеазы, обладающей меньшей степенью гомологии. 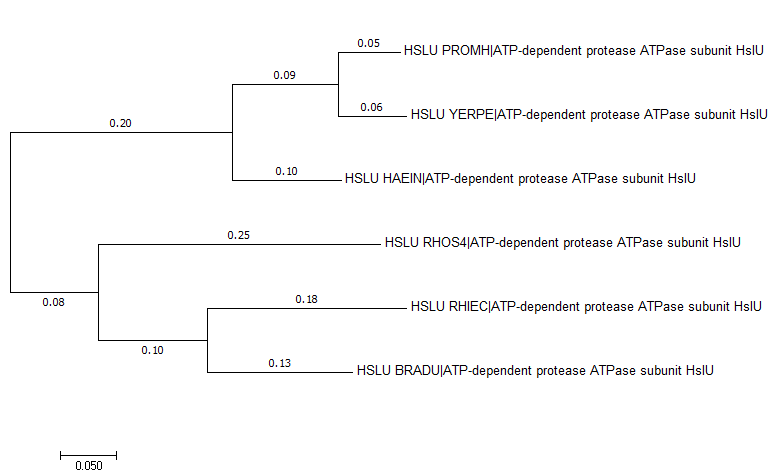 Дупликаций для каждого гена найдено не было, Neisseria Meningitidis не содержит HSLU-протеазы, и функции белков разделились скорее всего в результате дупликации и дальнейшей мутации: 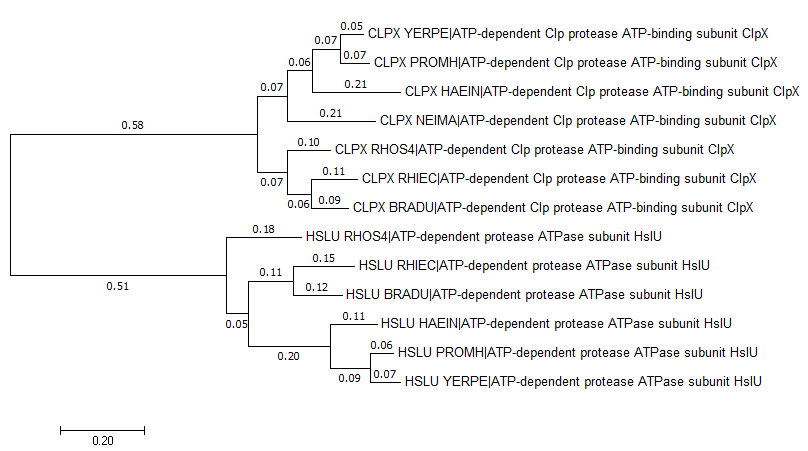 Остальные находки не имеют функционального сходства и сильно отличаются по качеству, поэтому в рассмотрение не входят. |