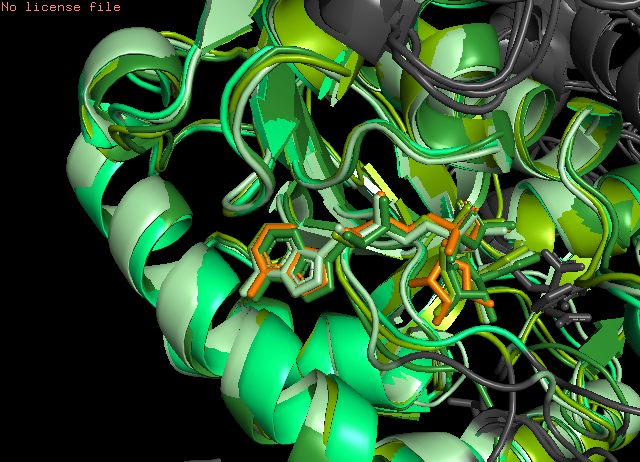
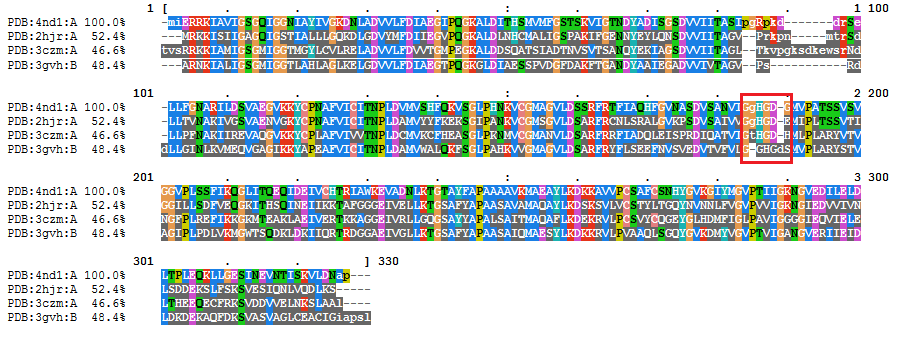
Структурное выравниваниеПоиск структурных гомологов и совмещение структурДля лактатдегидрогеназы (ЛДГ) Cyptosporidium parvum (PDB id: 4nd1, 4nd1.ccp4) в PDBeFold найдено 4 гомолога, удовлетворяющих условиям RMSD=[0.8, 2.5] Å и длина выровненного участка - не менее 50% от общей.
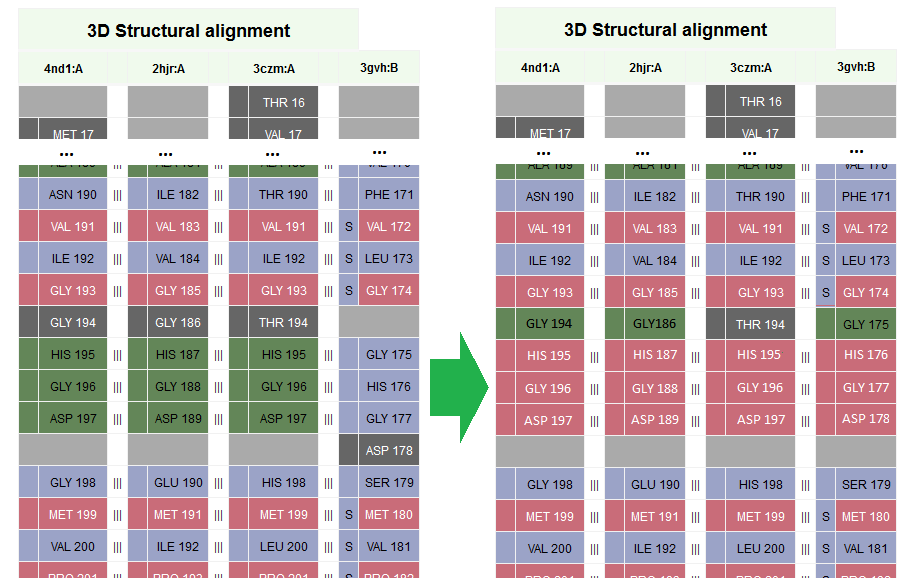
Выравнивание последовательностей, полученное при структурном выравнивании (в результатах выдачи PDBeFOLD по кнопке "download FASTA alignment"): al1.seq. Структурное выравнивание доступно в формате Rasmol send.rasmol и визуализировано в PyMol:
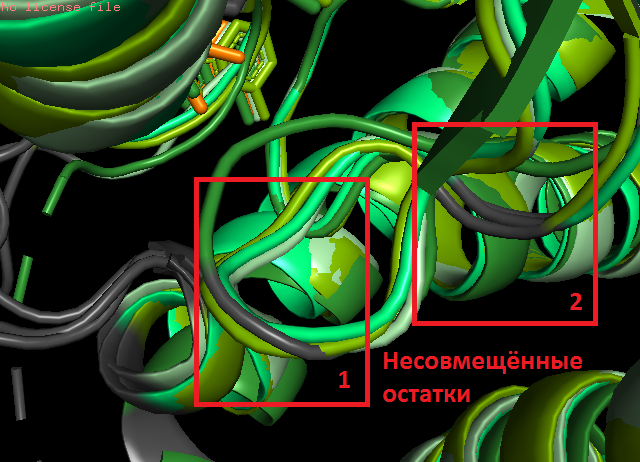
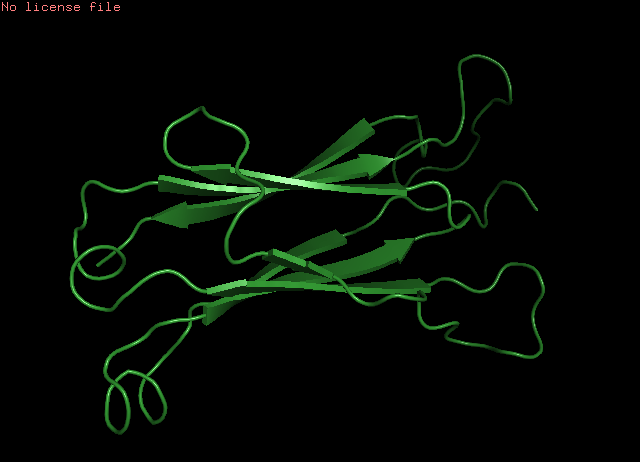
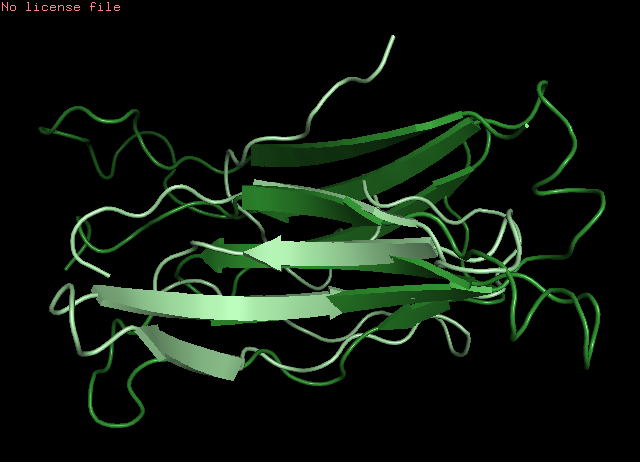
Цветами выделены участники выравнивания: palegreen - 4nd1:A, limegreen - 2hjr:A, splitpea - 3czm:B, forest - 3gvh:B  Рис. 1: Выровненный участок, видно расхождение хвостов цепей
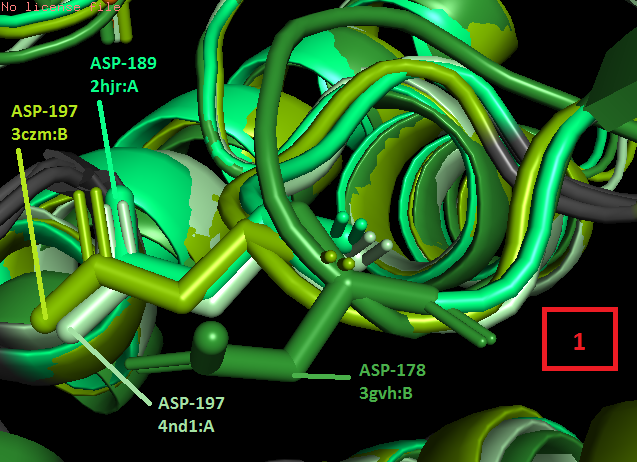
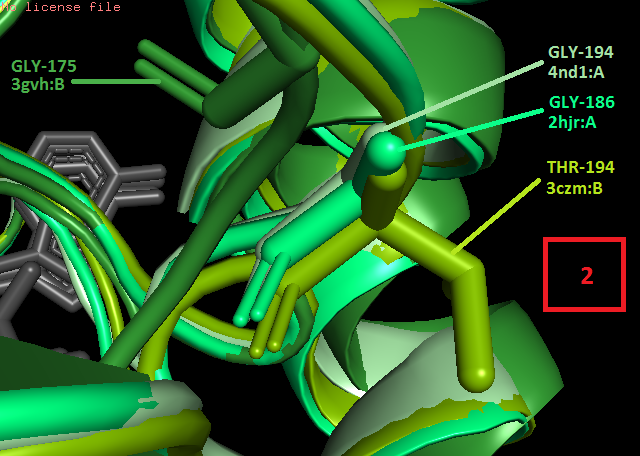
В качестве примера несовпадения можно привести следующий участок:
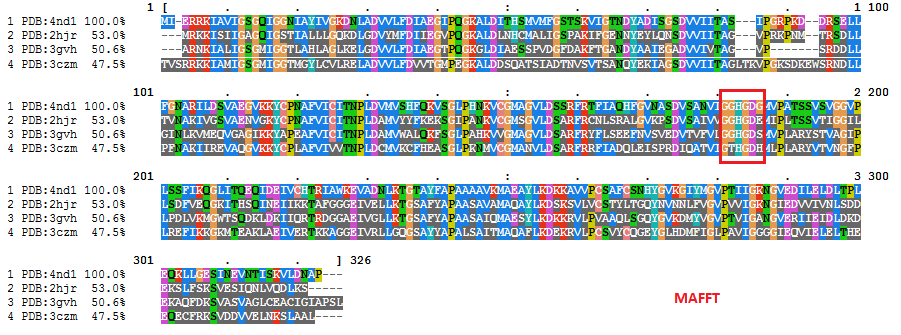
Выравнивание последовательностей с помощью MAFFT как раз и выдаёт представленный на рис. 6 вариант справа:
Совмещение по заданному выравниваниюДлля совмещения выбраны следующие константные домены субъединиц TCR (T-Cell receptor):
Домены извлечены в отдельные структурные файлы с помощью PyMol (select->extract->export molecule) (номера остатков, меж которыми содержится константный домен, взяты из SCOP) С помощью сервиса SheeP построены карты β-слоёв константных доменов:  Рис. 8: Карты β-слоёв, консервативные цистеины - в зелёной рамке
В единственном β-слое α-субъединицы и в первом β-слое β-субъединицы есть консервативный цистеин (Рис. 8, в зелёной рамке), по которому можно выполнить совмещение этих β-слоёв.
Использованные для совмещения команды: По выбранным остаткам совмещение прошло удачно с RMS = 0.533. Однако нижние тяжи β-слоёв сильно разошлись, и вряд ли сойдутся, если выбрать остатки для совмещения как-то иначе. Топология β-слоёв оказалась схожей (нумерация тяжей - сверху вниз по по Рис.9): хвост -> тяж 1, слева направо -> петля -> тяж 2, справа налево -> петля (в другой β-слой у β и просто петля у α-субъединицы) -> тяж 4, справа налево -> петля -> тяж 3, слева направо).
|