Сигналы и мотивы - 2
Задание 1.
В качестве объекта для выполнения поиска последовательности Шайна-Дальгарно была выбрана бактерия Kosakonia arachidis (из 1 семестра). Большинство бактерий рода Kosakonia являются симбионтами и комменсалами растений, также встречаются виды, условно патогенные для человека. Последовательность Шайна-Дальгарно находится на 6-8 нуклеотидов выше старт-кодона гена. Она представляет собой сайт связывания рибосом на молекуле мРНК, её консенсус - AGGAGG. На 3'-конце 16S рибосомной РНК находится комплементарная ей последовательность анти-Шайно-Дальгарно. Она нужна для связывания малой субъединицы рибосомы с последовательностью Шайна-Дальгарно на мРНК перед ATG. Там и собирается инициаторный комплекс для трансляции.
Для поиска соответствующего мотивы были upstream-последовательности длины 20 генов с обеих цепей ДНК (гены с -1 цепи были перевернуты и транскрибированы). Также были исключены гипотетические белки. Послная таблица генов из GenBank доступна по ссылке, отобранные из неё CDS - по другой ссылке. Файл с вырезанными upstream-последовательностями был создан с помощью следующего скрипта. Далее к ним была добавлена 3'-концевая часть последовательности 16S РНК (также перевернутая и транскирибированная). Файл был подан на вход программе МЕМЕ. Параметры программы настроены следующим образом: поиск 3 мотивов, встречающихся 0 или 1 раз в каждой последовательности (учитываем, что последовательность Шайно-Дальгарно может отсутствовать перед геном, но мы не ожидаем увидеть её в одной последовательности дважды), длина мотива от 6 до 10 нуклеотидов, поиск только по данной цепи. В результате получаем следующее:
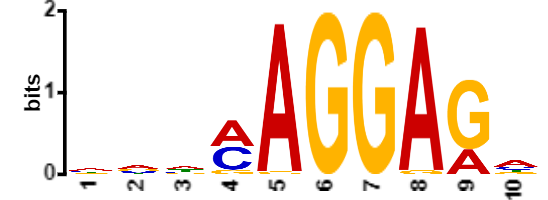
В целом, такой мотив соотвествует последовательности Шайна-Дальгарно. E-value для такого мотива равен 1.0e-018, информационное содержание - 10. Но из 4341 последовательностей мотив нашелся всего лишь в 495. Из-за большого количества поданных последовательностей выдача локализации мотива оказалась скрыта. Далее для полученного мотива была создана следующая позиционно-весовая матрица. Стоит сказать, что первоначально гипотетические белки не были исключены в ходе вырезания upstream-последовательностей. Соответствующая выдача программы MEME показала результат значительно хуже.
Задание 2.
Выдача программы МЕМЕ была перенаправлена в программу FIMO. В результате была получена таблица.
Для проверки матрицы с помощью FIMO, на вход программе подавался файл с геномом бактерии и позиционно весовая матрица, в результате было получено 888 находок. C помощью следующего скрипта было найдено число находок, попадающих в upstream-регион генов, равное 159. Таким образом, только 17,9% находок находятся в upstream-регионах генов, что говорит о низкой специфичности матрицы к последовательностям Шайна-Дальгарно.