JalView
Практикум №10
Практикум №10
1. Основные возможности JalView
| ID | Домен | Длина | Абсолютно консерв. позиции | Абсолютно функц. консервативыне позиции | Гэпы (без учета конц.) | 70% функционально консервативные позиции |
| DNAK_METBF | Bacteria | 620 | 179 | 269 | 43 | 334 |
| DNAK_THEVO | Bacteria | 613 | 179 | 269 | 47 | 284 |
| DNAK_STRCO | Archaea | 618 | 179 | 269 | 48 | 337 |
| DNAK_CORGB | Archaea | 618 | 179 | 269 | 51 | 339 |
| HSP7C_CHICK | Eukarya | 646 | 179 | 269 | 14 | 305 |
| HSP71_ORYLA | Eukarya | 639 | 179 | 269 | 23 | 307 |
Комментарий к таблице:
Остатки являются функционально идентичными, если при замене одного на другой в последовательности белка не происходит изменения свойств белка. Позиция является функционально консервативной, если ее аминокислотный остаток не менялся или менялся только на функционально идентичный. В частности, это замены в группах (D, E); (G, A, V, L, I) и т.п.
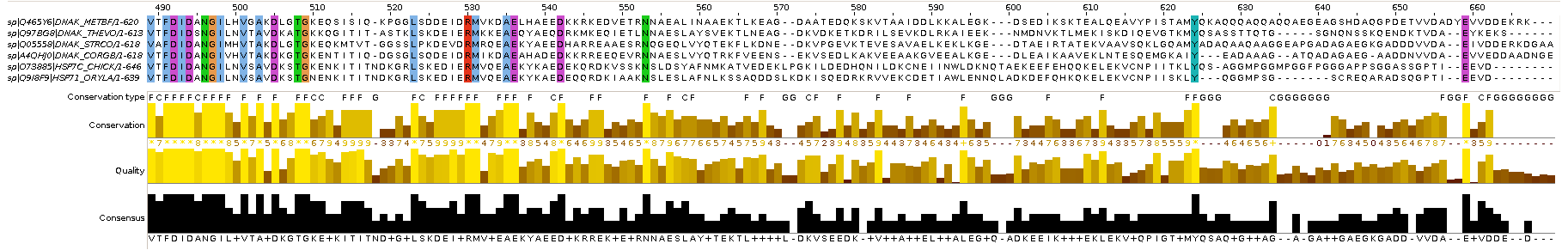 Рисунок 1. Выравнивание последовательностей в JalView
Рисунок 1. Выравнивание последовательностей в JalView
Позиции выравнивания: G - содержащие гэп; F - полностью функционально консервативные; С - консервативные на 80+%.
Описание конкретных позиций:
F - Полностью функционально консервативные позиции выравнивания, например:
491: F;F;F;F;F;F
575: A;V;V;V;I;I – аминокислоты (G, A, V, L, I) близки по свойствам
624: Y;Y;Y;Y;Y;Y
C - Позиции выравнивания, консервативные на 80+%, например:
495: A;S;A;A;A;A
510: K;K;K;K;K;N
541: E;Q;E;E;E;E
G - Позиции выравнивания, содержащие гэп, например:
518: -;-;-;-;N;N
571: -;-;-;-;L;L
625: Q;Q;A;-;Q;-
2. Эволюция последовательности белка
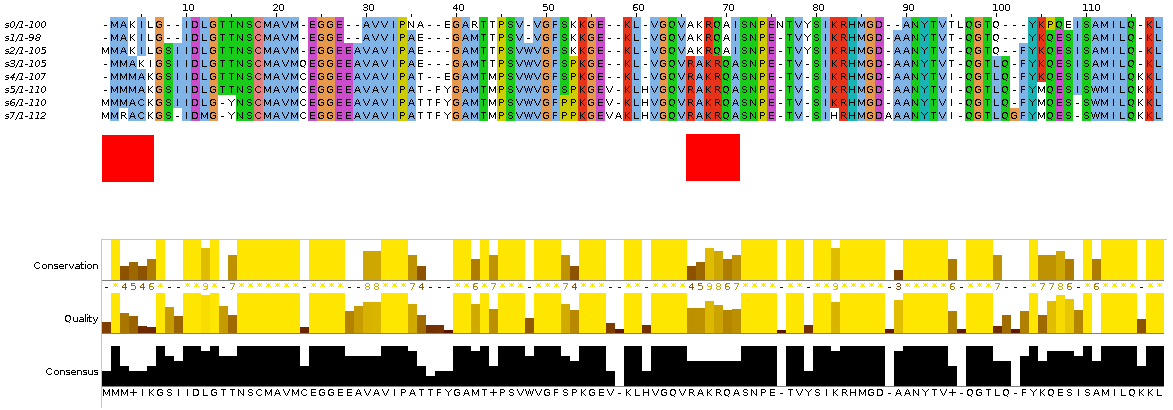 Рисунок 2. Автоматическое выравнивание в JalView (MUSCLE)
Рисунок 2. Автоматическое выравнивание в JalView (MUSCLE)
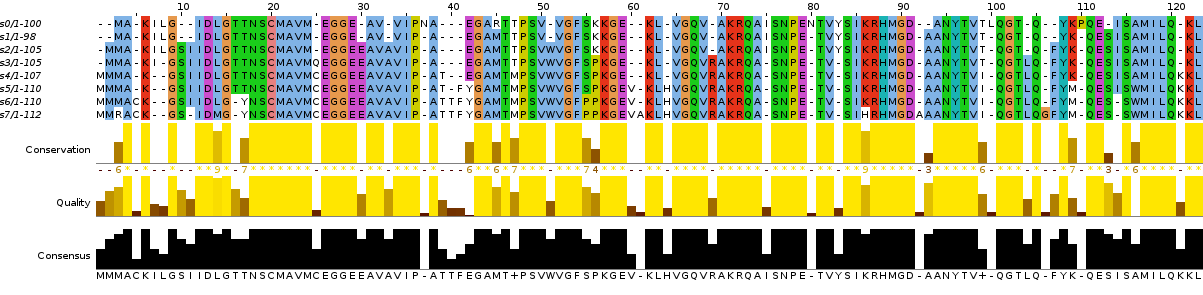 Рисунок 3. Исправленное выравнивание
Рисунок 3. Исправленное выравнивание
Комментарий:
Автоматическое выравнивание, конечно же, содержит ошибки, в том числе из-за того, что программа воспринимает последовательности как эволюционировавшие не последовательно, а параллельно.
Наиболее важные ошибки отмечены красными полями на рис.2.
| № | Позиция выравнивания | [Было] | [Стало] | Изменилось в... |
| 1 | 7 | I | – | s4 |
| 2 | 17 | T | Y | s6 |
| 3 | 45 | R | M | s1 |
| 4 | 51 | – | W | s2 |
| 5 | 86 | K | H | s7 |
| 6 | 93 | – | A | s1 |
| 7 | 99 | T | I | s4 |
| 8 | 104 | – | L | s3 |
| 9 | 109 | K | M | s5 |
| 10 | 110 | P | – | s1 |
Загрузить скрипт, моделирующий «небывалую эволюцию последовательности белка» для <X> поколений.
Синтаксис: ./script.sh <X> <Input sequence file> <Output file>.
© Arsenii Loginovskii, 2016-2017
