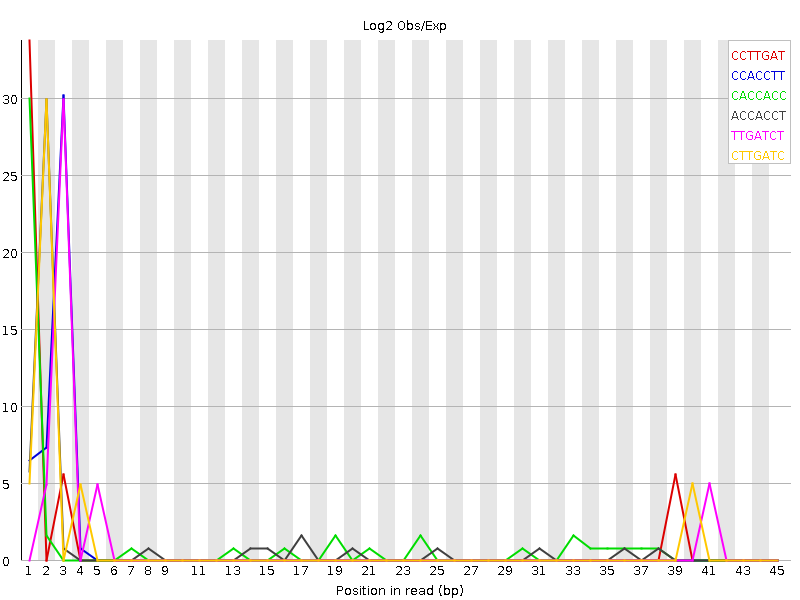
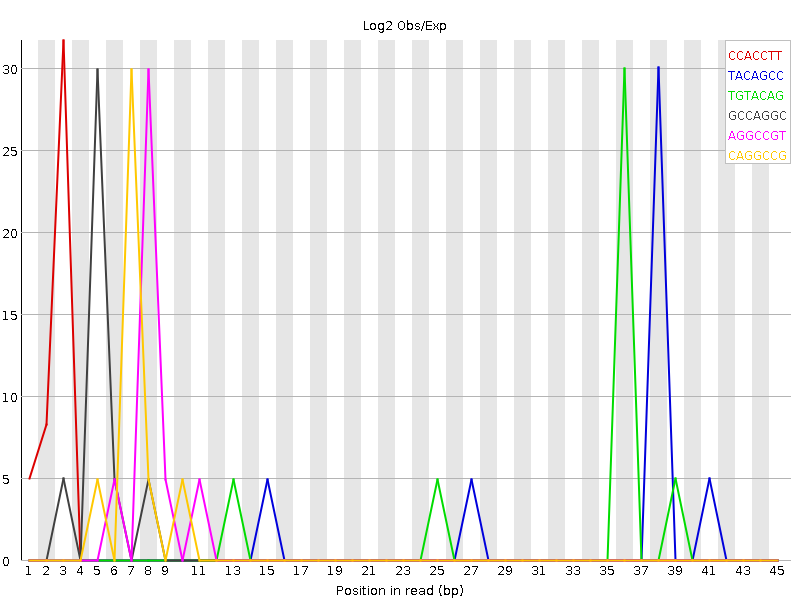
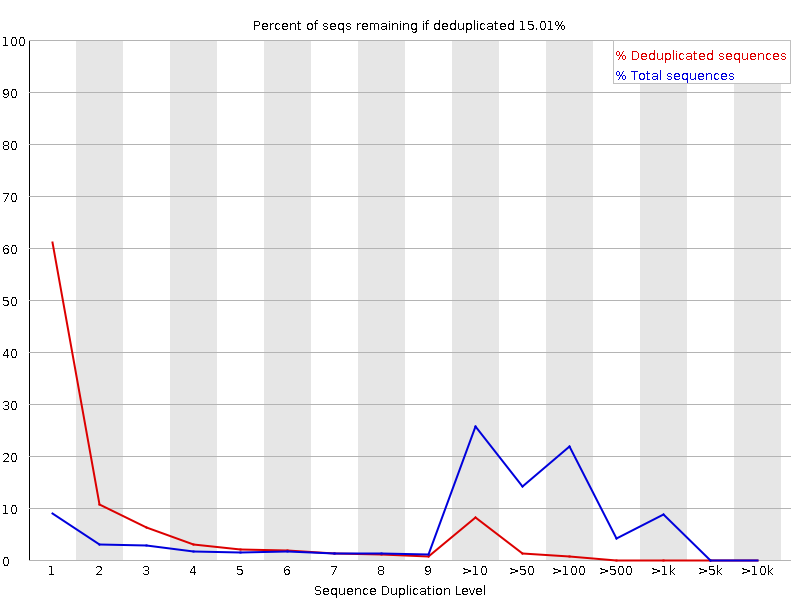
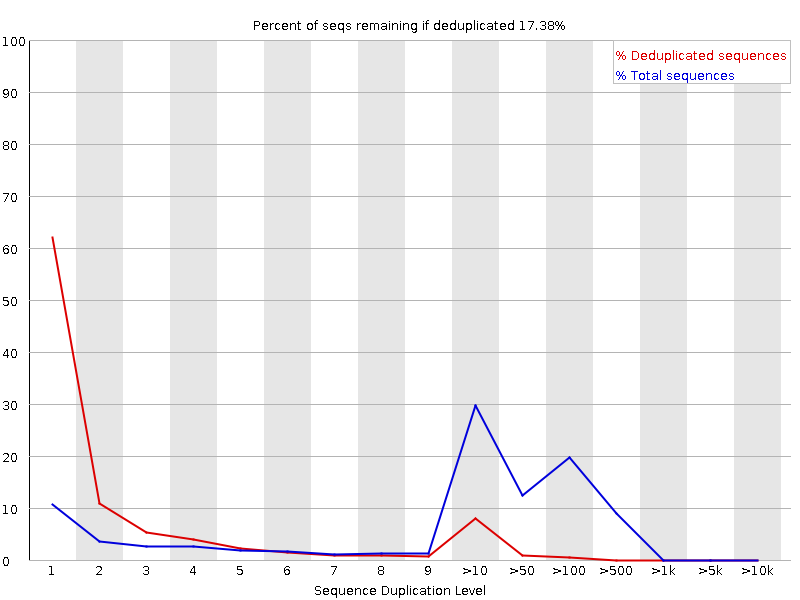
Остальная выдача FastQC имеет незначительные различия; упоминания стоит K-мерное распределение и анализ дубликатов последовательностей:
Из этой выдачи видно, что в репликах встречается несколько отдельных сильно экспрессированных последовательностей – на это указывают отдельные острые пики на графиках.
Видно, что в ридах присутствует очень много повторяющихся последовательностей (особенно в реплике 1) Это весьма характерно для RNA-Seq: для того, чтобы было возможно видеть слабоэкспрессированные последовательности, приходится многократно прочитывать хорошо экспрессированные участки, что создает достаточно большое количество дубликатов чтений.