Реконструкция эволюции доменной архитектуры
Практикум №7
Данные
Для работы был выбран домен MAGE (ID PF01454). Функция генов с этим доменом неясна, они оказываются экспрессированы в различных типах опухолей.
Список доменных архитектур с этим доменом.
Из них были выбраны две доменные архитектуры:
1. Содержит единственный домен MAGE (1072 последовательности).
2. Содержит 2 домена: MAGE_N и MAGE (514 последовательностей).
Для дальнейшего анализа был выбран таксон Mammalia. Было рассмотрено три его подтаксона: Proboscidea (P), Glires (G) и Primates (A).
Результаты работы:
Таблица Excel с описанием белков из Uniprot,
Выравнивание отобранных вами последовательностей домена (проект JalView)
Из выравнивания были удалены N- и C-концевые участки до блоков с удовлетворительным качеством выравнивания. Кроме того, из средней части выравнивания были удалены богатые гэпами блоки.
Филогенетическое дерево последовательностей домена:
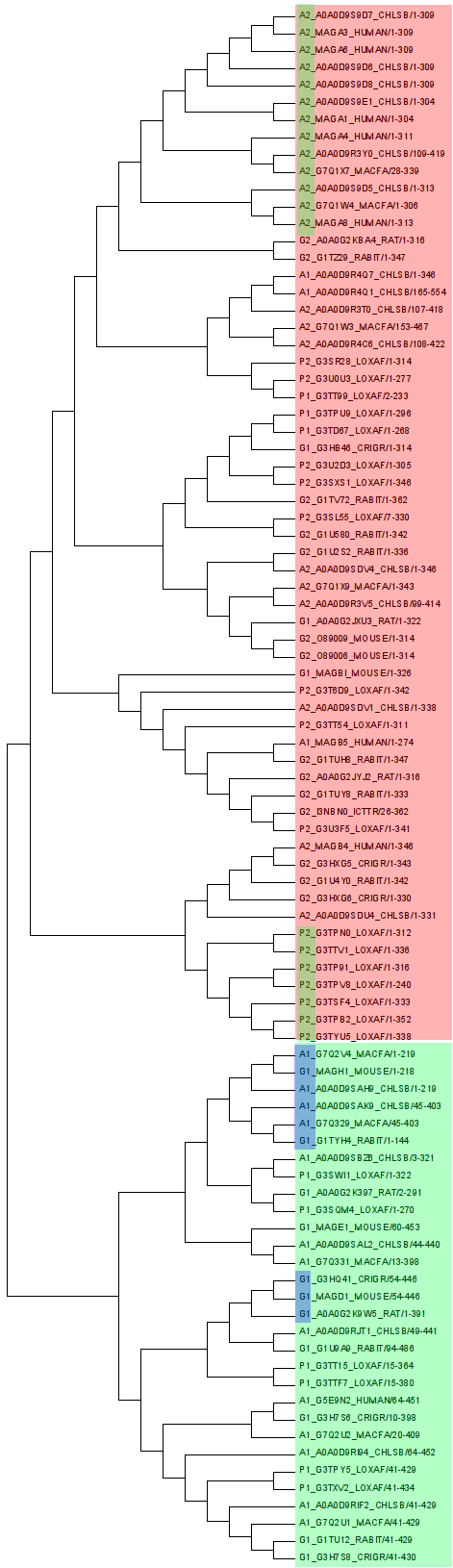
Рисунок 1. Филогенетическое дерево последовательностей домена.
Дерево было построено с помощью метода объединения соседей (Neighbor joining).
(((((((((((((('A2_A0A0D9S9D7_CHLSB/1-309':0.09588128,'A2_MAGA3_HUMAN/1-309':0.02190176):0.04150351,'A2_MAGA6_HUMAN/1-309':0.08415371):0.01194660,'A2_A0A0D9S9D6_CHLSB/1-309':0.00335657):0.03772003,'A2_A0A0D9S9D8_CHLSB/1-309':0.02251334):0.17281626,('A2_A0A0D9S9E1_CHLSB/1-304':0.01824887,'A2_MAGA1_HUMAN/1-304':0.01949146):0.09427110):0.06336867,('A2_MAGA4_HUMAN/1-311':0.01334382,('A2_A0A0D9R3Y0_CHLSB/109-419':0.02300800,'A2_G7Q1X7_MACFA/28-339':0.01473233):0.04474706):0.02361751):0.01707278,('A2_A0A0D9S9D5_CHLSB/1-313':0.10128911,('A2_G7Q1W4_MACFA/1-306':0.00000000,'A2_MAGA8_HUMAN/1-313':0.00000000):0.01649392):0.06875492):0.09605386,('G2_A0A0G2KBA4_RAT/1-316':0.29431991,'G2_G1TZ29_RABIT/1-347':0.16830361):0.02748917):0.02072628,((('P1_G3TT99_LOXAF/2-233':0.20283915,'P2_G3U0U3_LOXAF/1-277':0.25978437):0.02282087,'P2_G3SR28_LOXAF/1-314':0.15133248):0.01135304,(('A2_A0A0D9R4C6_CHLSB/108-422':0.00000000,'A2_G7Q1W3_MACFA/153-467':0.00000000):0.09099955,('A2_A0A0D9R3T0_CHLSB/107-418':0.26157538,('A1_A0A0D9R4Q1_CHLSB/165-554':0.15213473,'A1_A0A0D9R4Q7_CHLSB/1-346':0.14796987):0.21065542):0.03518191):0.05329921):0.03693091):0.02740703,((((('G2_O89006_MOUSE/1-314':0.06026581,'G2_O89009_MOUSE/1-314':0.01669523):0.29866655,'G1_A0A0G2JXU3_RAT/1-322':0.21837033):0.04010132,('A2_A0A0D9R3V5_CHLSB/99-414':0.00000000,'A2_G7Q1X9_MACFA/1-343':0.00000000):0.19121044):0.04585409,('A2_A0A0D9SDV4_CHLSB/1-346':0.10266007,'G2_G1U2S2_RABIT/1-336':0.10213434):0.06484875):0.04149113,(('G2_G1U580_RABIT/1-342':0.03973945,'P2_G3SL55_LOXAF/7-330':0.16505496):0.11510816,('G2_G1TV72_RABIT/1-362':0.15164538,(('P2_G3SXS1_LOXAF/1-346':0.04011303,'P2_G3U2D3_LOXAF/1-305':-0.00237270):0.14559732,('G1_G3HB46_CRIGR/1-314':0.29042693,('P1_G3TD67_LOXAF/1-268':0.03920671,'P1_G3TPU9_LOXAF/1-296':0.07857633):0.08472586):0.06809723):0.06475031):0.03172544):0.01374365):0.00861775):0.02724599,('G1_MAGBI_MOUSE/1-326':0.24262292,('P2_G3T6D9_LOXAF/1-342':0.23018413,('A2_A0A0D9SDV1_CHLSB/1-338':0.17396503,('P2_G3TT54_LOXAF/1-311':0.23188108,(('A1_MAGB5_HUMAN/1-274':0.37131988,'G2_G1TUH8_RABIT/1-347':0.09130364):0.07123394,('G2_A0A0G2JYJ2_RAT/1-316':0.17352097,('G2_G1TUY8_RABIT/1-333':0.23468185,('G2_I3NBN0_ICTTR/26-362':0.01616306,'P2_G3U3F5_LOXAF/1-341':0.14417959):0.04211580):0.06998833):0.03312592):0.01252981):0.02474728):0.02491530):0.01809812):0.05005212):0.04326771,((((('A2_MAGB4_HUMAN/1-346':0.10795433,'G2_G3HXG5_CRIGR/1-343':0.14336010):0.01819137,'G2_G1U4Y0_RABIT/1-342':0.06197996):0.05651427,'G2_G3HXG6_CRIGR/1-330':0.33744439):0.05980881,'A2_A0A0D9SDU4_CHLSB/1-331':0.31277975):0.01060913,(('P2_G3TPN0_LOXAF/1-312':0.14855796,'P2_G3TTV1_LOXAF/1-336':0.10275647):0.05942983,(('P2_G3TP91_LOXAF/1-316':0.03564127,'P2_G3TPV8_LOXAF/1-240':0.04131977):0.04263256,('P2_G3TSF4_LOXAF/1-333':0.04133131,('P2_G3TPB2_LOXAF/1-352':0.08551158,'P2_G3TYU5_LOXAF/1-338':0.03227146):0.03884002):0.05466697):0.05382759):0.04959619):0.10363677):0.13937630,((('A1_A0A0D9SAL2_CHLSB/44-440':0.00000000,'A1_G7Q331_MACFA/13-398':0.00000000):0.03090680,'G1_MAGE1_MOUSE/60-453':0.08687623):0.28689705,((('A1_A0A0D9SBZ6_CHLSB/3-321':0.03134527,'P1_G3SWI1_LOXAF/1-322':0.00639505):0.34045272,('G1_A0A0G2K397_RAT/2-291':0.13093060,'P1_G3SQM4_LOXAF/1-270':0.16917399):0.08400950):0.02620315,((('A1_G7Q2V4_MACFA/1-219':0.00000000,'G1_MAGH1_MOUSE/1-218':0.00000000):0.00000000,'A1_A0A0D9SAH9_CHLSB/1-219':0.00000000):0.31462706,('A1_A0A0D9SAK9_CHLSB/45-403':-0.00018861,('A1_G7Q329_MACFA/45-403':-0.00037294,'G1_G1TYH4_RABIT/1-144':0.03811327):0.00018861):0.59770700):0.04548510):0.06588461):0.02679474):0.03244016,((((('G1_G3HQ41_CRIGR/54-446':0.00000000,'G1_MAGD1_MOUSE/54-446':0.00000000):0.00000000,'G1_A0A0G2K9W5_RAT/1-391':0.00000000):0.02761633,('A1_A0A0D9RJT1_CHLSB/49-441':0.00000000,'G1_G1U9A9_RABIT/94-486':0.00000000):0.01012400):0.06661108,('P1_G3TT15_LOXAF/15-364':0.47477422,'P1_G3TTF7_LOXAF/15-380':0.18200532):0.12638405):0.08186973,((('A1_G5E9N2_HUMAN/64-451':0.00000000,'G1_G3H7S6_CRIGR/10-398':0.00000000):0.01967136,'A1_G7Q2U2_MACFA/20-409':0.01806897):0.02514504,('A1_A0A0D9RI94_CHLSB/64-452':0.02164346,(('P1_G3TPY5_LOXAF/41-429':0.00000000,'P1_G3TXV2_LOXAF/41-434':0.00000000):0.01657272,('A1_A0A0D9RIF2_CHLSB/41-429':0.00000000,('A1_G7Q2U1_MACFA/41-429':0.00000000,('G1_G1TU12_RABIT/41-429':0.00000000,'G1_G3H7S8_CRIGR/41-430':0.00000000):0.00000000):0.00000000):0.02116761):0.01683706):0.05640332):0.06038846):0.14379845);
Как достаточно хорошо видно по этому дереву, перестройка доменной архитектуры происходила ровно один раз. Как выяснилось, разные таксоны группируются вместе. Вспоминая значение этого домена в организме, можно предположить, что его консервативность достаточно низка. Из-за этого страдает качество филогенетической реконструкции.
© Arsenii Loginovskii, 2016-2018
