Построение и анализ поверхностей в PyMOL
Практикум №9
Выбор структуры для анализа
Для выполнения анализа требуется найти структуру биомолекулы, полученную методом ЯМР, содержащую как минимум 2 цепи и 10 моделей. Для этого я провел расширенный поиск по базе данных PDB.
Запрос:
number of chains (biol. assembly):
from 2
number of models:
from 10
Для анализа я выбрал белок устойчивости к перекиси водорода BupsA из Burkholderia pseudomallei с PDB ID 6UHW. PDB-файл содержит 20 моделей.
Построение поверхности белка
С помощью PyMol была построена поверхность белка BupsA.
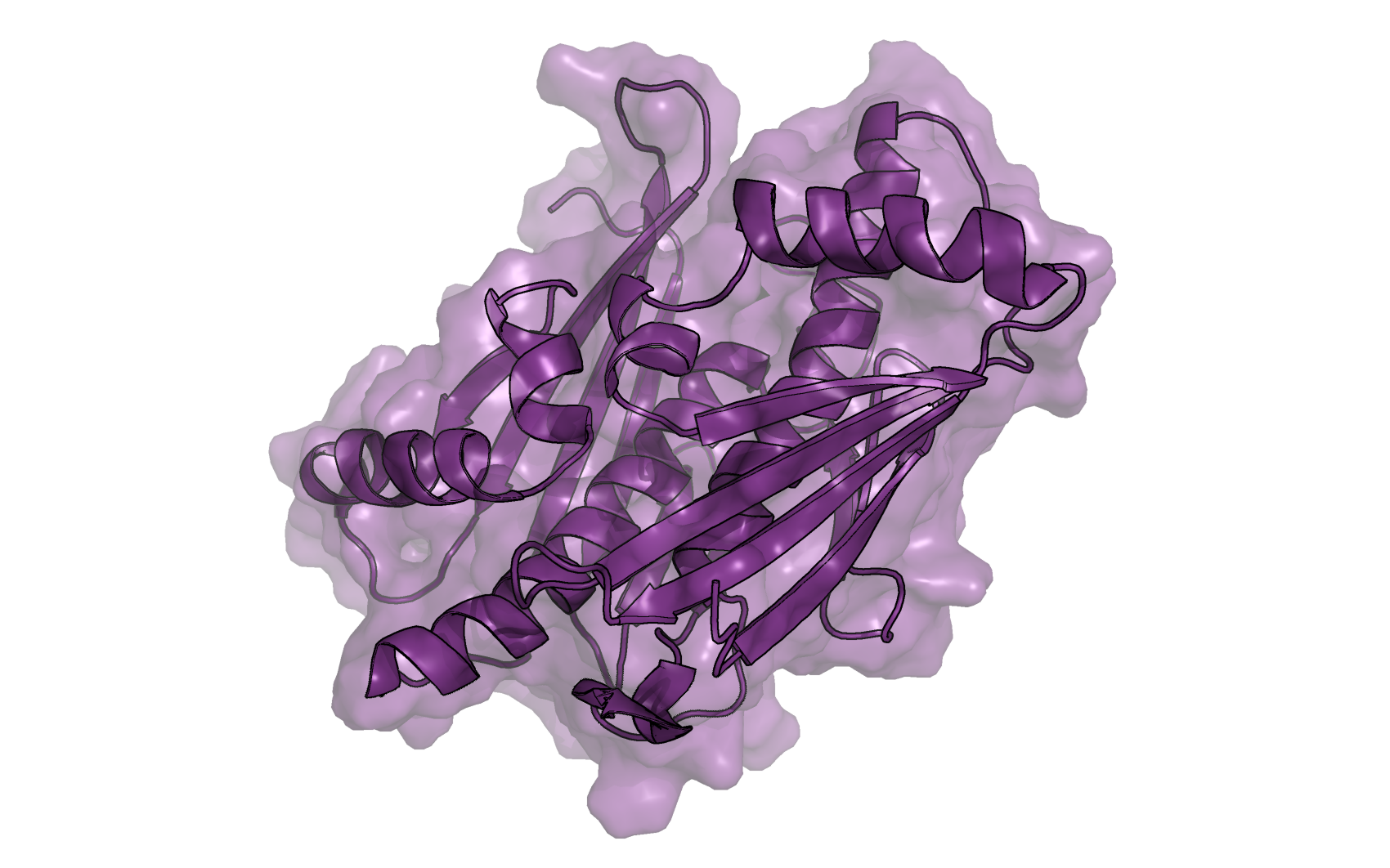 Рисунок 1. Общий вид и молекулярная поверхность белка BupsA (PDB ID 6UHW).
Рисунок 1. Общий вид и молекулярная поверхность белка BupsA (PDB ID 6UHW).
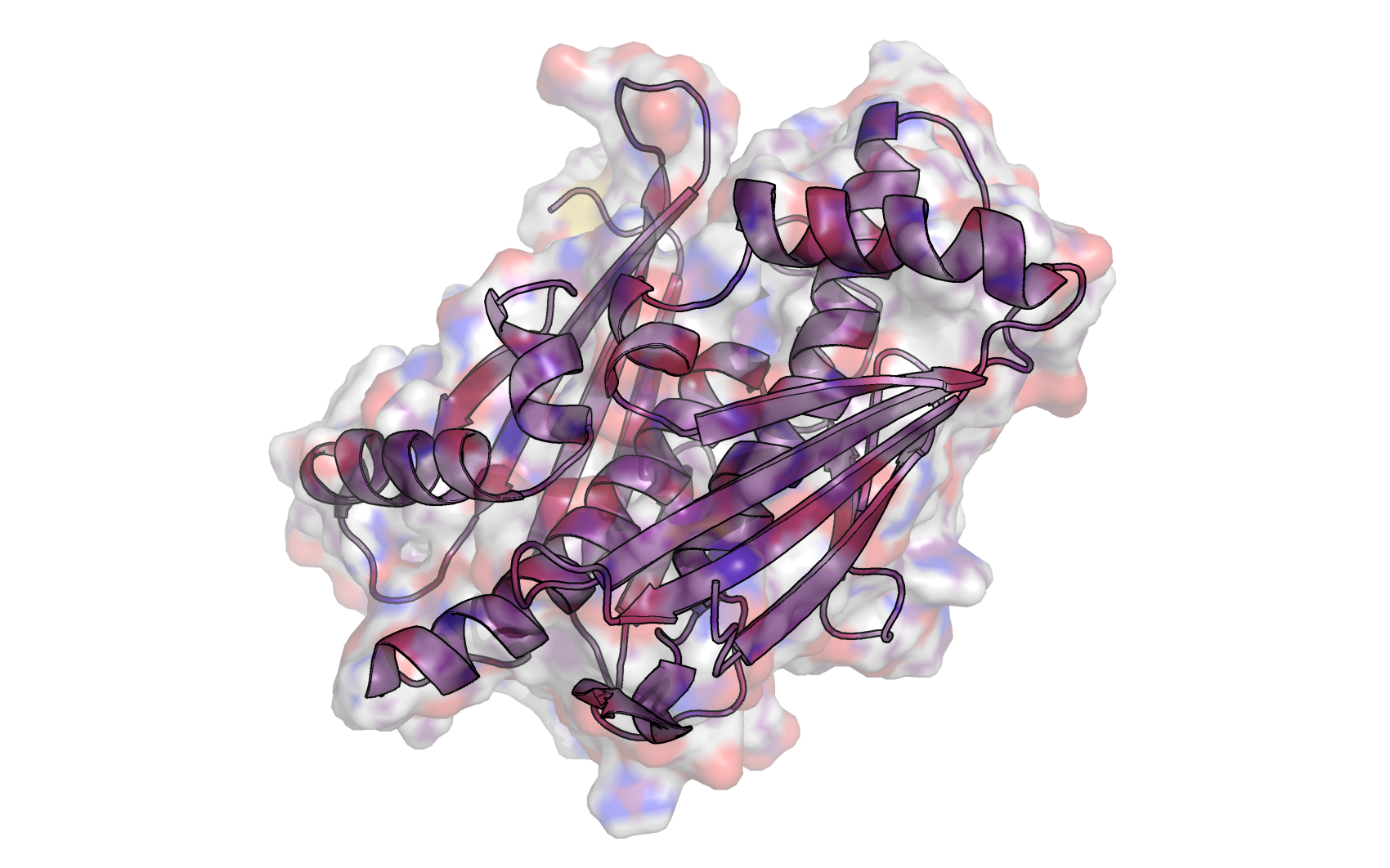 Рисунок 2. Общий вид и молекулярная поверхность белка BupsA (PDB ID 6UHW). Типы атомов выделены цветами (белый - водород, красный - кислород, синий - азот, желтый - сера)
Рисунок 2. Общий вид и молекулярная поверхность белка BupsA (PDB ID 6UHW). Типы атомов выделены цветами (белый - водород, красный - кислород, синий - азот, желтый - сера)
Подсчет зависимости площади молекулярной поверхности от площади SAS
Для каждой модели было вычислено значение площади молекулярной поверхности и доступной для растворителя поверхности (SAS). Для них было построено три графика:
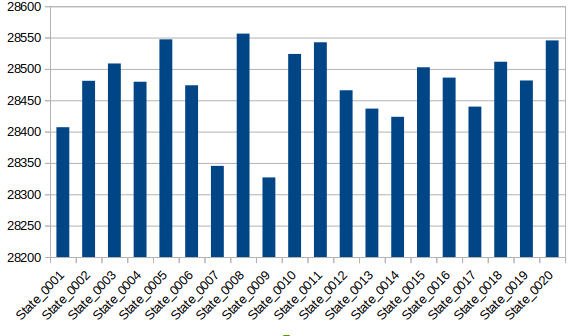
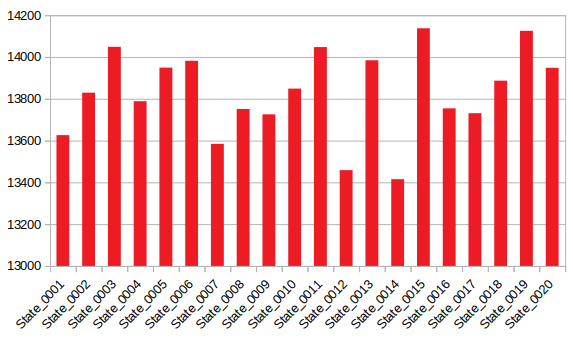 Рисунок 3, 4. Графики площадей поверхности для каждой модели. Слева данные по молекулярной поверхности, справа - по поверхности, доступной для растворителя (SAS).
Рисунок 3, 4. Графики площадей поверхности для каждой модели. Слева данные по молекулярной поверхности, справа - по поверхности, доступной для растворителя (SAS).
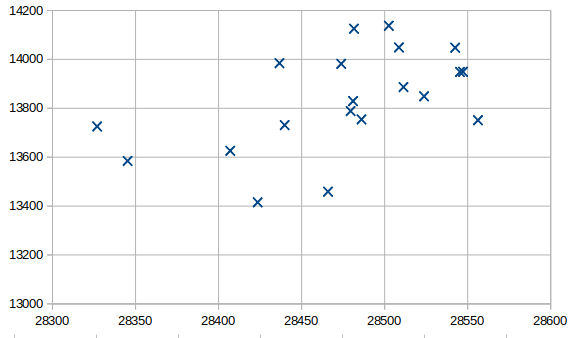
Рисунок 5. График распределения SAS относительно молекулярной поверхности. Каждая точка соответствует одной модели.
Как мы видим, площади общей молекулярной поверхности и поверхности, доступной для растворителя, коррелируют не очень хорошо. Возможно, это связано с наличием более "открытой" и, наоборот, "закрытой" конформаций белка.
Выделение межцепочечных контактов
С помощью программы PyMol были выделены остатки каждой цепи, находящиеся на расстоянии не более 3.5Å от другой цепи. Для наборов этих остатков в каждой цепи были построены поверхности, раскрашенные разными цветами в зависимости от цепи.
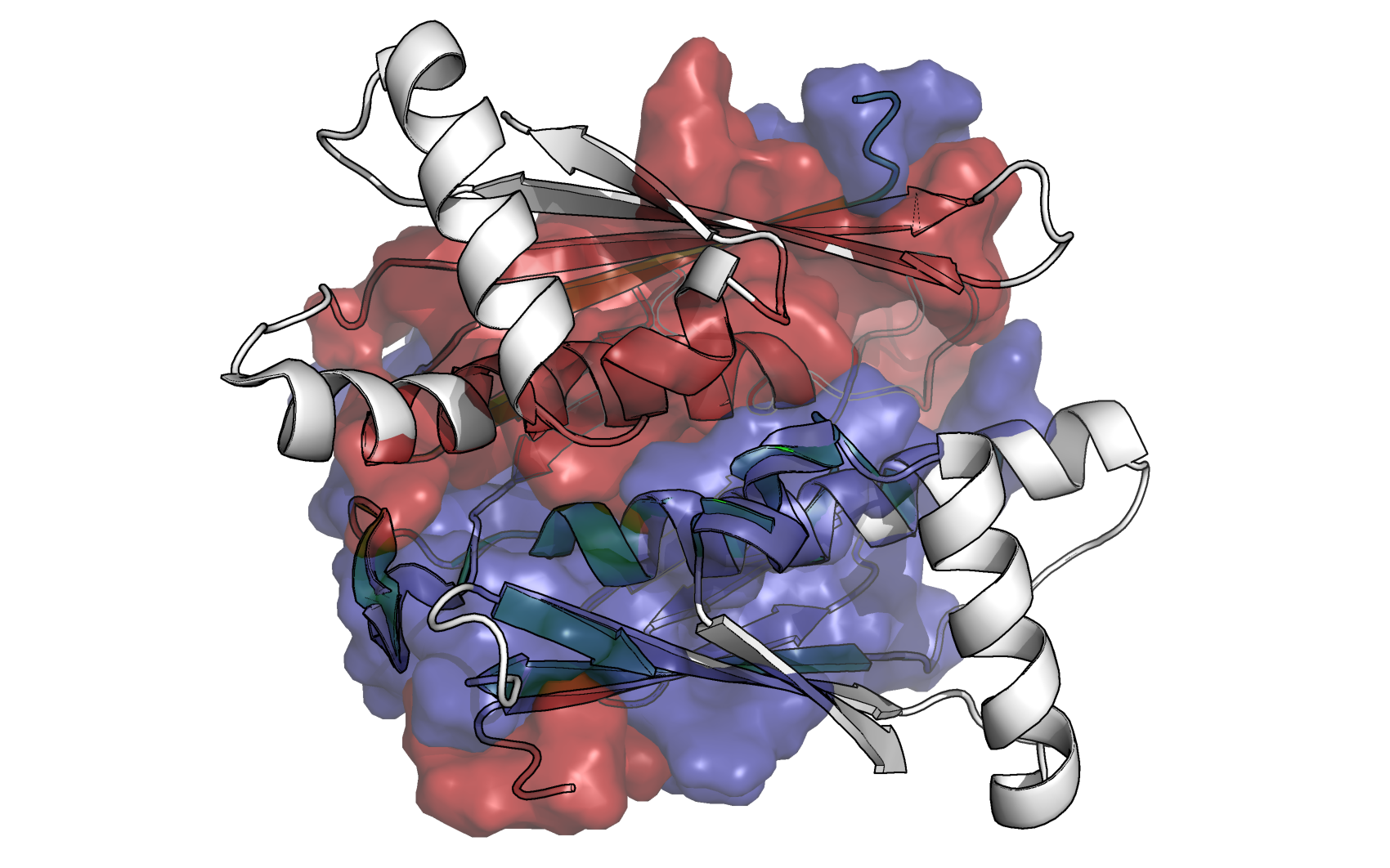 Рисунок 6. Поверхности межцепочечных контактов на схематичном виде структуры белка.
Рисунок 6. Поверхности межцепочечных контактов на схематичном виде структуры белка.
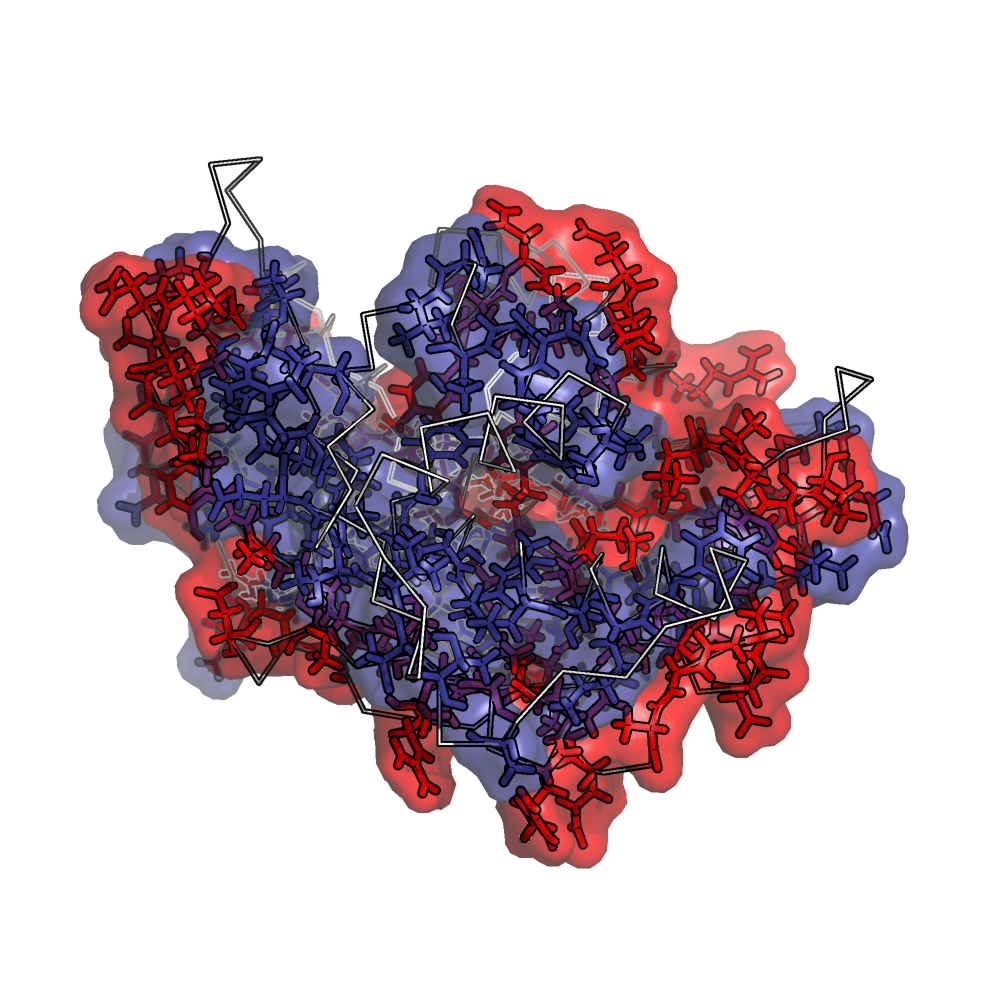
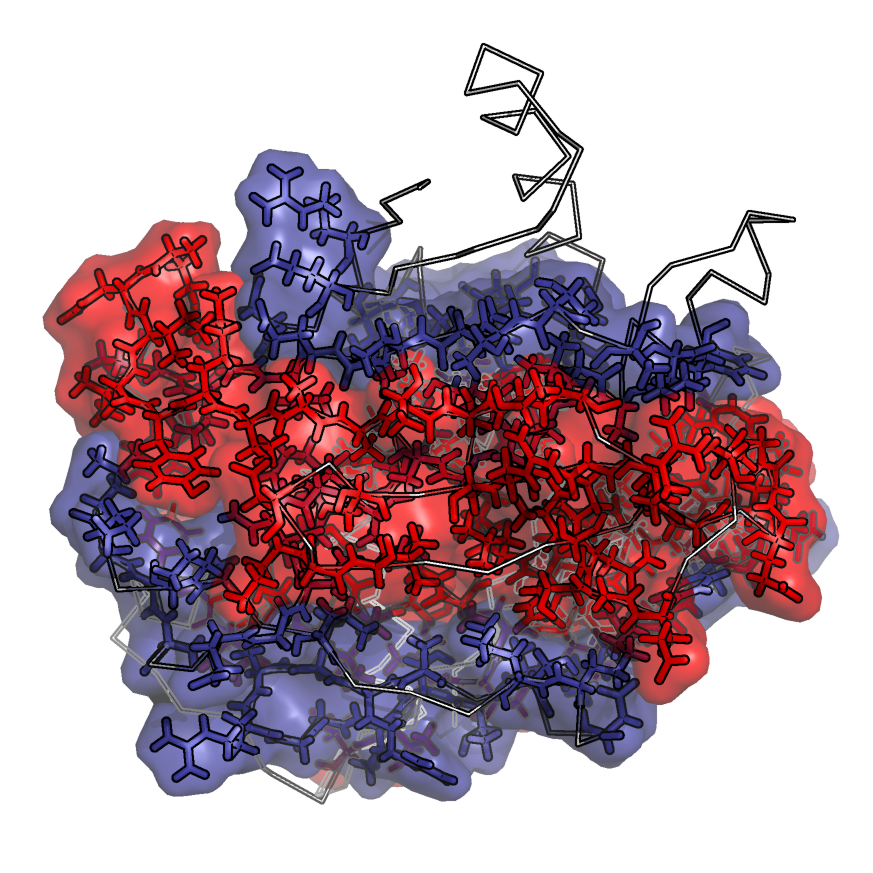 Рисунок 7, 8. Изображения поверхностей межцепочечных контактов с разных сторон.
Рисунок 7, 8. Изображения поверхностей межцепочечных контактов с разных сторон.
По полученным изображением видно, что каждая цепь белка тесно связана и почти целиком контактирует с другой цепью. Поверхности межцепочечных контактов неоднократно соприкасаются и фактически соответствуют друг другу.
© Arsenii Loginovskii, 2016-2020
