Практикум 12. Трансмембранные белки.
В этом практикуме я работаю с выданным мне трансмембранным белком TVP38/TMEM64 family membrane protein MT1538 (это его полное рекомендуемое название) с идентификатором Uniprot Y1491_MYCTO (далее будем называть его по идентификатору), он является альфа-спиральным по структуре, и с самостоятельно мною выбранным белком Мальтопорином кишечной палочки (E. coli) (выбран по критерию, что в его трансмембранной части содержатся бетта-листы).
Задание 1. Знакомство с базой данных OPM
Из этой базы данных я получил информацию о белке Мальтопорине, представленную в таблице 1.
Название белка |
Maltoporin (Мальтопорин) |
Идентификатор PDB |
1AF6 |
Идентификатор Uniprot |
LAMB_ECOLI |
Толщина гидрофобной части белка в мембране |
25.1 Å |
Координаты трансмембранных участков (номер участка(координаты)) |
1(2-13),2(39-48),3(58-68),4(75-88),5(98-103),6(125-132),7(138-146),8(170-179),9(185-194),10(213-221),11(227-235),12(269-278),13(284-293),14(305-314),15(320-329),16(343-352),17(361-370),18(411-420) |
Среднее количество остатков в одном β-тяже белка |
10 |
В какой мембране находится белок |
Внешняя мембрана Грамотрицательных бактерий |
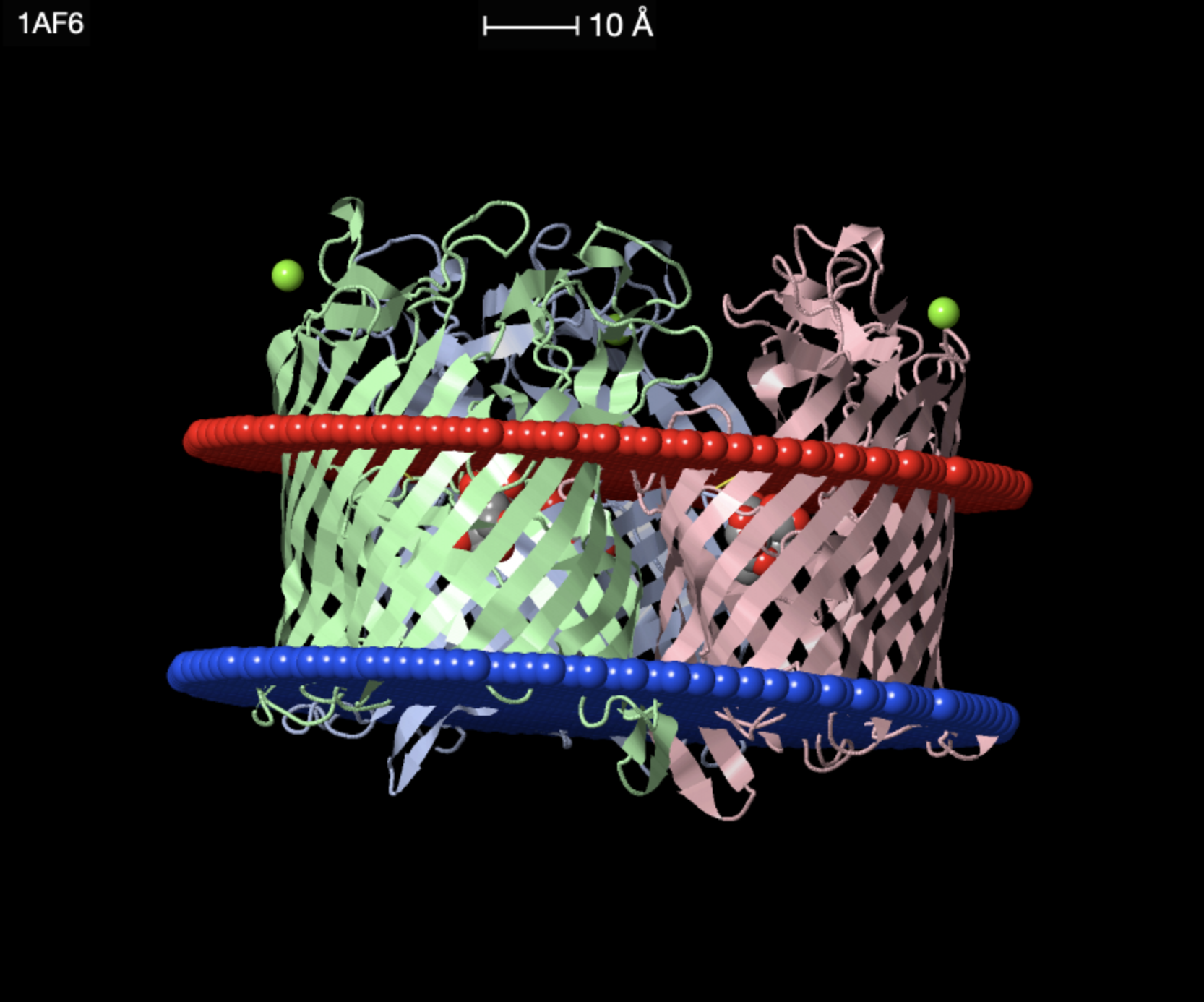
Ниже представлена визуализация данного белка с участком внешней мембраны грамотрицательной бактерии. Красный слой атомов обозначает внешнюю границу мембраны, снаружи которой внеклеточное пространство, синий слой обозначает внутреннюю сторону внешней мембраны, ограничивающую периплазматическое пространство. Белок является гомотримером, три идентичных субъединицы образуют функциональный комплекс. Субъединицы обозначены тремя разными пастельными цветами.
Задание 2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
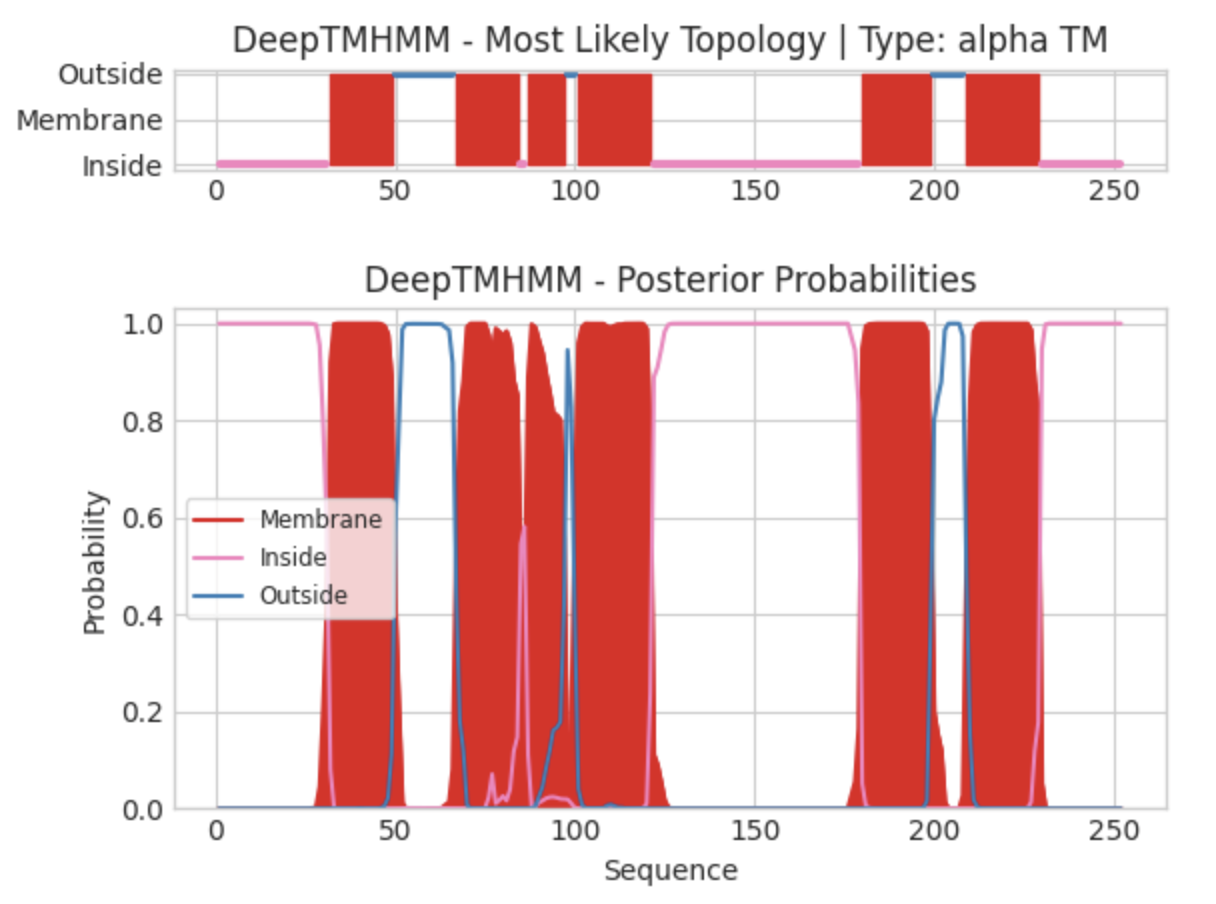
Результат применения алгоритма DeepTMHMM к последовательности белка Y1491_MYCTO доступен в текстовом формате по ссылке. Fasta-файл аминокислотной последовательности белка - по ссылке. На рисунке 2 изображено визуальное отображение предсказания, сделанного алгоритмом.
На горизонтальных осях показан номер аминокислотного остатка, по вертикали на верхнем графике показано отношение участка последовательности к тому или иному положению относительно мембраны (Membrane - трансмембранный фрагмент, Outside - внеклеточный, Inside - внутриклеточный). На нижнем графике показаны вероятности отнесения конкретного остатка к определенной топологии. Различные цвета аналогично указывают на разные местоположения: вне клетки, трансмембранный, внутри клетки В случае моего белка было предсказано наличие 6 трансмембранных альфа-спиралей. Согласно предсказанию, оба конца белка находятся внутри клетки.
Аналогичные действия я провел с бетта-листовым белком Мальтопорином, выбранным в задании 1. Fasta-файл аминокислотной последовательности белка - по ссылке. Результаты приведены на рисунке 3 и в текстовом файле.
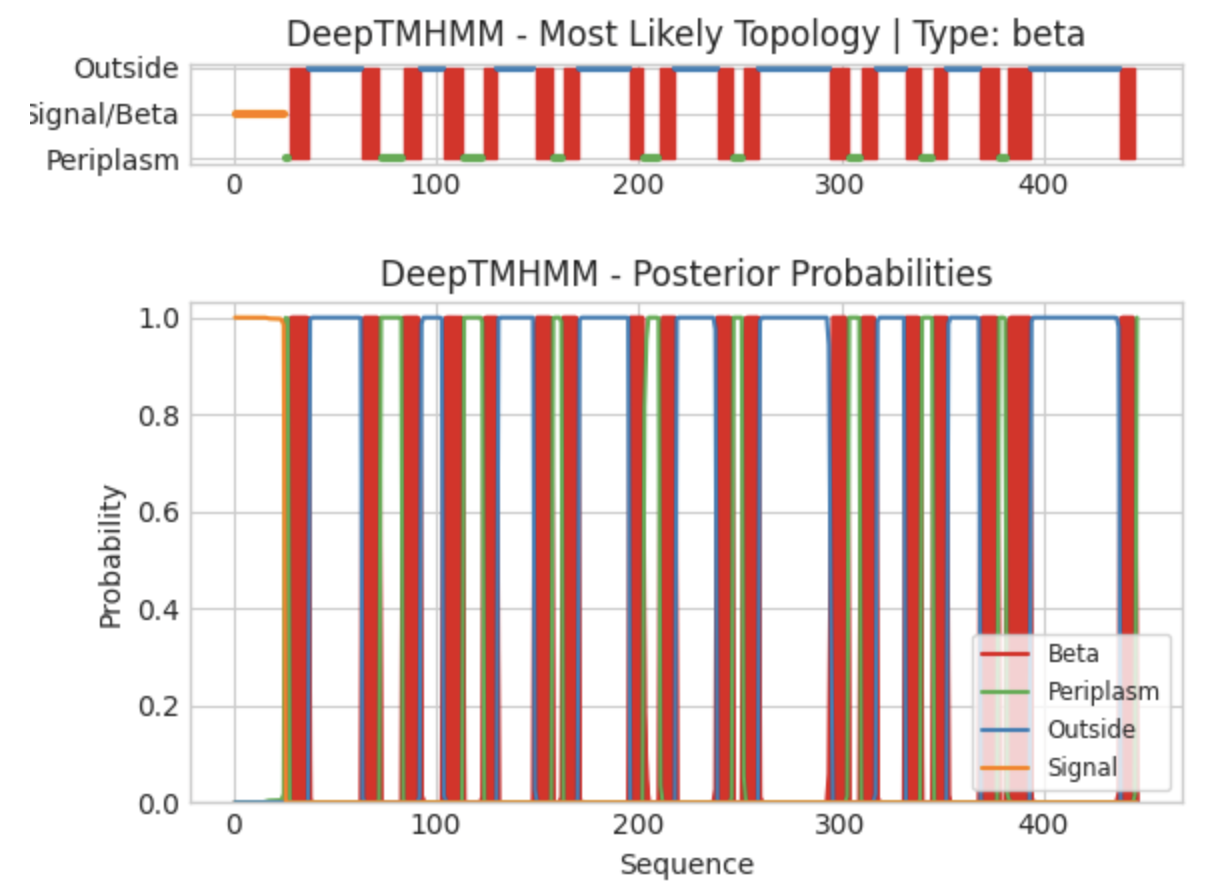
На N-конце предсказан сигнал локализации белка, зеленым обозначены участки белка в периплазматическом пространстве, синим - во внеклеточном, красным - трансмембранные фрагменты. Предсказано 18 трансмембранных бетта-листов, это соответствует экспериментально полученной модели, однако границы трансмембранных фрагментов в экспериментальной модели и в предстказанной совершенно не совпадают.
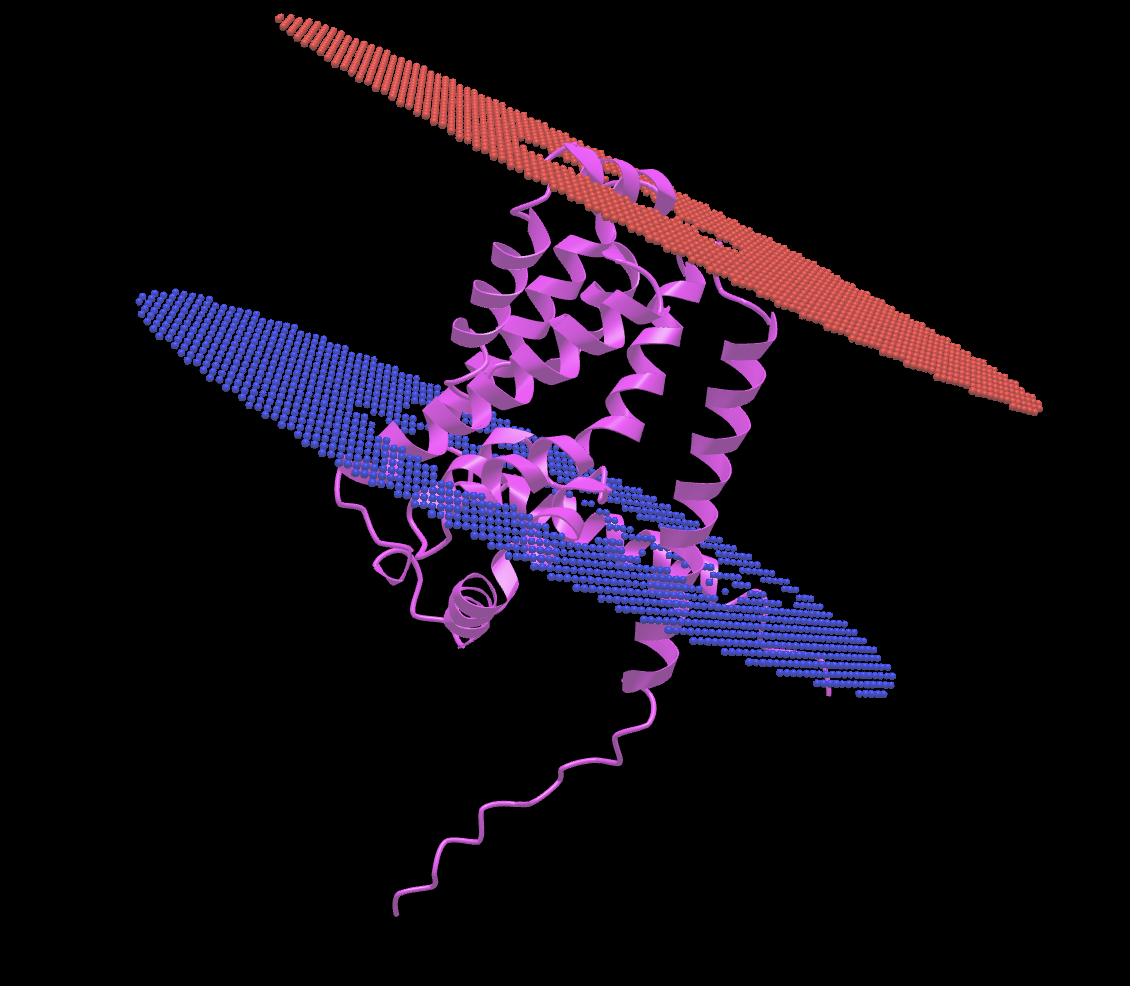
Задание 3. PPM: Предсказание положения выданного белка в мембране
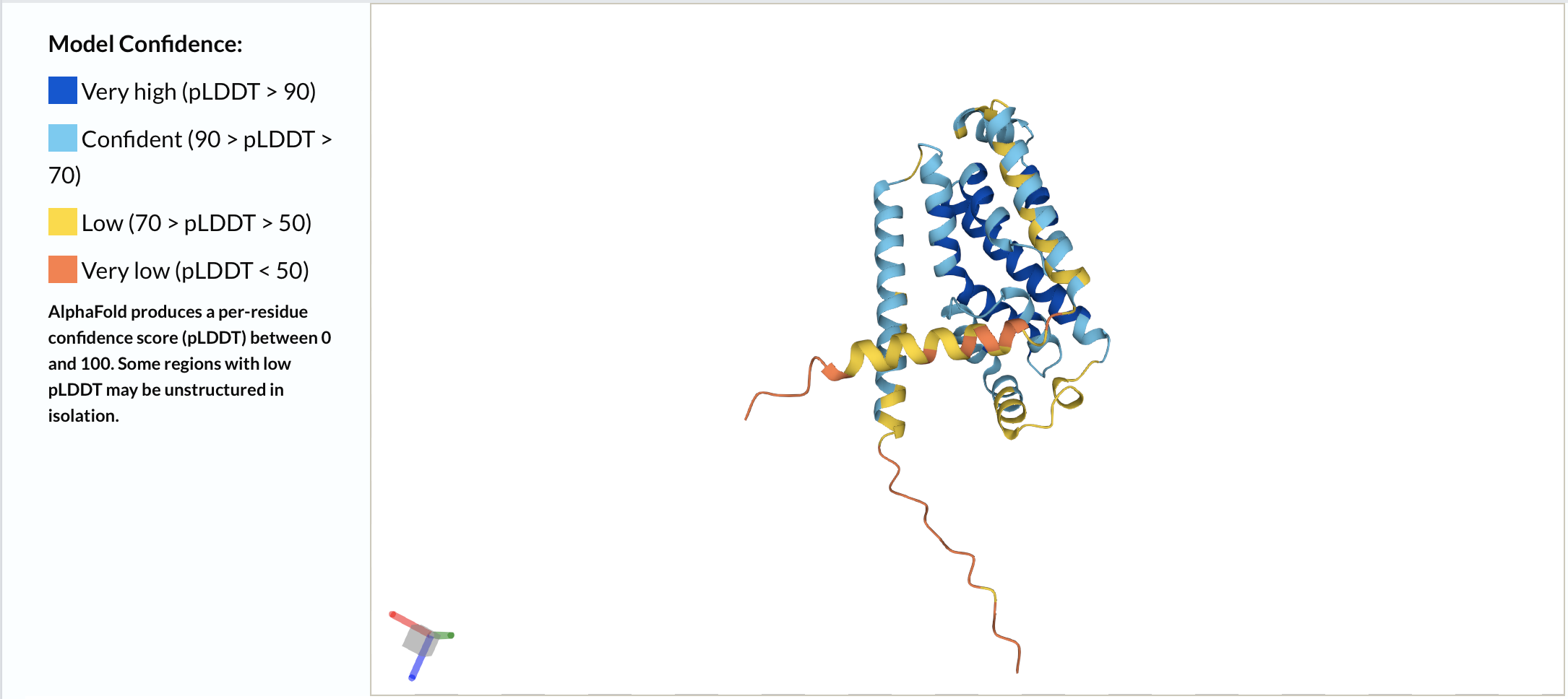
При формировании запроса на моделирование положения белка в мембране нужно ввести ряд параметров моделирования. Для предсказания положения в мембране выбрана плазмолемма грамположительной бактерии, таковой является палочка Коха, белок из которой мы берем для предсказания. Параметр "разрешить искривление" по умолчанию имеет значение "нет", я рассматриваю это как более распространенный среди белков случай (отсутствие искривления мембраны вокруг них). Топология N-конца белка - внутриклеточная, по результатам предсказания DeepTMHMM. Загружен для предсказания был PDB-файл Для простоты моделирования положения белка в мембране, игнорируется гетороатомы и вода, информация о которых имеется в PDB-файле. Результаты моделирования приведены в таблице 2 и на рисунке 4. Файл PDB, визуализированный ниже, доступен по ссылке.
Толщина внутримембранной части белка |
32.3 ± 1.2 Å |
Угол наклона белка в мембране |
13 ± 1° |
Координаты трансмембранных участков |
1( 29- 48), 2( 66- 82), 3( 86- 98), 4( 99- 117), 5( 180- 203), 6( 208- 227) |
Среднее количество остатков в одной альфа-спирали |
19 |
Задание 4.Сравнение алгоритмов предсказания трансмембранных участков белка Y1491_MYCTO
- Границы предсказанных трансмембранных участков согласно DeepTMHMM:
- 1( 32- 49), 2(67- 84), 3(87- 97), 4(101- 121) , 5(180- 199), 6( 209- 229)
- Границы предсказанных трансмембранных участков согласно PPM:
- 1( 29- 48), 2( 66- 82), 3( 86- 98), 4( 99- 117), 5( 180- 203), 6( 208- 227)