Практикум 4
Составление списка гомологичных белков, включающих паралоги
В этом практикуме я работаю с тем же списком видов бактерий, что и в практикумах 1, 2, видовые названия и мнемоники бактерий приведены в таблице 1 ниже. Первая задача практикума - найти в протеомах этих бактерий достоверные гомологи белка CLPX_ECOLI.
Название |
Мнемоника |
Acidothermus cellulolyticus |
ACIC1 |
Arthrobacter sp. |
ARTS2 |
Bifidobacterium longum |
BIFLO |
Clavibacter michiganensis |
CLAMS |
Corynebacterium diphtheriae |
CORDI |
Corynebacterium efficiens |
COREF |
Leifsonia xyli |
LEIXX |
Для этого я скопировал к себе в рабочую директорию на kodomo полные протеомы отобранных бактерий и последовательность белка CLPX_ECOLI, объединив их в один файл, проиндексировал его и провел поиск в нем командой blastp, установив порог на E-value, равный 0,001. Я получил следующие результаты (Табл.2):
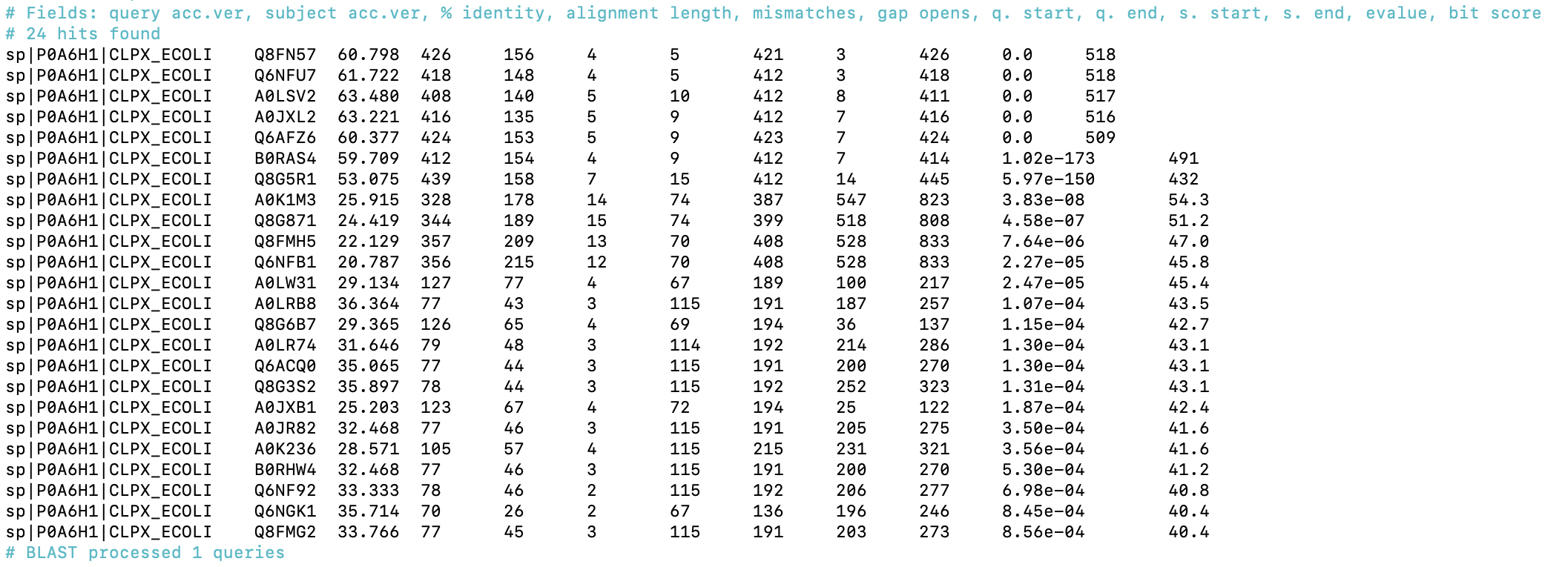
Реконструкция и визуализация
Последовательности находок blastp я поместил в отдельный fasta-файл, доступный по ссылке. Я заменил полные названия последовательностей на их ID для удобства чтения подписей филогенетического древа.
При помощи инструментов на сайте NGPhylogeny.fr, множественного выравнивания программой MAFFT и построения филогенетического древа по алгоритму минимальной эволюции FastME, я получил филогенетическое древо. Файл с его записью в формате Newick доступен по ссылке.
Примеры паралогов среди рассматриваемых белков: CLPX_BIFLO и Q8G871_BIFLO, CLPX_ARTS2 и A0JR82_ARTS2, Q6NFB1_CORDI и CLPX_CORDI.
Примеры ортологов среди рассматриваемых белков: CLPX_COREF и CLPX_CORDI, CLPX_COREF и CLPX_BIFLO, A0LRB8_ACIC1 и A0JR82_ARTS2.
Ниже представлены изображения полученного дерева, укорененного в среднюю точку. На рисунке 1 крупные группы ветвей, элементы которых образуют между группами пары ортологов, покрашены в разные цвета:
В красную группу входят ClpX субъединицы АТФ-зависимых протеаз Clp всех семи рассматриваемых видов бактерий.
В синюю группу также белки с предсказанной схожей функцией: АТФ-связывающие субъединицы протеазы Clp, пример - Q8FMH5_COREF - Putative endopeptidase Clp ATP-binding chain C. Возможно, что данные предсказания сделаны на основании схожести в аминокислотной последовательности этих гипотетических белков с исследованной лучше субъединицей ClpX. В этой группе содержатся белки из протеомов бактерий видов Arthrobacter sp., Bifidobacterium longum, Corynebacterium diphtheriae, Corynebacterium efficiens
В желтую группу входят АТФ-зависимые цинковые металлопротеазы всех видов изучаемых бактерий.
На следующем изображении рассмотренные группы ортологов схлопнуты, заменены единым цветным треугольником (Рисунок 2):