Практикум 6. Сигналы и мотивы
3'- и 5'-концевые последовательности интронов как пример сигнала, закодированного в геноме
Для сплайсинга мРНК в клетках эукариот необходимо наличие специальных 3'- и 5'-концевых последовательностей, а также точки ветвления в интроне (участке пре-мРНК, вырезаемом при её созревании). Различают разные интроны, с немного различными консервативными участками узнавания сплайсосомой. От этого зависит то, какой тип сплайсосомы их узнает. Наиболее распростаненный в геноме вариант "col-md-8"интронов имеет последовательности GU на 5'-конце и AG на 3'-конце интрона в пре-мРНК, и вырезается при созревании мРНК главной сплайсосомой. Сигнал является умеренно сильным, потому что в большинстве случаев из одного гена получается путем сплайсинга зрелая мРНК одного вида (с вырезанными интронами, но не экзонами). Но и пропуск сигнала имеет биологическое значение, когда сплайсосома взаимодействует с 5' и 3' последовательностями разных интронов, и может удалить из созревающей мРНК экзоны, находившиеся между ними. Это явление называется альтернативным сплайсингом. [1]
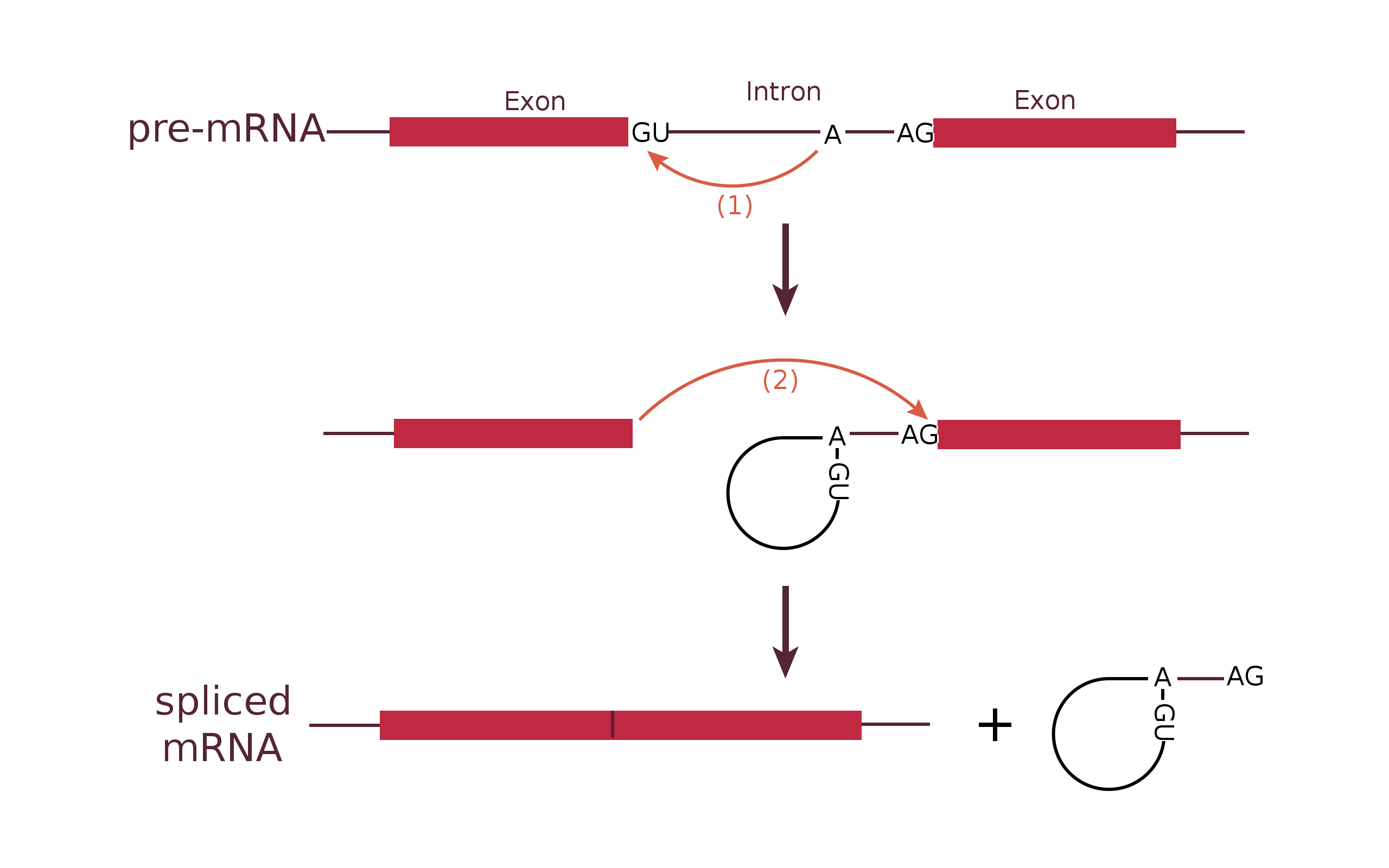
Источники:
- "PRE-mRNA SPLICING", Michael R. Green, 1986. https://www.annualreviews.org/doi/pdf/10.1146/annurev.ge.20.120186.003323?casa_token=oLH78d16pF4AAAAA%3AmWj9jrJTY3b9YwweHKr7b26bVK2ftidDfeoRj4Bm1FxlHxSfM9Fqpf0uSsYZT4laae90FUumgEGzBfUz
Построение PWM последовательности Козак Homo sapiens, поиск по ней сайтов в других генах
Техническую часть задания я выполнил в Colab Notebook при составлении которого я воспользовался скриптами авторства моего однокурсника Петра Милейко. Скрипт сначала работает с генами с известными координатами начальной позиции ATG, переписывая в файл learn_genes.txt построчно 100 последовательностей вида 7 нуклеотидов до ATG + ATG + 3 нуклеотида после ATG (обущающая выборка последовательностей Козак). С помощью этой выборки будет строится PWM.
Далее создается аналогичная выборка из 200 аналогичных случайных последовательностей в файле test_genes.txt (тестовая выборка последовательностей Козак).
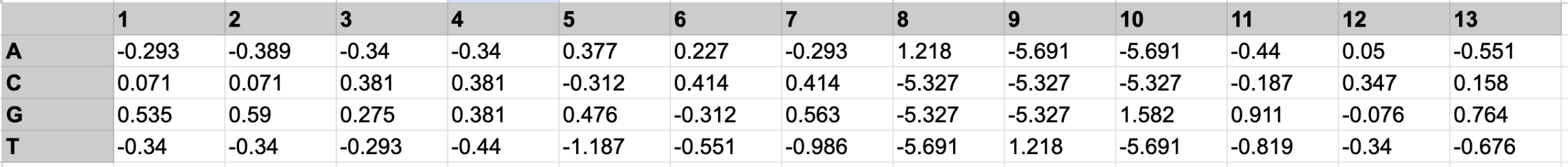
И создается выборка из окрестностей того же вида (7 нуклеотидов до ATG + ATG + 3 нуклеотида после ATG ) случайных двухсот сигналов ATG внутри генов в геноме. Это выборка негативного контроля. По результатам работы скрипта была получена таблица PWM последовательности Козак человека, представленная ниже (Таблица 1).
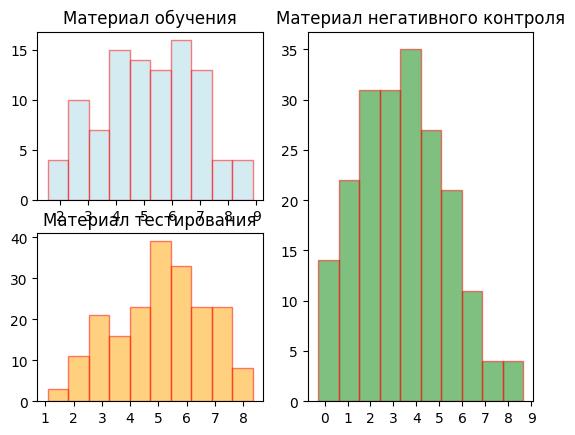
На рисунке 2 представлены диаграммы распределения весов в трех выборках. Визуально порог веса равный 4 отсекает много сигналов в отрицательном контроле, при этом выявляя сигнал в обучающей выборке. Результат проверки с порогом 4 представлен в таблице 2.
| Сигнал - | Сигнал + | Объем выборки | |
| Обучающая выборка | 27 | 73 | 100 |
| Тестовая выборка | 51 | 149 | 200 |
| Негативный контроль | 130 | 70 | 200 |
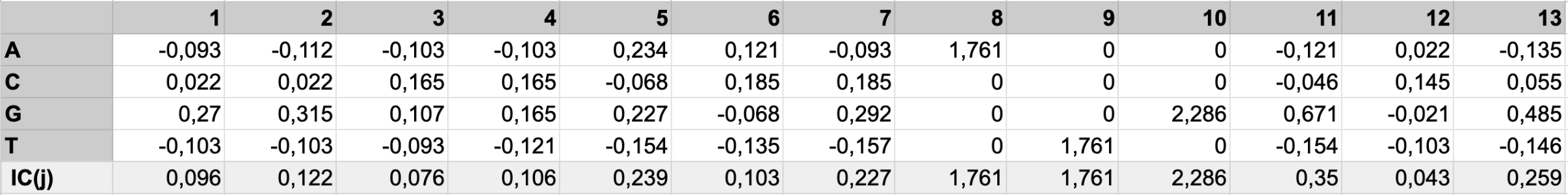
Задание 3. Информационное содержание и LOGO
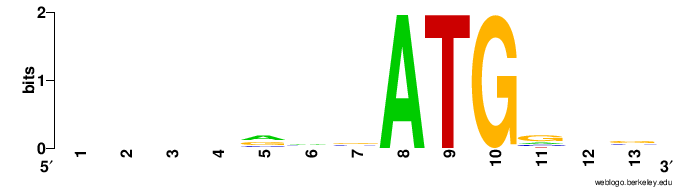
Задание 4.
Для выполнения этого задания я выбрала штамм Escherichia coli str. K-12 substr. MG1655. В геноме получен 646 сайт последовательности GAATTC. Ожидаемое число этих сайтов составило 1141. Считая, что количество лучайных сайтов одного вида в геноме распределено по Пуассону, была посчитана вероятность наблюдать такое или меньшее число сайтов вида GAATTC. Различие оказалось статичстически значимым (p-value = 1.77e-49).