Практикум 8
Задание 1. Консервативный мотив в выравнивании последовательностей гомологичных белков
В этом практикуме я работаю с доменом S-layer (PF07752). Для удобства составления паттерна по консервативному участку выравнивания seed, в выравнивании должно быть не больше нескольких десятков последовательностей. здесь их 44. По описанию на странице домена, Methanosarcinales S-layer Tile Proteins (STP) - это семейство белков, встречающихся почти исключительно у членов порядка Methanosarcinales, которые являются метаногенными археями. STP являются основным компонентом структуры защитного белкового поверхностного слоя (S-слоя), который покравает снаружи мембрану Methanosarcinales и выполняет многие важные функции, включая защиту от вредных внеклеточных веществ.
Я скачал файл с выравниванием seed, открыл его в Jalview и визуально определил интересный мне консервативный участок (выделение на рисунке 1). Его паттерн в Jalview-формате: [YF]..[IMVL][GPYN][FWL][MFLN][AGCS].[KERPLNQ][YW][FVIA][AVPY]; в PROSITE-формате: [YF]-x-x-[IMVL]-[GPYN]-[FWL]-[MFLN]-[AGCS]-x-[KERPLNQ]-[YW]-[FVIA]-[AVPY]
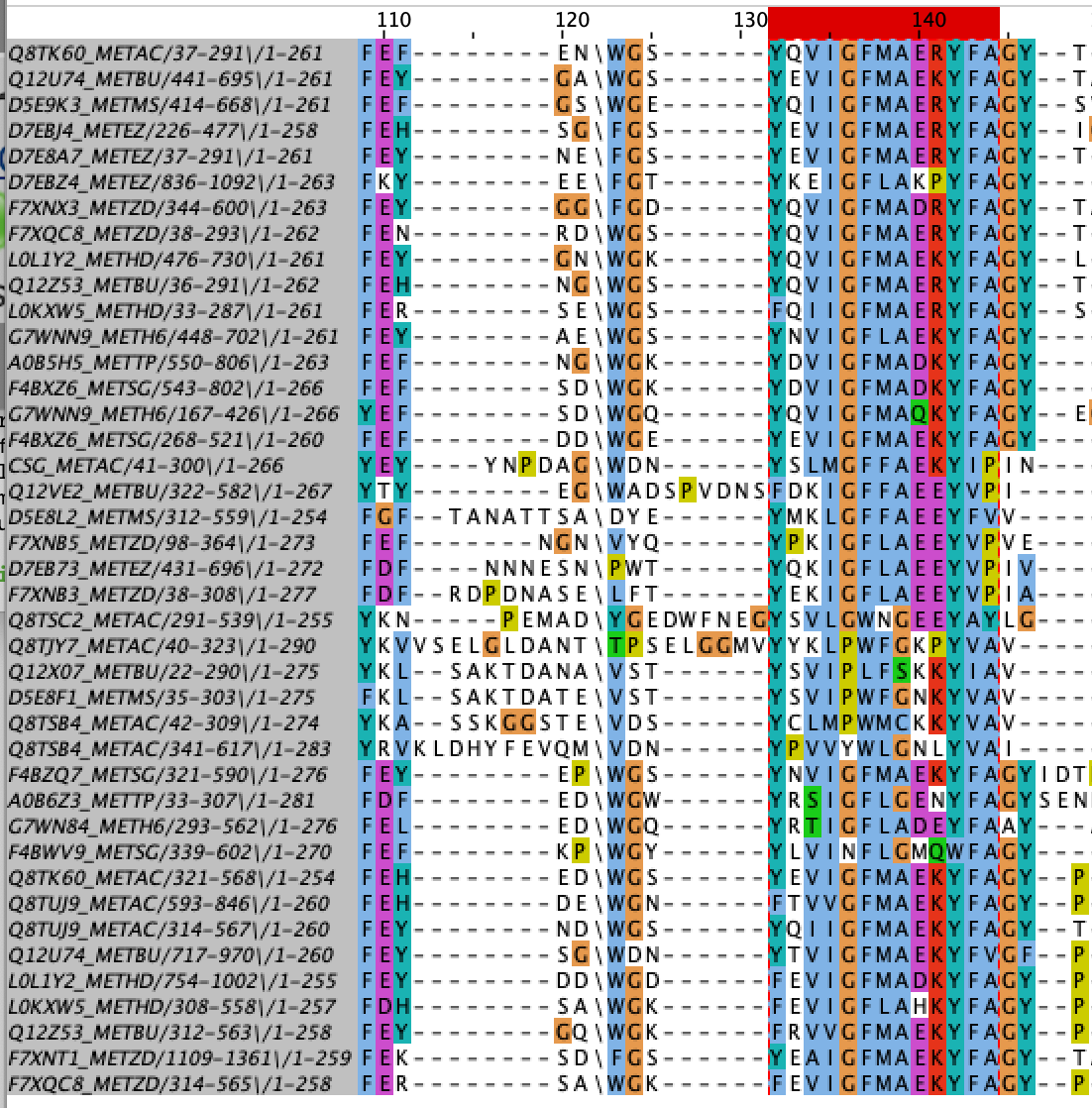
Я проверил с помошью функции поиска в Jalview, что паттерн описывает все 44 последовательности. При этом в базе SwissProt при помощи сервиса myhits нашлось всего два белка из архей. Идентификаторы: FIMA1_PORGN и FIMA1_PORG3. В них не описано домена PF07752, они не относятся к изучаемому семейству. Эти белки - Type-1 fimbrial protein, A chain - субъединицы белков, функция которых связана с микроворсинками на поверхности бактерий. Видна функциональная параллель белков с этим консервативным участком в организмах двух разных доменов - Бактерий и Архей.
Задание 2. Мотив, специфичный для одной клады филогенетического дерева
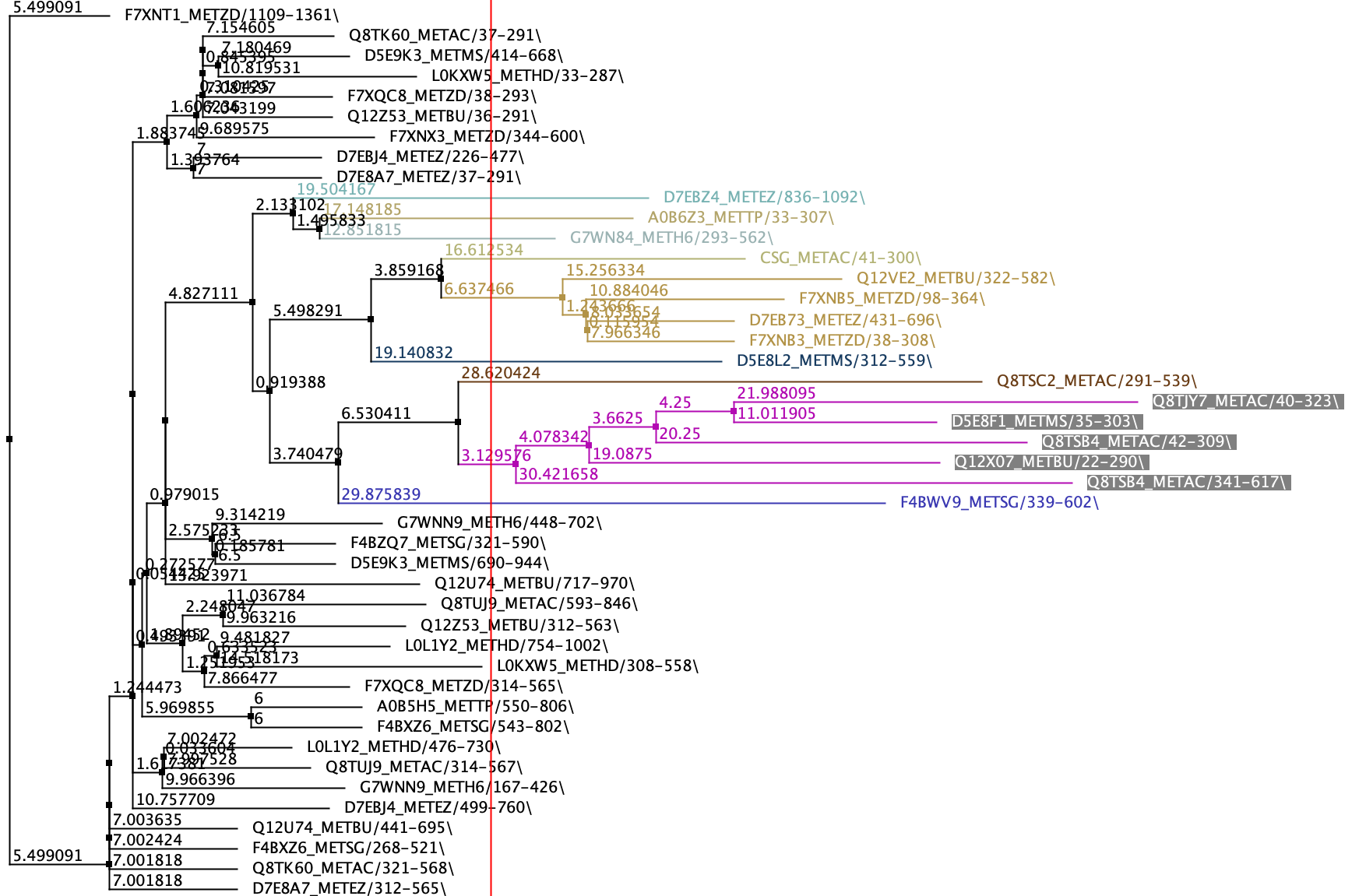
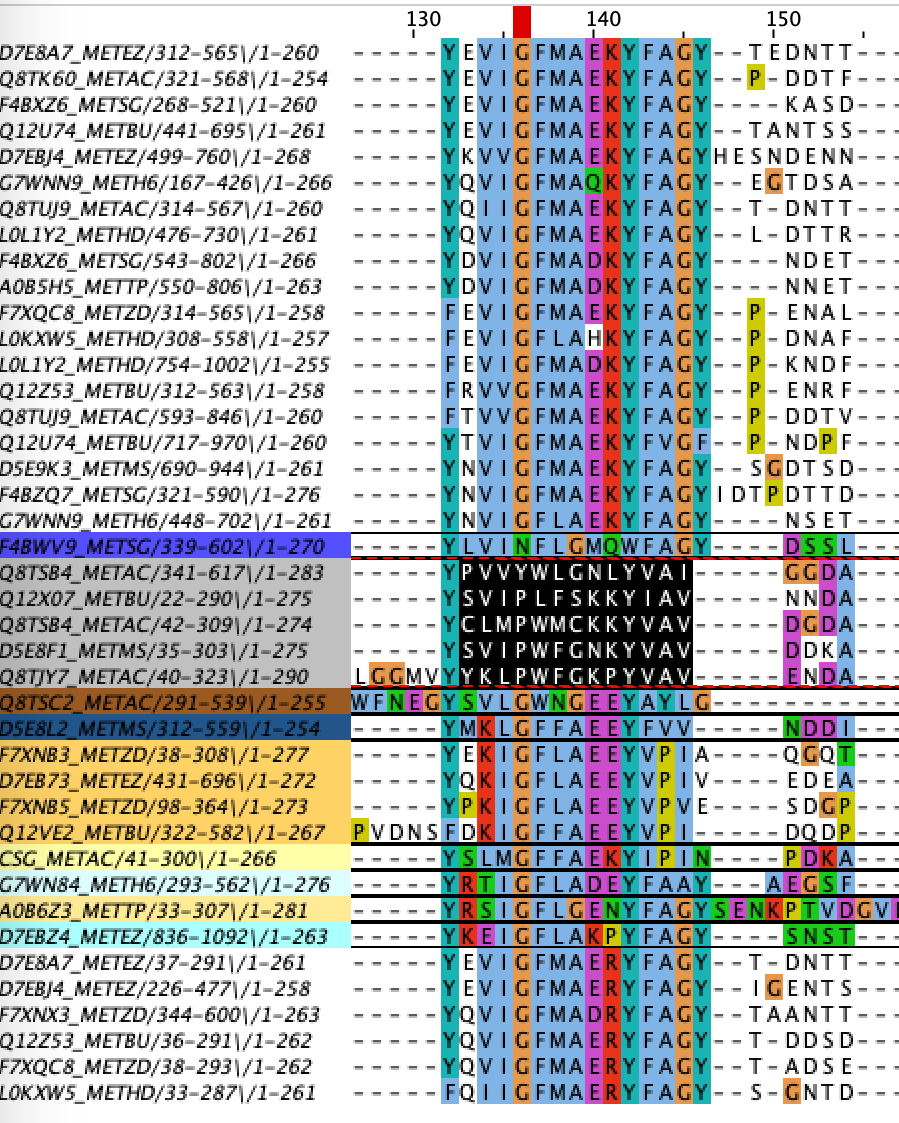
При помощи встроенного функционала Jalview я построил филогенетическое древо белков из seed. Алгоритм построения дерева - Neighbor Joining. Дерево с выделением цветом части ветвей изображено на Рисунке 2.
Задание 3. PSI-BLAST последовательности белка B2V8C0
По материалам Википедии, Z-кольцо - оно же септальное кольцо — кольцевая органелла грамотрицательных бактерий, расположенная примерно посередине клетки и способная сокращаться, образуя перетяжку между двумя новыми дочерними клетками. Согласно описанию функции белка B2V8C0 в Uniprot, Probable septum site-determining protein MinC - ингибитор деления клеток, который блокирует образование полярных Z-кольцевых перегородок. Он быстро колеблется между полюсами клетки, чтобы дестабилизировать нити FtsZ, которые сформировались, до того, как они созрели в полярные Z-кольца. Предотвращает полимеризацию FtsZ.
Я запустил psi-blast этого белка по базе SwissProt и пронаблюдал за количеством и E-value находок на каждой итерации (продолжал до момента, пока перестали появляться новые находки). Результаты - в таблице, эта же таблица приведена ниже:
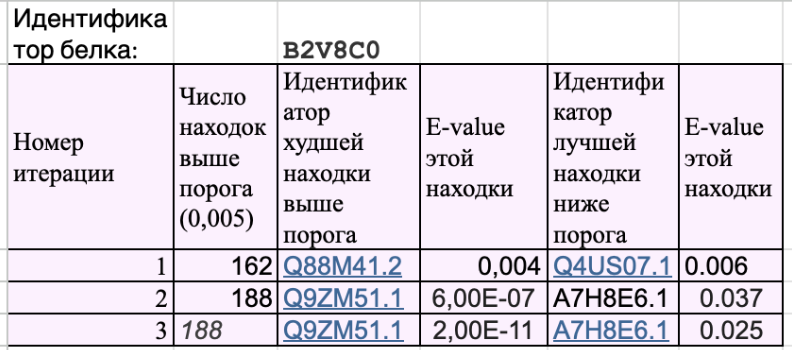
На третьей итерации не было новых находок. Обратим внимание на разницу в E-value ближайших над- и подпороговых находок. Разница растет с каждой итерацией, и на первой итерации многие находки близки к порогу, при более строгом пороге они бы отсеялись как незначимые. Однако повторный запуск не только находит новые белки, но и усиливает "неслучайность" каждой находки. В третьем запуске разница в E-value ближайших над- и подпороговых находок имеет вид большого скачка значимости, сравнивая их, можно объединить надпороговые находки в семейство гомологичных белков.
Задание 3. Недопредставленность слова TA в геноме
Проверим гипотезу, что слово TA встречается в геноме меньше раз, чем это ожидается исходя из GC-состава генома. Я взал референсный геном всем известной Bacillus subtilis subsp. subtilis str. 168, ожидаемое количество TA в геноме по формуле "(число нуклеотидов в хромосоме) x (частота A в хромосоме) x (частота T в хромосоме)"= 336259, наблюдаемое = 218025; Наблюдаемое количество в 0.648 раз меньше ожидаемого. Гипотеза верна.