8. UniProt Proteomes, EMBOSS, bash
Исследование протеома родственных Burkholderia seminalis бактерий
1. Выбор и скачивание протеомов
В прошлом практикуме я обозревал белок Burkholderia seminalis. При поисковом запросе (organism_id:488731)(id Burkholderia seminalis) был выдан только один референсный протеом, но проблема заключалась в том, что он был полностью дуплицирован. В связи с этим было принято решение искать протеомы ближайших родственников. При поисковом запросе (organism_name:Burkholderia) представлялся довольно скудный выбор. Был выбран референсынй протеом Burkholderia plantarii (PG1) (Proteome ID: UP000031838), потому что во-первых, значение его CPD – Standard, то есть размер протеома близок к среднему по систематической группе. Во-вторых, BUSCO показывал 99.6% покрытия ортологичных генов , при этом на Single приходится 99%. В третьих жизнедеятельность Burkholderia plantarii (PG1) ассоциирована с растениями [2], как и у Burkholderia seminalis.
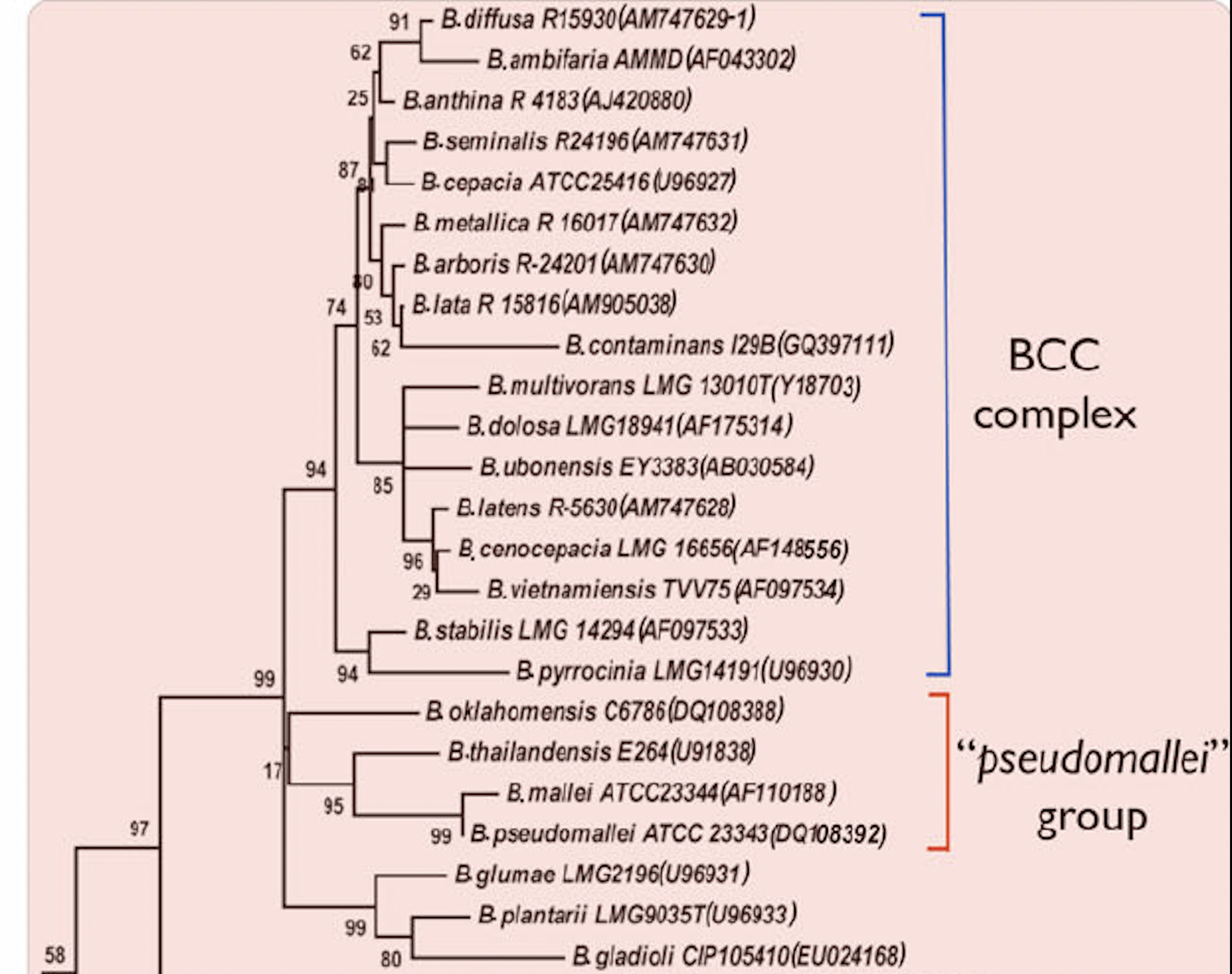
Выбор контрольного протеома осуществлялся с помощью запроса (organism_name:Burkholderia) и был выбран реферненсный протеом Burkholderia multivorans (Proteome ID: UP000008815). Она систематически близка к исследуемой бактерии [1], но при этом является патогенным для человека организмом, вызывающим одну из форм муковисцидоза и бактериальный менингит. Значение CPD – Standard, BUSCO: C:99.7% (S:99.1% D:0.6%). Выбранный протеом хороший. Размер протеома Burkholderia plantarii - 6,432, Burkholderia multivorans - 6,040.
2. Сравнение протеомов по представленности определенных групп белков
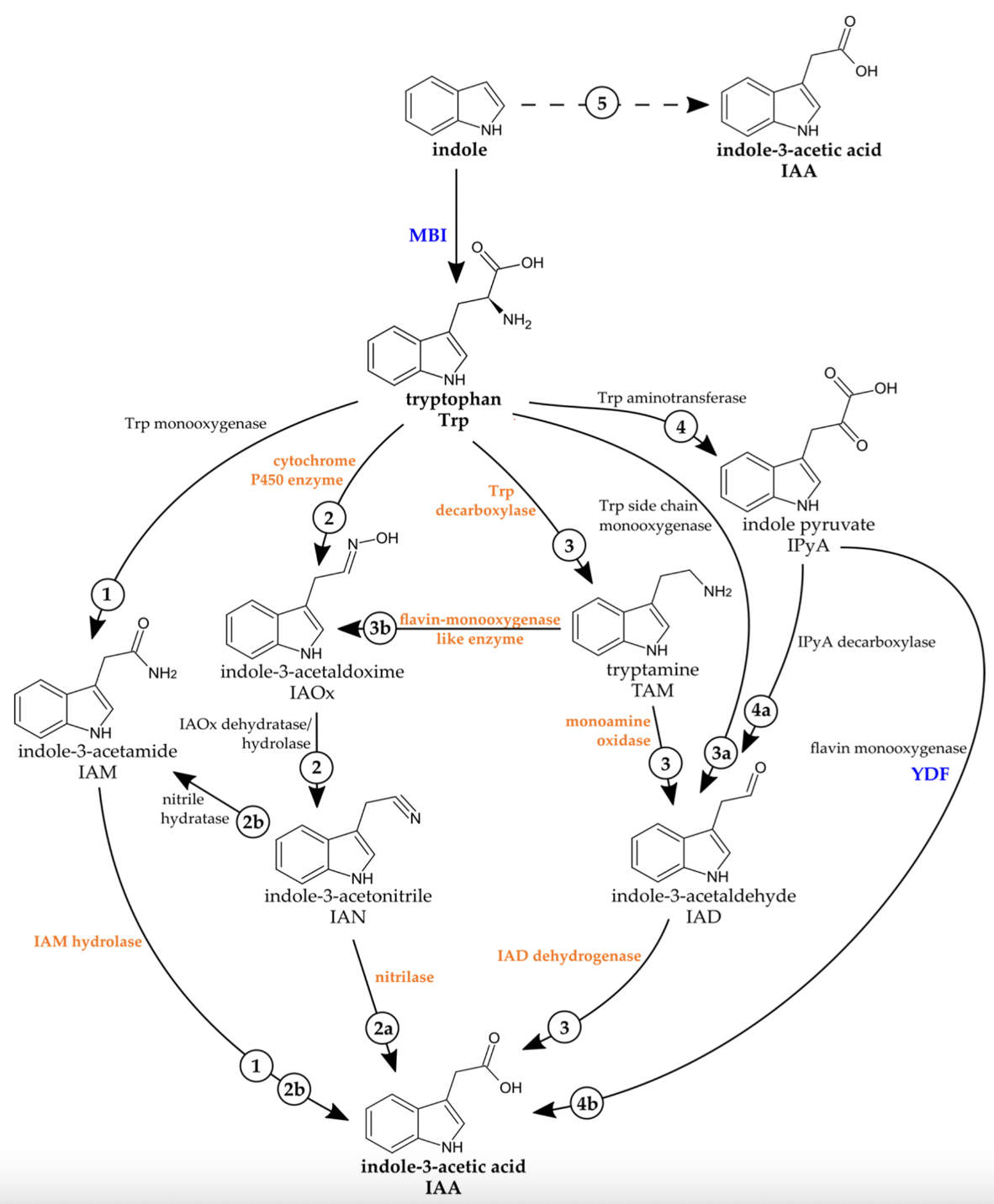
Поисковые запросы для подсчета количества белков из разных функциональных групп указаны ниже. Как видно из таблицы, количество трансмембранных белков и ферментов у обоих исследуемых бактерий примерно равное количество. В качестве двух других функциональных групп были выбраны ферменты флавинмонооксигеназа и нитрилаза. Эти ферменты используются для синтеза индол-3-уксусной кислоты (ауксина) – растительного гормона, ингибирующего рост корней, что и было отмечено при заражении риса B.plantarii [3]. Из таблицы видно, что представленность ферментов синтеза ауксина в протеоме у B.multivorans ниже, чем у B.plantarii.
Поисковые запросы:
Burkholderia plantarii:
(proteome:UP000031838) AND (ft_transmem:*)
(proteome:UP000031838) AND (ec:*)
(proteome: UP000031838) AND (protein_name:flavin monooxygenase)
(proteome: UP000031838) AND (protein_name:nitrilase)
Burkholderia multivorans:
(proteome: UP000008815) AND (ft_transmem:*)
(proteome: UP000008815) AND (ec:*)
(proteome: UP000008815) AND (protein_name:flavin monooxygenase)
(proteome: UP000008815) AND (protein_name:nitrilase)
| Функциональная группа белков | Burkholderia plantarii | Burkholderia multivorans |
|---|---|---|
| Трансмембранные | 1 147 | 1 162 |
| Ферменты | 1 460 | 1 579 |
| Флавинмонооксигеназы | 7 | 2 |
| Нитрилазы | 5 | 2 |
3. Сравнение протеомов оп ключевым словам
Я провел сравнение двух протеомов по ключевым словам с помощью скрипта (ссылка на скрипт в google colab.). В таблице для наглядности представлены топ-10 по встречаемости слов в описании протеомов двух бактерий. Я составил список слов, входящих в 10% самых встречающихся слов и присутсвтующих в обоих протеомах. Количество этих слов поделил на количество слов, входящих в топ-10% у отдельной бактерии. Таким образом получилось, что состав ключевых слов описания протеомов совпадает на 42%. В основном в топе расположены слова, характеризующие белки базового метаболизма. Из интересного встречаются слова "methyltransferase", "iron-sulfur", "flagellum".
| # | Burkholderia plantarii | кол-во | Burkholderia multivorans | кол-во |
|---|---|---|---|---|
| 1 | reference | 6432 | reference | 6040 |
| 2 | proteome | 6432 | proteome | 5699 |
| 3 | transmembrane | 2518 | transmembrane | 2490 |
| 4 | membrane | 2219 | membrane | 2174 |
| 5 | helix | 1176 | helix | 1166 |
| 6 | cell | 987 | cell | 1027 |
| 7 | transcription | 834 | transcription | 729 |
| 9 | transferase | 739 | signal | 672 |
| 10 | transport | 636 | transport | 644 |
Ссылки
[1] DOI:10.1007/s00248-011-9929-1
[2] DOI:10.1094/PDIS-07-15-0765-PDN
[3] DOI:10.1016/j.idcr.2018.01.002