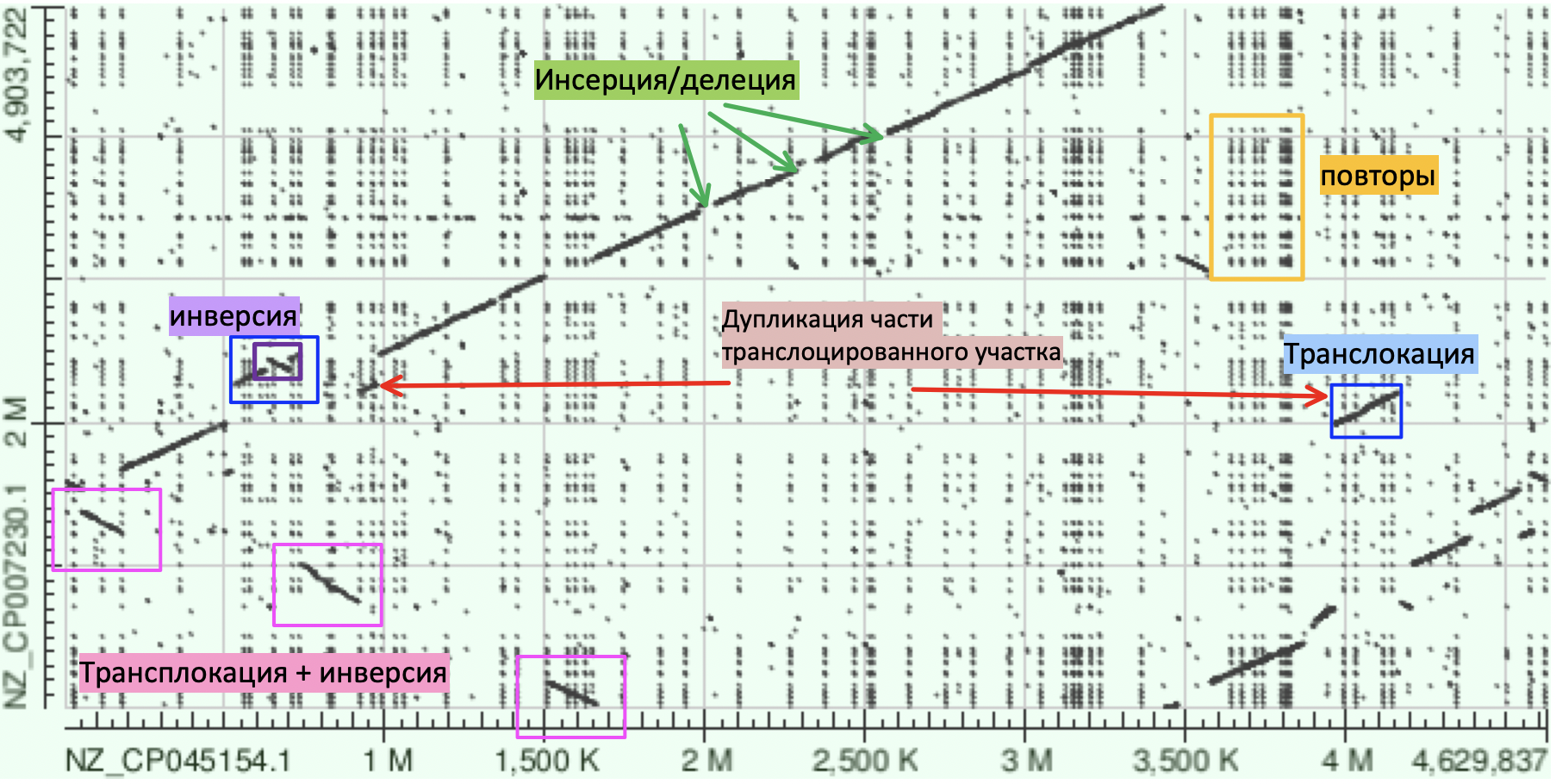
На графике видно, что у этих двух последовательностей разная точка начала отсчета (как бы место разрыва кольцевой молекулы ДНК). Об этом говорит съехавший вниз участок выравнивания с 3500К позиции по 4629К. Мы видимо что даже при выставленной длине слова 28 megablast находит большое количество повторов в геноме (точки на графике не образующие четких линий). Аналогичный график, полученный с помощью blastn содержит еще большее повторов (см fig.2). Мы так же видим, что есть участки которые притерпели транслокацию, для примера некоторые из них обведены в синий квадрат. При этом некоторые из притерпевших транслокацию учатсков притерпели еще и инверсию, такие участки обведены в розовый квадрат на графике. Левый участок, выделенный в синий квадрат, помимо транслокации притерпел еще и инверсию своей части, выделенной фиолетовым квадратом. Еще мы можем видеть, что правый участок, выделенный синим квадратом, помимо транслокации подвергнулся дупликации своей части. Эта небольшая часть и участок из которого она вероятно получилась помечены красными стрелками. Зелеными стрелками обозначены участки, где произошла инсерция/делеция участков последовательности, на это указывает характерный разрыв прямой. Несмотря на все геномные перестройки довольно приличная часть генома остается консервативной.
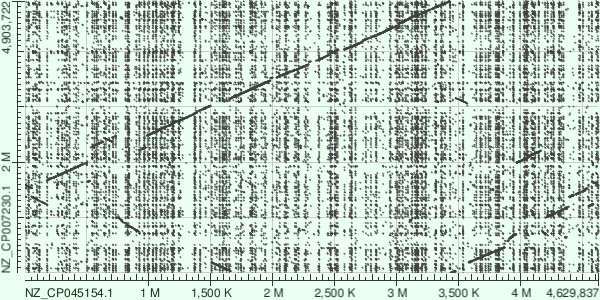
График, полученный в результате работы blastn дает нам более полное представление о том, что рассматриваемые геномы очень богаты повторами. Кратно большее количество замеченных повторов обеспечено меньшей длиной слова при работе blastn, что позволяет их лучше выцеплять.