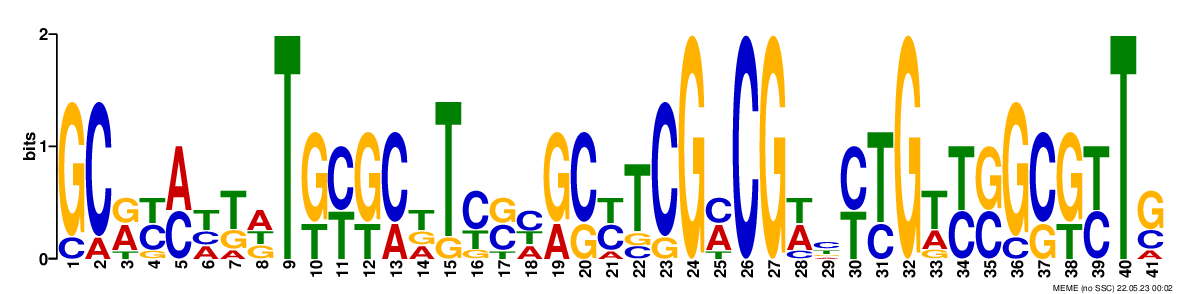2. Запуск MEME
Для поиска сигналов в последовательностях был использован локальный MEME со следующими параметрами:
поиск в ДНК (-dna)
число мотивов для поиска — 3 (-nmotifs 3)
длина находки от 6 до 50 нуклеотидов (-minw 6 -maxw 50)остальное по умолчанию
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50Скачать выдачу MEME
| Находка | E-value |
|---|---|
| GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS | 8.9e-009 |
| TAGCABTACSCCYKCGTGAHDCCACCTTYYAKCGWDTVAGDGSBTGCCA | 1.9e-002 |
| WTSGACRCCTYTTTCWSTTT | 1.3e-001 |
Был выбран мотив c наименьшим E-value (первый):
GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTSЕго паттерн (пункт "regular expression" в текстовой выдаче):
GC[AG][CT][AC][TAC][TG][AGT]T[GT][CT][GT][CA][TAG]T[CT][CG][CAT][GA][CG][CT]TCG[AC]CG[AT][CT][CT][TC]G[TA][TC][GC]G[CG][GT][TC]T[GC]
3. Запуск FIMO
Для поиска мотива в положительном и отрицательном контроле были запущены следующие команды:
fimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.0125 ./meme_out/meme.txt promoters.fastafimo --norc -motif GCRYMHKDTGYGCDTYSHGCYTCGMCGWYYTGWYSGCGYTS -thresh 0.0125 ./meme_out/meme.txt negative.fasta
Поиск проводился только по одной цепи (--norc), сначала порог на p-value (-thresh) был взят 0.01, получилось 3368 находок (fimo_001.tsv) в материале тестирования. Это маловато, ведь всего в нем 4445 промоторных последовательностей, плюс не все сайты посадки нашлись. С порогом на p-value 0.05 было найдено 15285 находок (fimo_005.tsv), это избыточно. Таким образом был взят, наилучший вариант порога 0.0125 – 4175 находок (fimo_00125.tsv), примерно столько же, сколько и промоторов.
В негативном контроле с теми же параметрами было найдено всего 449 последовательностей (fimo_00125_negative.tsv)
| Порог E-value | Ссылка на файл выдачи FIMO |
|---|---|
| 0.01 | Скачать |
| 0.05 | Скачать |
| 0.0125 | Скачать |