с штрафами по умолчанию:
* 180 * 200 * 220 * 240
PURT_ECOLI : QTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEGVVKFDFEITLLTVSA--VDG---VHFCAPVGHRQEDGDYRESWQPQQMSPL : 242
PURT_RHOFD : QSKISGPADVQKAWDYAMAGGRVSHGRVIVEGFIDFDYEITLLTVRAKGADGQIETHFCEPVGHLQVNGDYVESWQPHPMHPA : 249
Q3 I 6 AW YA GGR GRVIVEG 6 FD5EITLLTV A DG HFC PVGH Q 1GDY ESWQP M P
с увеличенными штрафами:
* 180 * 200 * 220 * 240
PURT_ECOLI : QTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEGVVKFDFEITLLTVSAVDG-----VHFCAPVGHRQEDGDYRESWQPQQMSPL : 242
PURT_RHOFD : QSKISGPADVQKAWDYAMAGGRVSHGRVIVEGFIDFDYEITLLTVRAKGADGQIETHFCEPVGHLQVNGDYVESWQPHPMHPA : 249
Q3 I 6 AW YA GGR GRVIVEG 6 FD5EITLLTV A HFC PVGH Q 1GDY ESWQP M P
с штрафами по умолчанию:
* 100 * 120 * 140 * 160
PURT_ECOLI : EAIATDMLIQLEEEG-LNVVPCARATKLTMNREGIRRLAAEELQLPTSTYRFADS-ESLFREAVADIGYPCIVKPVMSSSGKG : 164
PURT_RHOFD : EAIATPMLEVLEATGVVRVIPTARAARLTMDREGIRRLAAETLGLPTSPYQFCDSFEELQAAIEGGIGYPCIVKPVMSSSGKG : 166
EAIAT ML LE G 6 V6P ARA 4LTM1REGIRRLAAE L LPTS Y F DS E L IGYPCIVKPVMSSSGKG
с уменьшенными в 2 раза штрафами:
* 100 * 120 * 140 * 160
PURT_ECOLI : EAIATDMLIQLEEEG-LNVVPCARATKLTMNREGIRRLAAEELQLPTSTYRFADSESLFRE---AV-ADIGYPCIVKPVMSSS : 161
PURT_RHOFD : EAIATPMLEVLEATGVVRVIPTARAARLTMDREGIRRLAAETLGLPTSPYQFCDS---FEELQAAIEGGIGYPCIVKPVMSSS : 163
EAIAT ML LE G 6 V6P ARA 4LTM1REGIRRLAAE L LPTS Y F DS F E A6 IGYPCIVKPVMSSS
с увеличенными в 2 раза штрафами:
* 180 * 200 * 220 * 240
PURT_ECOLI : QTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEGVVKFDFEITLLTVSAVDG-----VHFCAPVGHRQEDGDYRESWQPQQMSPL : 242
PURT_RHOFD : QSKISGPADVQKAWDYAMAGGRVSHGRVIVEGFIDFDYEITLLTVRAKGADGQIETHFCEPVGHLQVNGDYVESWQPHPMHPA : 249
Q3 I 6 AW YA GGR GRVIVEG 6 FD5EITLLTV A HFC PVGH Q 1GDY ESWQP M P
с штрафами по умолчанию:
* 180 * 200 * 220 * 240
PURT_ECOLI : QTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEGVVKFDFEITLLTVSA--VDG---VHFCAPVGHRQEDGDYRESWQPQQMSPL : 242
PURT_RHOFD : QSKISGPADVQKAWDYAMAGGRVSHGRVIVEGFIDFDYEITLLTVRAKGADGQIETHFCEPVGHLQVNGDYVESWQPHPMHPA : 249
Q3 I 6 AW YA GGR GRVIVEG 6 FD5EITLLTV A DG HFC PVGH Q 1GDY ESWQP M P
с штрафами по умолчанию:
* 100 * 120 * 140 * 160
PURT_ECOLI : EAIATDMLIQLEEEG-LNVVPCARATKLTMNREGIRRLAAEELQLPTSTYRFADS-ESLFREAVADIGYPCIVKPVMSSSGKG : 164
PURT_RHOFD : EAIATPMLEVLEATGVVRVIPTARAARLTMDREGIRRLAAETLGLPTSPYQFCDSFEELQAAIEGGIGYPCIVKPVMSSSGKG : 166
EAIAT ML LE G 6 V6P ARA 4LTM1REGIRRLAAE L LPTS Y F DS E L IGYPCIVKPVMSSSGKG
с уменьшенными в 2 раза штрафами:
* 100 * 120 * 140 * 160
PURT_ECOLI : EAIATDMLIQLEEEG-LNVVPCARATKLTMNREGIRRLAAEELQLPTSTYRFADSESLFRE---AV-ADIGYPCIVKPVMSSS : 161
PURT_RHOFD : EAIATPMLEVLEATGVVRVIPTARAARLTMDREGIRRLAAETLGLPTSPYQFCDS---FEELQAAIEGGIGYPCIVKPVMSSS : 163
EAIAT ML LE G 6 V6P ARA 4LTM1REGIRRLAAE L LPTS Y F DS F E A6 IGYPCIVKPVMSSS
* 260 * 280 * 300 * 320 *
PURT_ECOLI : ALERAQEIARKVVLALG--------GYGLFGVELFVCGDEVIFSEVSPRPHDTGMVTLISQDLSEFALHVRAFLGLPVGGIRQ : 317
PURT_RHOFD : ALERARHIAKTVTDDLGIAVDGQASGLGIFGVELFVKGEQVWFSEVSPRPHDTGLVTLTTQWQSEFELHARAILGLPVNTALR : 332
ALERA IA4 V LG G G6FGVELFV G 2V FSEVSPRPHDTG6VTL 3Q SEF LH RA LGLPV
По данным записи в банке данных UniProt для белка PURT_ECOLI и белка PURT_RHOFD : у белков есть АТФ-связывающий домен, для белка PURT_ECOLI 119-308 а.о., для белка PURT_RHOFD 120-323 а.о., область сходства попадает в область домена (чуть выходит из области домена)
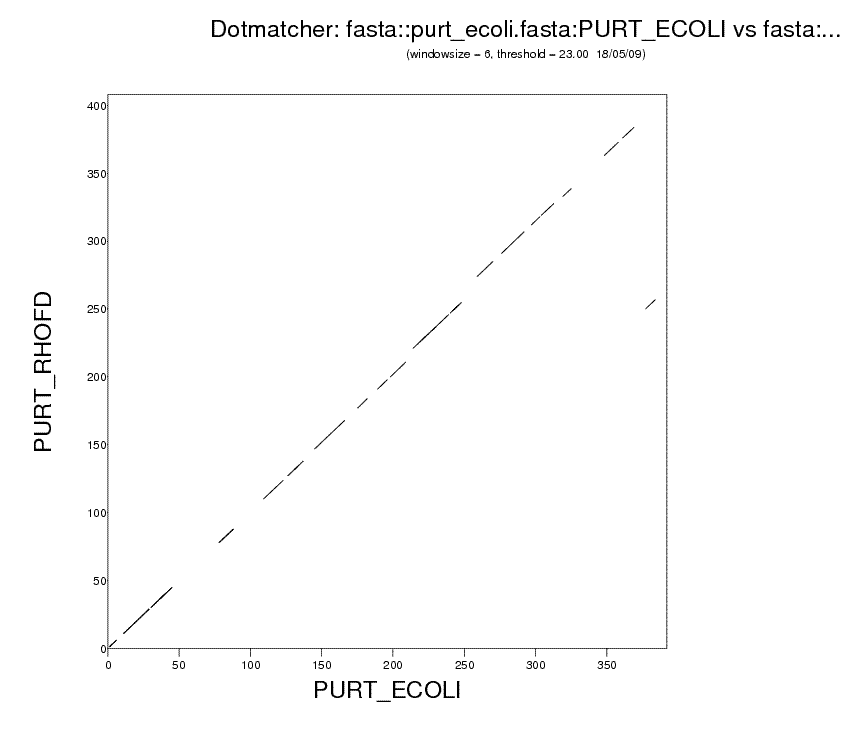
Построение карты локального сходства последовательностей с помощью программы dotmatcher пакета EMBOSS:
С помощью команды dotmatcher получен файл с расширением ps (dotmatcher.ps) (команда: dotmatcher purt_ecoli.fasta purt_rhofd.fasta -graph cps), в формате gif это выглядит так:
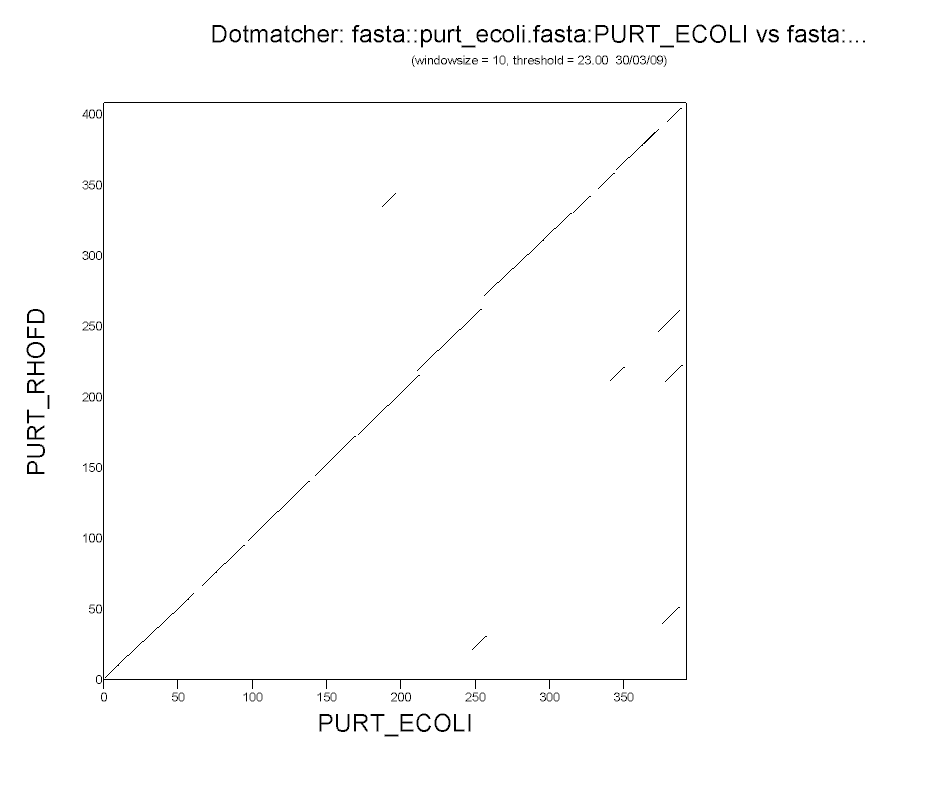
Из рисунка видно, что белки обладают высокой степенью сродства: линии идущие вдоль диагонали почти складывают непрерывную прямую, что говорит о том, что белки почти совпадают, причем схожие элементы расположены в них в схожих порядках. Видно также сходство конца 1 белка (по оси x) с началом 2 белка (по оси у). Линии идущие вдоль диагонали несколько сдвинуты по вертикали друг относительно друга, что говорит о гэпах: так, например, в районе 250 а.о 1 белка линия поднимается наверх - следующим а.о. 1 белка соответствуют а.о. 2 белка идущие с пропуском от предыдущих - во втором белке есть гэп в этом месте.
Получить несколько субоптимальных локальных выравниваний заданных последовательностей с помощью программы matcher пакета EMBOSS. Выбрать лучшие из них, в которых фрагменты последовательностей не совпадают с выровненными фрагментами в оптимальном выравнивании.
Субоптимальное локальное выравнивание используется для получения нескольких хороших локальных выравниваний при одних и тех же параметрах поиска.
Файл с 10 выравниваниями (aln_10_mat.matcher), штраф за гэп: 14, за продолжающийся гэп: 4.
На мой взгляд интересно выравнивание :
360
PURT_E GKPEIDGSRRLGV
:: : .::::
PURT_R GKEVIIALQRLGV
30
В нем представлены начало 2-го белка и конец 1-го - в глобальном выравнивании они не могли быть сопоставлены, ведь был бы слишком большой гэп слева от верхней последовательности и справа от нижней, но мы можем наблюдать эти фрагменты на карте локального выравнивания - в нижнем правом углу.
Аналогично выравнивание:
290 300 310 320
PURT_E HDTGMVTLISQDLSEFALHVRAFLGLPVGGIRQYGPA
: : .. : : .: . . ::. . .: :
PURT_R HATKVMLLGSGELGKEVIIALQRLGVETIAVDRYNHA
20 30 40
не могло наблюдаться в глобальном выравнивании, но странно, что оно не представлено и на карте локального выравнивания.
Интересно также, почему выравнивание:
10 20
PURT_E ALRPAATRVMLLGSGE
::: : . : : :
PURT_R ALRVPGTDLRLFGKPE
360 370
не представлено на карте локальных сходств.
Вышеперечисленные выравнивания обладают хорошим весом (порядка 24-29).
Если поменять параметры dotmatcher и построить новую карту локального сходства (windowsize=6 а не стандартные 10), на ней все равно не будет этого участка: