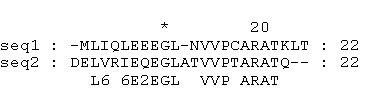
Программа BlastP http://www.ncbi.nlm.nih.gov/blast/
Код доступа белка PURT_ECOLI - P33221
Поиск гипотетических гомологов изучаемого белка PURT_ECOLI в разных БД:
| Поиск по БД Swiss-Prot | Поиск по БД PDB | Поиск по БД "nr" | |
| Лучшая находка | |||
| Идентификатор БД | PURT_ECOLI(P33221) (соответсвует заданному белку) | 1EYZ Chain A (кластеризован с 1EYZ Chain B, 1EZ1 Chain A, 1EZ1 Chain B ) (белок гомодимер - цепи A и B одинаковы) (соответсвует заданному белку) | NP_416363.1 (кластеризован с еще несколькими записями) (соответсвует заданному белку) |
| E-value | 0.0 | 0.0 | 0.0 |
| Вес (в битах) | 796 | 796 | 796 |
| % идентичности | 100% | 100% | 100% |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? | Найдено еще 12 белков с значением E-value=0.0, но выравнивания с ними обладают меньшим весом (например PURT_SHIBS(Q322G6.1) (вес=791, %идентичности=99%)-белок из родственного Escherichia coli организма Shigella boydii, относящегося к тому же семейству энтеробактерий) | Найден еще 1 белок с E-value=0.0, но вес выравнивания меньше=793, процент идентичности=100%, это тот же белок из того же организма, но 1 а.о. - N-формилметионин отщеплена, что часто бывает после трансляции. 1KJ8 Chain A , вместе с ним кластеризованы еще несколько структур с различными лигандами: Mg, АТФ, негидролизуемыми аналогами АТФ, GAR - субстратом. | Найдено еще 50 белков с E-value=0.0, но вес выравнивания меньше для всех |
| Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний, Descriptions, с E-value < 1E-10) | 190 | 6 | 1203 |
| "Худшая" находка (последняя в выдаче с E-value < 1) | |||
| Номер находки в списке описаний (Descriptions) | 406 | 14 | 2522 |
| Идентификатор БД | ACCC_BACHD(Q9KDS9.1) | 2VQD Chain A , с ним кластеризованы еще 2 структуры | P29753.1|VANC_ENTGA |
| E-value | 0.98 | 0.21 | 0.99 |
| Вес (в битах) | 34.7 | 32.7 | 38.5 |
| % идентичности | 22% | 23% | 27% |
| % сходства | 38% | 39% | 41% |
| Длина выравнивания | 323 | 203 | 221 |
| Координаты выравнивания (номера первых и последних а.о. белка query (заданный белок - запрос); затем белка sbjct (сравниваемый белок)) | 14, 311; 4, 322 | 14, 194; 4, 198 | 94, 288; 109, 312 |
| % гэпов | 8% | 14% | 19% |
Белок PURT_ECOLI удалось найти во всех 3 БД. Параметры выравнивания изучаемого белка с самим собой при поиске по разным БД не отличаются.
При поиске в некурируемой безызбыточной БД ("nr") найдено больше всего потенциальных гомологов (1203) из-за того, что эта БД самая большая, меньше потенциальных гомологов (190) найдено в курируемой базе данных Swiss-Prot, где содержатся меньше записей, ведь аннотирование последовательностей белков - процесс трудоемкий. Меньше всего потенциальных гомологов найдено в базе данных пространственных структур белков PDB, она самая маленькая, поскольку для получения третичной структуры белка необходимо его выделить, закристаллизовать и провести рентгеноструктурный анализ, что еще сложнее. Записи PDB на сайте NCBI, как правило, кластеризованы со структурами тех же белков, но с другими лигандами.
"Худшие" гомологи белка различны. Лучший гомолог найден в базе данных PDB (самое низкое E-value=0.21), однако база данных PDB - самая маленькая, а E-value прямо пропорционален размеру базы данных, поэтому возможно лучший гомолог найден в базе данных "nr", ведь E-value находки больше в 5 раз, в то время как размр базы данных "nr" больше PDB в большее число раз. В пользу этого говорит и то, что %идентичности и %сходства находки из "nr" выше.
Поиск гипотетических гомологов белка PURT_ECOLI в организмах таксона филогенетически далекого от E. coli:
| Поиск по БД Swiss-Prot | Поиск по БД "nr" | |
| Идентификатор БД | P31327.2|CPSM_HUMAN | dbj|BAD92974.1 |
| E-value | 4e-04 | 1e-04 |
| Вес (в битах) | 42.7 | 47.0 |
| % идентичности | 20% | 24% |
| % сходства | 44% | 44% |
| Длина выравнивания | 207 | 188 |
| Координаты выравнивания (номера первых и последних а.о. белка query (заданный белок - запрос); затем белка sbjct (сравниваемый белок)) | 86, 283; 1060, 1259 | 13, 183; 66, 253 |
| % гэпов | 7% | 9% |
| Идентификатор БД | Q96RQ3.3|MCCA_HUMAN | dbj|BAA14328.1 |
| E-value | 0.001 | 8e-04 |
| Вес (в битах) | 41.2 | 44.3 |
| % идентичности | 21% | 20% |
| % сходства | 44% | 46% |
| Длина выравнивания | 187 | 206 |
| Координаты выравнивания (номера первых и последних а.о. белка query (заданный белок - запрос); затем белка sbjct (сравниваемый белок)) | 13, 186; 50, 236 | 86, 283; 1060, 1259 |
| % гэпов | 13% | 6% |
Найденные гомологи - митохондриальные белки человека, их близость к бактериальному белку согласуется с теорией симбиогенеза митохондрий.
В базе данных PDB гомологи с E-value <=0.001 не найдены.
Как видно из выравниваний белок P31327.2|CPSM_HUMAN и dbj|BAA14328.1 очень похожи, посмотрим на их выравнивание: E-value=0.0, вес (в битах) = 3069, % идентичности = 98%, % сходства = 99%, длина выравнивания = 1500, % гэпов = 0%
Заметим также, что E-value выравниваний этих белков с заданным белком PURT_ECOLI отличаются в 2 раза (в "nr" больше), а поскольку белки с которыми сравнивается PURT_ECOLI отличаются незначительно, и E-value прямо пропорционален размеру базы данных, база данных "nr" больше Swiss-Prot в 2 раза.
| Поиск по БД Swiss-Prot | Поиск по БД PDB | Поиск по БД "nr" | |
| Идентификатор БД | Q58881.2|PURT_METJA | pdb|2CZG|A , с ним кластеризованы еще несколько записей структур белков с разными лигандами | NP_248492.1 |
| E-value | 1e-125 | 2e-100 | 9e-125 |
| Вес (в битах) | 444 | 357 | 444 |
| % идентичности | 57% | 49% | 57% |
| % сходства | 72% | 63% | 72% |
| Длина выравнивания | 389 | 404 | 389 |
| Координаты выравнивания (номера первых и последних а.о., сначала координаты белка PURT_ECOLI, затем найденного белка) | 4, 303; 3, 302 | 4, 391; 11, 414 | 4, 391; 7, 393 |
| % гэпов | примерно 0% | 3% | примерно 0% |
| Поиск по БД Swiss-Prot | Поиск по БД "nr" | |
| Идентификатор БД | sp|A4QHG1.1|PURT_CORGB , сним кластеризован белок sp|Q8NM23.2|PURT_CORGL из близкого организма | ref|YP_002779151.1 , с ним кластеризован белок dbj|BAH50206.1 из родственного организма |
| E-value | 1e-125 | 1e-125 |
| Вес (в битах) | 444 | 449 |
| % идентичности | 57% | 60% |
| % сходства | 71% | 72% |
| Длина выравнивания | 396 | 393 |
| Координаты выравнивания (номера первых и последних а.о., сначала координаты белка PURT_ECOLI, затем найденного белка) | 4, 391; 7, 401 | 4, 389; 9, 400 |
| % гэпов | 2% | 2% |
Поиск по таксону Actinobacteria (taxid:201174) в базе данных PDB не дал результатов (для E-vakue<=0.001), что и понятно, ведь размер базы данных PDB меньше, и вероятность встретить там гомолога белку - меньше.
| Поиск по БД Swiss-Prot | Поиск по БД "nr" | |
| Идентификатор БД | sp|Q0HS20.1|PURT_SHESR | ref|ZP_01898754.1 |
| E-value | 7e-157 | 4e-161 |
| Вес (в битах) | 547 | 565 |
| % идентичности | 67% | 70% |
| % сходства | 81% | 83% |
| Длина выравнивания | 389 | 390 |
| Координаты выравнивания (номера первых и последних а.о., сначала координаты белка PURT_ECOLI, затем найденного белка) | 3, 390; 1, 389 | 3, 390; 1, 390 |
| % гэпов | примерно 0% | примерно 0% |
В базе данных PDB гомологов опять не найдено.
| Поиск по БД Swiss-Prot | Поиск по БД "nr" | |
| Идентификатор БД | sp|Q6LRI4.1|PURT_PHOPR | ref|ZP_01223027.1 |
| E-value | 8e-164 | 2e-163 |
| Вес (в битах) | 570 | 573 |
| % идентичности | 70% | 70% |
| % сходства | 82% | 83% |
| Длина выравнивания | 389 | 389 |
| Координаты выравнивания (номера первых и последних а.о., сначала координаты белка PURT_ECOLI, затем найденного белка) | 3, 390; 1, 389 | 3, 390; 1, 389 |
| % гэпов | примерно 0% | примерно 0% |
Поиск гомологв по базе данных PDB не дал результата.
Белок широко распространен по разным таксонам, не является специфичным для E.coli.
Поиск белка по его фрагменту в Swiss-Prot:
Ищем белок по фрагменту DELVRIEQEGLATVVPTARATQ
| Поиск по фрагменту | Поиск по полной последовательности |
|
| АС лучшей находки | Q8FMB3.2|PURT_COREF | Q8FMB3.2|PURT_COREF - тот же белок |
| E-value | 5e-13 | 0.0 |
| Вес (в битах) | 71.0 | 816 |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? |
нет, у остальных найденных последовательностей - вес меньше, а E-value больше | Найден белок A4QHG1.1|PURT_CORGB с таким же значением E-value, но с меньшим весом (730) |
Последовательность найденного белка в фаста-формате
Так как фрагмент белка короче его полной последовательности, вероятность найти фрагмент выше, поэтому в поиске по фрагменту E-value больше, по той же причине вес меньше - ведь количество сопоставленных аминокислотных остатков больше у выравнивания белка целиком, для фрагмента белка найдено небольшое число белков (8) с низким E-value (<=0.003, хотя это значение достаточно большое), для полного же белка их очень много (больше 250), что тоже вызвано большой вероятностью найти малое количество букв случайно.
Выравнивание найденного белка с белком PURT_ECOLI: E-Value = 3e-122, вес выравнивания = 438, %идентичности = 57%, %сходства = 70%, длина выравнивания = 397, координаты выравнивания (сначала найденный белок): 7, 402; 4, 392, %гэпов = 2%, интересно, что в одном кластере с изучаемым белком PURT_ECOLI идет белок Q3Z2K0.1|PURT_SHISS из родственного Escherichia coli организма Shigella sonnei (они относятся к одному семейству энтеробактерий).
Query - найденный белок PURT_COREF, Sbjct - изучаемый белок PURT_ECOLI
Query 7 IGTPLSPNATKVMILGSGELGKEVTIAFQRLGVEVHAVDRYDNAPAHQVAHFSYVIDMTD 66
+GT L P AT+VM+LGSGELGKEV I QRLGVEV AVDRY +APA VAH S+VI+M D
Sbjct 4 LGTALRPAATRVMLLGSGELGKEVAIECQRLGVEVIAVDRYADAPAMHVAHRSHVINMLD 63
Query 67 AAAVRELVTTIKPDFIIPEIEALATDELVRIEQEGLATVVPTARATQLTMNREGIRRLAS 126
A+R +V KP +I+PEIEA+ATD L+++E+EGL VVP ARAT+LTMNREGIRRLA+
Sbjct 64 GDALRRVVELEKPHYIVPEIEAIATDMLIQLEEEGL-NVVPCARATKLTMNREGIRRLAA 122
Query 127 EELGLPTSGYEFCSTFEEFTAAAERLGYPNVVKPVMSSSGKGQSVVTSAEDLESAWEYAM 186
EEL LPTS Y F + F A +GYP +VKPVMSSSGKGQ+ + SAE L AW+YA
Sbjct 123 EELQLPTSTYRFADSESLFREAVADIGYPCIVKPVMSSSGKGQTFIRSAEQLAQAWKYAQ 182
Query 187 SGARVSNQRVIVEQFVEFDYEITLLTVRGIDPATGKPATWFCEPIGHRQQDGDYVESWQP 246
G R RVIVE V+FD+EITLLTV +D FC P+GHRQ+DGDY ESWQP
Sbjct 183 QGGRAGAGRVIVEGVVKFDFEITLLTVSAVD------GVHFCAPVGHRQEDGDYRESWQP 236
Query 247 MEMTAPALENARSVAARITNALGGRGVFGVELFVSGDDVYFSEVSPRPHDTGLVTLATQR 306
+M+ ALE A+ +A ++ ALGG G+FGVELFV GD+V FSEVSPRPHDTG+VTL +Q
Sbjct 237 QQMSPLALERAQEIARKVVLALGGYGLFGVELFVCGDEVIFSEVSPRPHDTGMVTLISQD 296
Query 307 FSEFELHAKAVLGLPV-DVTLTSPGASAVIYGGVDSPGVSYAGLAEALAVAETDVRLFGK 365
SEF LH +A LGLPV + P ASAVI + S V++ + A+ A+ +RLFGK
Sbjct 297 LSEFALHVRAFLGLPVGGIRQYGPAASAVILPQLTSQNVTFDNVQNAVG-ADLQIRLFGK 355
Query 366 PEAFTKRRMGVAVSTAEDTATARDRATLAAAAVTVHG 402
PE RR+GVA++TAE A +RA AA V V G
Sbjct 356 PEIDGSRRLGVALATAESVVDAIERAKHAAGQVKVQG 392
При выравнивании вручную фрагмента белка PURT_ECOLI MLIQLEEEGLNVVPCARATKLT с фрагментом белка PURT_COREF DELVRIEQEGLATVVPTARATQ получено:
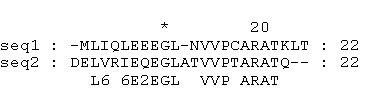
| фрагмент белка PURT_ECOLI: 90-111 а.о (seq1), фрагмент белка PURT_COREF: 92-113 а.о (seq2). |
оба выравнивания совпадают: есть только 1 внутренний гэп - на против а.о A белка PURT_COREF (номер а.о 113), этот гэп между а.о 99(L) и 100(N) белка PURT_ECOLI.
Сравнение выравниваний, выданных программой BLASTP, с оптимальными глобальным и локальным выравниваниями:
Выравнивание, полученное программой BLASTP, с штрафами за гэп = 11 и за удлинение гэпа = 1 (qyery - белок PURT_COREF, sbjct - белок PURT_ECOLI) :
Query 7 IGTPLSPNATKVMILGSGELGKEVTIAFQRLGVEVHAVDRYDNAPAHQVAHFSYVIDMTD 66
+GT L P AT+VM+LGSGELGKEV I QRLGVEV AVDRY +APA VAH S+VI+M D
Sbjct 4 LGTALRPAATRVMLLGSGELGKEVAIECQRLGVEVIAVDRYADAPAMHVAHRSHVINMLD 63
Query 67 AAAVRELVTTIKPDFIIPEIEALATDELVRIEQEGLATVVPTARATQLTMNREGIRRLAS 126
A+R +V KP +I+PEIEA+ATD L+++E+EGL VVP ARAT+LTMNREGIRRLA+
Sbjct 64 GDALRRVVELEKPHYIVPEIEAIATDMLIQLEEEGL-NVVPCARATKLTMNREGIRRLAA 122
Query 127 EELGLPTSGYEFCSTFEEFTAAAERLGYPNVVKPVMSSSGKGQSVVTSAEDLESAWEYAM 186
EEL LPTS Y F + F A +GYP +VKPVMSSSGKGQ+ + SAE L AW+YA
Sbjct 123 EELQLPTSTYRFADSESLFREAVADIGYPCIVKPVMSSSGKGQTFIRSAEQLAQAWKYAQ 182
Query 187 SGARVSNQRVIVEQFVEFDYEITLLTVRGIDPATGKPATWFCEPIGHRQQDGDYVESWQP 246
G R RVIVE V+FD+EITLLTV +D FC P+GHRQ+DGDY ESWQP
Sbjct 183 QGGRAGAGRVIVEGVVKFDFEITLLTVSAVD------GVHFCAPVGHRQEDGDYRESWQP 236
Query 247 MEMTAPALENARSVAARITNALGGRGVFGVELFVSGDDVYFSEVSPRPHDTGLVTLATQR 306
+M+ ALE A+ +A ++ ALGG G+FGVELFV GD+V FSEVSPRPHDTG+VTL +Q
Sbjct 237 QQMSPLALERAQEIARKVVLALGGYGLFGVELFVCGDEVIFSEVSPRPHDTGMVTLISQD 296
Query 307 FSEFELHAKAVLGLPV-DVTLTSPGASAVIYGGVDSPGVSYAGLAEALAVAETDVRLFGK 365
SEF LH +A LGLPV + P ASAVI + S V++ + A+ A+ +RLFGK
Sbjct 297 LSEFALHVRAFLGLPVGGIRQYGPAASAVILPQLTSQNVTFDNVQNAVG-ADLQIRLFGK 355
Query 366 PEAFTKRRMGVAVSTAEDTATARDRATLAAAAVTVHG 402
PE RR+GVA++TAE A +RA AA V V G
Sbjct 356 PEIDGSRRLGVALATAESVVDAIERAKHAAGQVKVQG 392
Оптимальное глобальное выравнивание ( в msf-формате ) , полученное программой needle пакета EMBOSS, с теми же штрафами:
PURT_COREF 1 MFIPEKIGTPLSPNATKVMILGSGELGKEVTIAFQRLGVEVHAVDRYDNA 50
...:||.|.|.||:||:||||||||||.|..|||||||.|||||.:|
PURT_ECOLI 1 ---MTLLGTALRPAATRVMLLGSGELGKEVAIECQRLGVEVIAVDRYADA 47
PURT_COREF 51 PAHQVAHFSYVIDMTDAAAVRELVTTIKPDFIIPEIEALATDELVRIEQE 100
||..|||.|:||:|.|..|:|.:|...||.:|:|||||:|||.|:::|:|
PURT_ECOLI 48 PAMHVAHRSHVINMLDGDALRRVVELEKPHYIVPEIEAIATDMLIQLEEE 97
PURT_COREF 101 GLATVVPTARATQLTMNREGIRRLASEELGLPTSGYEFCSTFEEFTAAAE 150
|| .|||.||||:||||||||||||:|||.||||.|.|..:...|..|..
PURT_ECOLI 98 GL-NVVPCARATKLTMNREGIRRLAAEELQLPTSTYRFADSESLFREAVA 146
PURT_COREF 151 RLGYPNVVKPVMSSSGKGQSVVTSAEDLESAWEYAMSGARVSNQRVIVEQ 200
.:|||.:||||||||||||:.:.|||.|..||:||..|.|....|||||.
PURT_ECOLI 147 DIGYPCIVKPVMSSSGKGQTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEG 196
PURT_COREF 201 FVEFDYEITLLTVRGIDPATGKPATWFCEPIGHRQQDGDYVESWQPMEMT 250
.|:||:|||||||..:| ...||.|:||||:||||.|||||.:|:
PURT_ECOLI 197 VVKFDFEITLLTVSAVD------GVHFCAPVGHRQEDGDYRESWQPQQMS 240
PURT_COREF 251 APALENARSVAARITNALGGRGVFGVELFVSGDDVYFSEVSPRPHDTGLV 300
..|||.|:.:|.::..||||.|:|||||||.||:|.||||||||||||:|
PURT_ECOLI 241 PLALERAQEIARKVVLALGGYGLFGVELFVCGDEVIFSEVSPRPHDTGMV 290
PURT_COREF 301 TLATQRFSEFELHAKAVLGLPV-DVTLTSPGASAVIYGGVDSPGVSYAGL 349
||.:|..|||.||.:|.||||| .:....|.|||||...:.|..|::..:
PURT_ECOLI 291 TLISQDLSEFALHVRAFLGLPVGGIRQYGPAASAVILPQLTSQNVTFDNV 340
PURT_COREF 350 AEALAVAETDVRLFGKPEAFTKRRMGVAVSTAEDTATARDRATLAAAAVT 399
..|:. |:..:|||||||....||:|||::|||....|.:||..||..|.
PURT_ECOLI 341 QNAVG-ADLQIRLFGKPEIDGSRRLGVALATAESVVDAIERAKHAAGQVK 389
PURT_COREF 400 VHG 402
|.|
PURT_ECOLI 390 VQG 392
Оптимальное локальное выравнивание ( в msf-формате ) , полученное с помощью программы water пакета EMBOSS, с теми же штрафаи:
PURT_COREF 7 IGTPLSPNATKVMILGSGELGKEVTIAFQRLGVEVHAVDRYDNAPAHQVA 56
:||.|.|.||:||:||||||||||.|..|||||||.|||||.:|||..||
PURT_ECOLI 4 LGTALRPAATRVMLLGSGELGKEVAIECQRLGVEVIAVDRYADAPAMHVA 53
PURT_COREF 57 HFSYVIDMTDAAAVRELVTTIKPDFIIPEIEALATDELVRIEQEGLATVV 106
|.|:||:|.|..|:|.:|...||.:|:|||||:|||.|:::|:||| .||
PURT_ECOLI 54 HRSHVINMLDGDALRRVVELEKPHYIVPEIEAIATDMLIQLEEEGL-NVV 102
PURT_COREF 107 PTARATQLTMNREGIRRLASEELGLPTSGYEFCSTFEEFTAAAERLGYPN 156
|.||||:||||||||||||:|||.||||.|.|..:...|..|...:|||.
PURT_ECOLI 103 PCARATKLTMNREGIRRLAAEELQLPTSTYRFADSESLFREAVADIGYPC 152
PURT_COREF 157 VVKPVMSSSGKGQSVVTSAEDLESAWEYAMSGARVSNQRVIVEQFVEFDY 206
:||||||||||||:.:.|||.|..||:||..|.|....|||||..|:||:
PURT_ECOLI 153 IVKPVMSSSGKGQTFIRSAEQLAQAWKYAQQGGRAGAGRVIVEGVVKFDF 202
PURT_COREF 207 EITLLTVRGIDPATGKPATWFCEPIGHRQQDGDYVESWQPMEMTAPALEN 256
|||||||..:| ...||.|:||||:||||.|||||.:|:..|||.
PURT_ECOLI 203 EITLLTVSAVD------GVHFCAPVGHRQEDGDYRESWQPQQMSPLALER 246
PURT_COREF 257 ARSVAARITNALGGRGVFGVELFVSGDDVYFSEVSPRPHDTGLVTLATQR 306
|:.:|.::..||||.|:|||||||.||:|.||||||||||||:|||.:|.
PURT_ECOLI 247 AQEIARKVVLALGGYGLFGVELFVCGDEVIFSEVSPRPHDTGMVTLISQD 296
PURT_COREF 307 FSEFELHAKAVLGLPV-DVTLTSPGASAVIYGGVDSPGVSYAGLAEALAV 355
.|||.||.:|.||||| .:....|.|||||...:.|..|::..:..|:.
PURT_ECOLI 297 LSEFALHVRAFLGLPVGGIRQYGPAASAVILPQLTSQNVTFDNVQNAVG- 345
PURT_COREF 356 AETDVRLFGKPEAFTKRRMGVAVSTAEDTATARDRATLAAAAVTVHG 402
|:..:|||||||....||:|||::|||....|.:||..||..|.|.|
PURT_ECOLI 346 ADLQIRLFGKPEIDGSRRLGVALATAESVVDAIERAKHAAGQVKVQG 392
| выравнивание BLASTP | выравнивание needle | выравнивание water | |
| вес выравнивания | 1126 | 1082 | 1087 |
| %идентичности | 57% | 56.8% | 57.7% |
| %сходства | 70% | 69.5% | 70.5% |
| длина выравнивания | 397 | 403 | 397 |
| координаты выравнивания (вначале номера а.о белка PURT_COREF, затем PURT_ECOLI) | 7, 402; 4, 392 | 1, 402; 1, 392 | 7, 402; 4, 392 |
| %гэпов | 2% | 3% | 2.3% |
Выравнивание BLASTP имеет больше сходств с локальным выравниванием, возможно потому что само строиться по принципу локального выравнивания.
Во всех трех выравниваниях есть гэп в последовательности белка PURT_ECOLI между 213 аминокислотой (D) и 214 аминокислотой (G), протяженность гэпа - 6, в последовательности белка PURT_COREF гэпу соответствуют а.о с 218 (P) по 223 (P).
Выравнивания очень похожи, так глобальное выравнивание, локальное выравнивание и выравнивание BLASTP полностью совпадают после этого гэпа (с 214 а.о (G) по 392 а.о (G) белка PURt_ECOLI, с 224 а.о (A) по 402 а.о (G) белка PURT_COREF).
Выравнивание BLASTP и локальное выравниван ие отличается от глобального, тем, что они начинаются не с начала последовательностей а с 4 аминокислоты белка PURT_ECOLI, с 7 аминокислоты белка PURT_COREF, локальное выравнивание вообще совпадает с выравнивание BLASTP, но они имеет разный вес. Странно, ведь для подсчета используется одна и та же матрица замен аминокислот BLOSUM 62, но на некоторых позициях выравнивания есть различая начисления веса при одних и тех же а.о: так положительная замена в выравнивании BLASTP обозначается +, а в выравнивании water пишется ее вес, но на против позиции 197 белка PURT_COREF (E) - 196 белка PURT_ECOLI (K) стоит плюс в выравнивании BLASTP, но ничего нет в water, также в позициях 90 PURT_COREF - 90 PURT_ECOLI, таких примеров еще несколько.
О гомологах белка PURT_ECOLI и истории открытия:
До открытия белка PURT , был известен другой фермент, участвующий в биосинтезе пуринов - PURN , также присоединяющий остаток муравьиной кислоты к GAR (GAR - фосфорибозилглицинамид), однако он присоединяет, его не на прямую, как это делает PURT, а переносит с соединения формата с фолиевой кислотой. Несмотря на сходство реакции - формилирование GAR, не выявлено гомологии между PURT и PURN белками. PURT гомологичен белку PURK , также участвующему в биосинтезе пуринов. Что необычно на первый взгляд, ведь PURK - фермент карбоксилаза, а не трансфераза как PURT. (на основании статьи Cloning and characterization of a new purine biosynthetic enzyme: a non-folate glycinamide ribonucleotide transformylase from E. coli. [PMID: 8117714] )
И действительно, при выравнивании PURT_ECOLI и PURN_ECOLI в программ BLASTP - не выявлено гомологии (No significant similarity found)
при выравнивании же PURT_ECOLI и PURK_ECOLI выявлен общий домен, соответствующий 123-312 а.о белка PURT и 88-271 а.о белка PURK (E-value=8e-16, %идентичности=27%, %сходства=46%, длина выравнивания=192, %гэпов=5% - при стандартных штрафах за гэпы)
выравнивание согласуется с данными базы данных UniProt о белке PURT (p33221) и белке PURK (p09029) :
белок PURT содержит АТФ-связывающий домен (119-308 а.о), АТФ-связывающий домен одержит также белок PURK (84-267 а.о), белки принадлежат к АТФ-связывающему суперсемейству.
наглядно можно увидеть схожие области на карте локального выравнивания: