Ознакомление с программой Muscle:
Для множественного выравнивания возьмем вирусные белки - дельта-антигены ( файл с последовательностями в fasta-формате )
В файлах с выравниванием белки даны в другой последовательности; в начальном файле тоже были консервативные колоники, однако в оптимальном выравнивании консервативные колоники не всегда совпадают с ними.
Выравнивание набора гомологов белка PURT_ECOLI (P33221):
Для выравнивания найдем BLAST'ом 10 гомологов белка PURT_ECOLI (считаем гомолог достоверным, если E-value<0.001), из них берем те, для которых процент идентичности с белком PURT_ECOLI < 90% - чтобы белки были не слишком похожи и выравнивание было интереснее. Чтобы гомологи не были слишком близки друг к другу, выбираем их из разных родов организмов.
| Идентификатор | E-value | %идентичности | таксономия организма |
| >sp|Q57NB7.1|PURT_SALCH | 0.0 | 87% | Salmonella choleraesuis. Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella |
| >sp|A7MEA0.2|PURT_ENTS8 | 0.0 | 82% | Enterobacter sakazakii (strain ATCC BAA-894). Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Cronobacter. |
| >sp|Q1C8V8.1|PURT_YERPA | 0.0 | 78% | Yersinia pestis bv. Antiqua (strain Antiqua). Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia. |
| >sp|Q6D4B6.1|PURT_ERWCT | 7e-175 | 78% | Erwinia carotovora subsp. atroseptica (Pectobacterium atrosepticum). Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Pectobacterium. |
| >sp|Q2NTH8.1|PURT_SODGM | 1e-174 | 75% | Sodalis glossinidius (strain morsitans). Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Sodalis. |
| >sp|Q6LRI4.1|PURT_PHOPR | 6e-162 | 70% | Photobacterium profundum (Photobacterium sp. (strain SS9)). Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Photobacterium. |
| >sp|Q9KSM8.2|PURT_VIBCH | 8e-161 | 71% | Vibrio cholerae. Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Vibrio. |
| >sp|Q0HS20.1|PURT_SHESR | 3e-155 | 67% | Shewanella sp. (strain MR-7). Bacteria; Proteobacteria; Gammaproteobacteria; Alteromonadales; Shewanellaceae; Shewanella. |
| >sp|Q1I5Y3.1|PURT_PSEE4 | 9e-155 | 69% | Pseudomonas entomophila (strain L48). Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas. |
| >sp|A0KK42.1|PURT_AERHH | 3e-151 | 68% | Aeromonas hydrophila subsp. hydrophila (strain ATCC 7966 / NCIB 9240). Bacteria; Proteobacteria; Gammaproteobacteria; Aeromonadales; Aeromonadaceae; Aeromonas. |
По низким значениям E-value видно, что выбранные белки - достоверные гомологи для PURT_ECOLI, файл с последовательностями гомологов в fasta-формате
Файл с выравниванием в msf-формате
Файл с выравниванием в fasta-формате
| координаты по столбцам выравнивания | координаты по остаткам белка PURT_ECOLI |
| 21-64 | 21-64 |
| 110-130 | 110-130 |
| 220-237 | 220-237 |
| 257-270 | 257-270 |
| 277-312 | 277-312 |
Интересно, что по данным UniProt белок PURT_ECOLI ( p33221 ) содержит АТФ-связывающий домен (119-308 а.о), и наиболее длинные консервативные участки находятся в этой области. Сравниваемые гомологи также имеют АТФ-связывающий домен (такого же размера, расположенный в схожей области), выравнивание подтвердило это, белки относятся к АТФ-связывающему суперсемейству. PURT_ECOLI - магний-связывающий белок, металл связывается с аминокислотам E в позициях 267 и 279, отсальные белки также связывают магний (Q57NB7, A7MEA0, Q1C8V8, Q6D4B6, Q2NTH8, Q1I5Y3 - с теми же аминокислотам в тех же позициях, Q6LRI4, Q9KSM8, Q0HS20, A0KK42 - с теми же аминокислотам, но в позициях 265, 277), выравнивание выявило это, перечисленные аминокислоты попарно находятся в одних и тех же колонках. Любопытно, что в гомологах, где магний-связывающие аминокислоты сдвинуты на 2 позиции, на 2 позиции и в ту же сторону сдвинуты также и АТФ-связывающие домены (у белка PURT_ECOLI, Q57NB7, A7MEA0, Q1C8V8, Q6D4B6, Q2NTH8, Q1I5Y3: 119-308, Q6LRI4, Q9KSM8, Q0HS20, A0KK42: 117-306), возможно в белках со сдвигом домена произошла делеция 2 аминокислот (во время эволюционного процесса формирования белка) (или наоборот у других белков вставка 2 аминокислот), на выравнивании это отражено двумя гэпами в начале последовательностей со сдвигом домена
| координаты по столбцам выравнивания | координаты по остаткам белка PURT_ECOLI |
| 332-344 | 322-344 |
| 379-391 | 377-390 |
Думаю здесь выравнивание, возможно, не имеет биологического смысла, т.к консервативные столбцы чередуются с колонками с разными а.о (с структурной точки зрения - вряд ли эту частки обладают схожей третичной структурой, с эволюционной точки зрения - мало вероятны мутации идущие триплетно, меняющие аминокислоты через одну, с функциональной точки зрения - такие участки выполняют разные функции, ведь значимы не только отдельные а.о, но и аминокислоты, окружающие их)
Программы mafft и edialign:
Программа mafft:
Для выравнивания возьмем PURT_ECOLI и те же его гомологи: файл с выравниванием в fasta-формате
файл с выравниванием в msf-формате
В файлк с выравниванием - белки в том же порядке, что и в исходном файле с полседовательностями. Выравнивание похоже на выравнивание программой muscle, в последовательностях со сдвигом домена, так же есть двойной гэп в начале последовательности, однако этот гэп стоит не в самом начале, а начиная со второй позиции выравнивания. Таким образом все первые M аминокислоты белков стоят в одной колонке, в программе muscle такого не было, ведь краевые гэпы не штрафуются, mafft же пошел на это, возможно из-за других штрафов за гэпы. С другой стороны выравнивание первых метионинов, думаю, не имеет биологического смысла, ведь все бактериальные белки начинаются с метионина (с N-формилметионина), но часто он отщепляется после трансляции.
Программа edialign:
Для выравнивания возьмем те же белки: файл с выравниванием в fasta-формате
файл с выравниванием в msf-фортмате
файл с выравниванием в dialign-формате
В файле с выравниванием - белки в том же порядке, что и в исходном файле с последовательностями. Выравнивание очень похоже на результат muscle, отличаются они тольео расстановкой гэпов в 345-350 колонках выравнивания.
Обработка множественных выравниваний:
Программа consambig:
Для обработки возьмем выравнивание, сделанное программой muscle: файл с результатом в fasta-формате
Программа позволяет наглядно показать наиболее консервативные позиции выравнивания и аминокислоты, стоящие на этих позициях.
Программа distmat:
Для обработки возьмем выравнивание muscle: результат по алгоритму, неучитывающему повторы (does not make any corrections for multiple substitutions)
результат по алгоритму Jukes-Cantor (дает поправку на повторы)
В результате обработки получаем мтрицу, в столбцах и строках которой - сравниваемые белки, а в ячейках эволюционное расстояние между парами белков (на пересечении строки и столбца), по главной диагонали стоят нули - т.к. это сравнение белка с самим собой, отностельно этой диагонали - матрица симметрична, поэтому можно рассматривать только ее половину (в данном случае верхную)
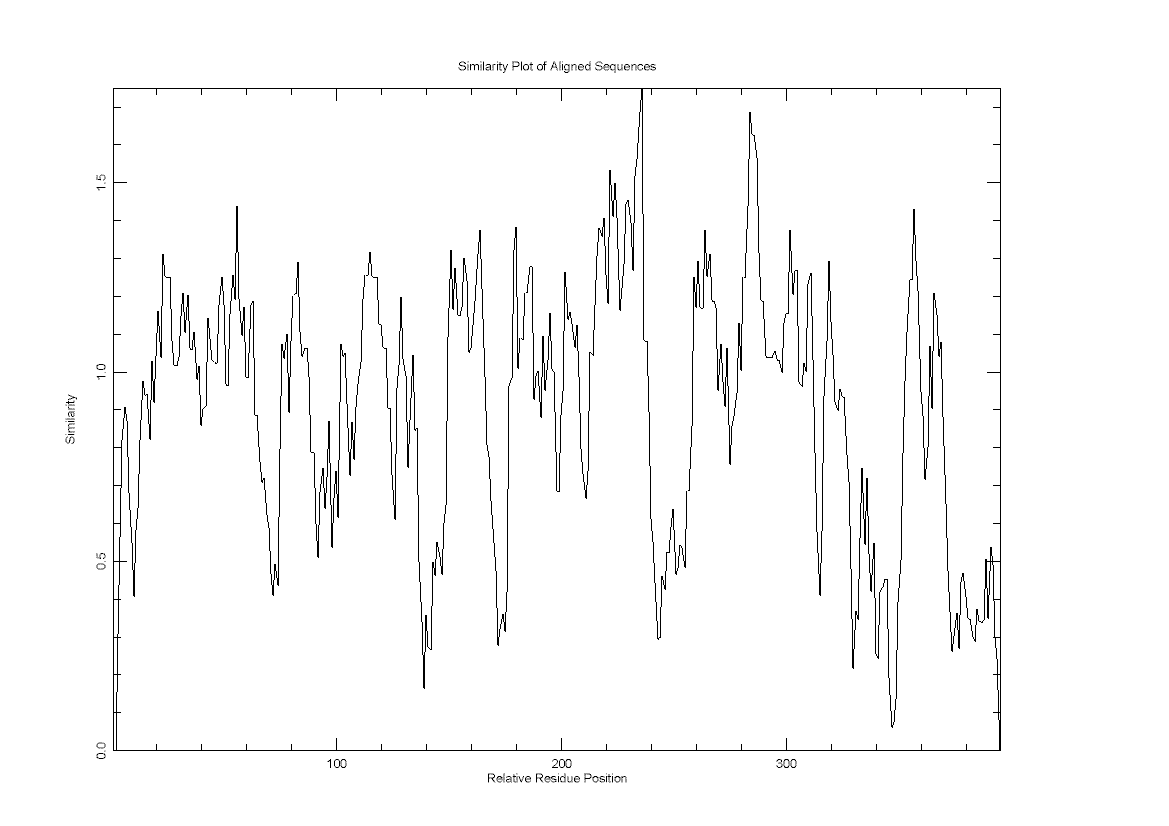
Программа plotcon :
также возьмем выравнивание muscle

график показывает консервативность различных участков выравнивания