Создание паттернов аминокислотных последовательностей:
Паттерн по выравниванию программы muscle:
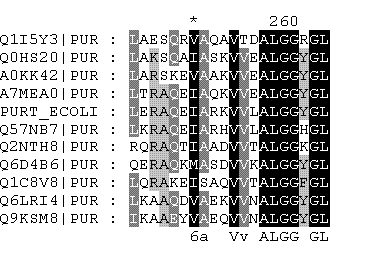
выравнивание белка PURT_ECOLI с гомологами
Возьмем фрагмент выравнивания 244-263 (соответсвует 244-263 а.о белка PURT_ECOLI ( ID=P33221 )):

| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? (поиск в базе данных PROSITE | Все ли последовательности из Вашего выравнивания найдены? |
| Фрагмент последовательности белка PURT_ECOLI (P33221) | L-E-R-A-Q-E-I-A-R-K-V-V-L-A-L-G-G-Y-G-L | в 10 (Q8XCJ9, A1AC10, Q0TGY3, P33221, Q1RAT6, Q322G6, Q32HB3, Q0T3V7, Q83KS0, Q3Z2K0), во всех найденных белках мотив имеет координаты 244-263 а.о | найден только мой белок |
| Сильный | [LIRQ]-[ATEKQ]-[EKRA]-[SA]-[QKE]-[RAETKDY]-[VIM]-[AS]-[QSARE]-[AKHDQ]-V-[VT]-[DELTKN]-A-L-G(2)-[RYHKF]-G-L | в 36 ( открыть файл с результатом ) | найдены все 11 белков из выравнивания |
| Слабый | [LIVARQG]-x-[EDKRAV]-{CDEFHKMNPQRWYG}-[QNKRED]-{WPHCMG}-{PWYDEKQRNFST}-{CDEFHKMNPQRWY}-{WFGPH}-{WYP}-[VAILG]-[VAILGTS]-{PWYHFLIAVG}-[AVLIG]-[LIVA]-[GAV]-[GAV]-{AVILGED}-[VAG]-[LIVAG] | в 69 белках ( открыть файл с результатом ) | найдены все 11 белков из выравниания |
Фрагмент белка - самый сильный паттерн, поскольку все аминокислоты мотива описаны однозначно, поэтому находок по нему меньше, все найденные белки - из близких организмов, принадлежащих семейству энетеребоктерий. Все белки выполняют одну и ту же функцию ( формат-зависимые-GARтрансформилазы).
В каждую позицию сильного паттерна включены соответсвующие аминокислоты белков выравнивания, поэтому все белки выравнивания находятся в поиске по нему, кроме них найдены белки, выполняющие те же функции в организмах гамма-протеобактерий (найдены белки семйства PURT из разных штаммов организмов, представленных в выравнивании).
При составлении слабого паттерна, область аминокислот, подоходящих для каждой позиции паттерна расширялась: например к алифатическим аминокислотам A, L добавлялись V, I; к заряженным D, E, K додобавляю R, гидрофильные N, Q можно и T, S добавить, а вот Y в такой ситуации добавлять не желательно, т.к тирозин - ароматическая аминокислота, а предыдущие нет. Если вариантов было и так много, то разрешаем на этой позиции любую аминокислоту - х. Т.к за основу паттерна брали изначальный самый строгий вариант, то белки выравнивания находятся, но кроме них находится много белков из далеких к энетробактериям организмов, выполняющие разные другие функции.
Белок PURT_ECOLI имеет АТФ-связывающий домен: 119-308 (по данным UniProt P33221 ), выбранный для паттерна участок относится к этому домену, все белки найденные по сильному выравниванию также имеют АТФ-связывающий домен, причем описанный мотив также находится в домене. Среди белков, найденных по слабому выравниванию много белков не из семейства PURT, но некоторые как и PURT_ECOLI также связывают АТФ (например Q9CEB5 , O28994 , для белка A1R6Z3 мотив даже попадает на область АТФ-связывающего домена, вообще найдено несколько белков из семейства CARB - бактериальные белки; найдены белки грибов также связывающие АТФ - Q9HG71 - белок аспергилла). У найденных белков, связывающих АТФ, как и у PURT_ECOLI есть участки связывающие магний (возможно потому что АТФ в клетке существует в комплексе с магнием). 2 из найденных белков хордовых ( O60518 и Q8BIV3 ) - работают рецепторами при транспорте белков в ядро клетки, вряд ли они как-то связаны с белками PURT, скорее всего нашлись случайно.
| Идентификатор документа PROSITE (AC) | Название мотива | Краткое описание мотива | Тип подписи (паттерн, профиль) | Паттерн (регулярное выражение) | Специфична ли подпись? | Сколько мотивов нашлось в белке? | координаты мотива в белке |
| PS50975 | ATP_GRASP | АТФ-связывающий домен | профиль | страница с матрицей | спецефична | 1 | 119 - 308 |
| PS00005 | PKC_PHOSPHO_SITE | сайт фосфорилирования (присоединения фосфата) протеинкиназой C | паттерн | [ST] - x - [RK] [фосфорилируется ОН-группа S или T] | неспецефична | 4 | 131 - 133 161 - 163 281 - 283 361 - 363 |
| PS00008 | MYRISTYL | сайт N-миристилирования (миристилирование - присоединения остатка миристиновой кислоты - насыщенной жирной кислоты (С14)) | паттерн | G - {EDRKHPFYW} - x(2) - [STAGCN] - {P} [миристат присоеденяется к G] | неспецефична | 4 | 184 - 189 214 - 219 309 - 314 365 - 370 |
| PS00006 | CK2_PHOSPHO_SITE | сайт фосфорилирования казеинкиназой 2 | паттерн | [ST] - x(2) - [DE] [фосфорилируется S или T] | неспецефична | 2 | 210 - 213 373 - 376 |
| PS00007 | TYR_PHOSPHO_SITE | сайт фосфорилирования тирозинкиназой | паттерн | [RK] - x(2) - [DE] - x(3) - Y or [RK] - x(3) - [DE] - x(2) - Y [фосфорилируется Y] | неспецефична | 1 | 224 - 230 |
| PS00001 | ASN_GLYCOSYLATION | сайт N-гликозилирования | паттерн | N - {P} - [ST] - {P} [гликозилируется N] (P в фигурных скобках, т.к его присутсвтие в этих позициях ингибирует гликозилирование) | неспецефична | 1 | 334 - 337 |