Описание белка PURT_ECOLI
Белок PURT_ECOLI - один из белков бактерии Escherichia coli (штамм K12) - удобного объекта исследований, чей геном и протеом сейчас полностью известен. Белока был открыт и впервые выделен в 1994 году, белок - фермент, участвующие в метаболизме клетки - биосинтезе пуриновых нуклеотидов, без которых невозможно построение ДНК и РНК. Интересен он и тем, что до 1994 года, в течении 30 лет не был открыт ни один фермент, участвующий в пуриновом синтезе. Иследование его продолжается и сейчас, в февральском выпуске 2009 года журнала Mol Cell Proteomics вышла статья об ацетилирование аминокислоты лизина, как о значаимом механизме регуляции метаболизма клеток организмов, начиная от бактреий и до млекопитающих, среди объектов ислледования был и наш белок.
Полное название белка: фосфорибозилглицинамидформилтрансфераза 2, краткое: GART 2 (GAR - фосфорибозилглицинамид, а T означает трансфераза). Белок из организма Escherichia coli (штамм K12), работает в цитоплазме клетки, закодирован в гене PURT (на главной цепи, длина гена - 1179 азотистых оснований) (имя локуса b1849, JW1838).
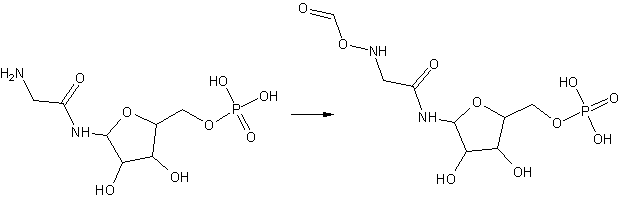
Функции белка: белок - фермент из класса трансфераз, переносит остаток муравьиной кислоты (формилтрансфераза). Код фермента EC=2.1.2.- (к ферментам с кодом 2 - относятся трансферазы, с 2.1 - трансферазы, перносящие одноуглеродные группы). Участвуте в метаболизме пуринов: биосинтез IMP - N(2)-формил-N(1)-(5-фосфо-D-рибозил)глицинамида из N(1)-(5-фосфо-D-рибозил)глицинамидa (путем добавления формата):
Катализируемая реакция: (реакция протекает в 1 стадию)

С работай фермента связана и побочная реакция: получения ацетилфосфата и АДФ из уксусной кислоты и АТФ.
До открытия белка PURT , был известен другой фермент, участвующий в биосинтезе пуринов - PURN , также присоединяющий остаток муравьиной кислоты к GAR, однако он присоеденяет, его не на прямую, как это делает PURT, а переносит с соединения формата с фолиевой кислотой. Несмотря на схожесть реакции - формилирование GAR, не выявлено гомологии между PURT и PURN белками. PURT гомологичен белку PURK , также участвующему в биосинтезе пуринов. Что необычно на первый взгяд, ведь PURK - фермент карбоксилаза, а не трансфераза как PURT. Было бы интересно сделать выравнивание аминокислотных последовательностей этих трех белков для определения гомологичности, но его пока я делать не умею. Общим для для белков PURT и PURK является наличие АТФ-связывающего домена (о нем речь пойдет дальше, в переводе аннотации статьи).
Функция белка, пространственная структура определяется его первичной структурой - последовательностью аминокислот, обратимся к ней.
Аминокислотная последовательность белка и ее особенности:
>sp|P33221|PURT_ECOLI Phosphoribosylglycinamide formyltransferase 2 OS=Escherichia coli (strain K12) GN=purT PE=1 SV=3 MTLLGTALRPAATRVMLLGSGELGKEVAIECQRLGVEVIAVDRYADAPAMHVAHRSHVIN MLDGDALRRVVELEKPHYIVPEIEAIATDMLIQLEEEGLNVVPCARATKLTMNREGIRRL AAEELQLPTSTYRFADSESLFREAVADIGYPCIVKPVMSSSGKGQTFIRSAEQLAQAWKY AQQGGRAGAGRVIVEGVVKFDFEITLLTVSAVDGVHFCAPVGHRQEDGDYRESWQPQQMS PLALERAQEIARKVVLALGGYGLFGVELFVCGDEVIFSEVSPRPHDTGMVTLISQDLSEF ALHVRAFLGLPVGGIRQYGPAASAVILPQLTSQNVTFDNVQNAVGADLQIRLFGKPEIDG SRRLGVALATAESVVDAIERAKHAAGQVKVQG(на основании записи о белке в базе данных UniProt ) Длина последовательности - 392 аминокислотных остатка, молекулярная масса белка - 42434 грамм/моль. Первая аминокислота - N-формилметионин, как это часто бывает у бактерий, отщепляется после трансляции. 179-ый аминокислотный остаток модифицирован - N6-ацетиллизин. Белок - гомодимер (состоит из 2 одинаковых субчастиц).
| Функция | Участок аминокислотной последовательности | Длина участка в аминокислотных остатках |
| АТФ-связывающий домен | 119 - 308 | 190 |
| 5'-фосфорибозилглицинамид-связывающий участок | 22 - 23 | 2 |
| 5'-фосфорибозилглицинамид-связывающий участок | 362 - 363 | 2 |
| Магний-связывающий участок | 267 | 1 |
| Магний-связывающий участок | 279 | 1 |
| 5'-фосфорибозилглицинамид-связывающий участок | 82 | 1 |
| 5'-фосфорибозилглицинамид-связывающий участок | 286 | 1 |
| 5'-фосфорибозилглицинамид-связывающий участок | 355 | 1 |
Статьи про белок:
Аннотация статьи: "Molecular structure of Escherichia coli PurT-encoded glycinamide ribonucleotide transformylase." [PubMed: 10913290]
In Escherichia coli, the PurT-encoded glycinamide ribonucleotide transformylase, or PurT transformylase, catalyzes an alternative formylation of glycinamide ribonucleotide (GAR) in the de novo pathway for purine biosynthesis. On the basis of amino acid sequence analyses, it is known that the PurT transformylase belongs to the ATP-grasp superfamily of proteins. The common theme among members of this superfamily is a catalytic reaction mechanism that requires ATP and proceeds through an acyl phosphate intermediate. All of the enzymes belonging to the ATP-grasp superfamily are composed of three structural motifs, termed the A-, B-, and C-domains, and in each case, the ATP is wedged between the B- and C-domains. Here we describe two high-resolution X-ray crystallographic structures of PurT transformylase from E. coli: one form complexed with the nonhydrolyzable ATP analogue AMPPNP and the second with bound AMPPNP and GAR. The latter structure is of special significance because it represents the first ternary complex to be determined for a member of the ATP-grasp superfamily involved in purine biosynthesis and as such provides new information about the active site region involved in ribonucleotide binding. Specifically in PurT transformylase, the GAR substrate is anchored to the protein via Glu 82, Asp 286, Lys 355, Arg 362, and Arg 363. Key amino acid side chains involved in binding the AMPPNP to the enzyme include Arg 114, Lys 155, Glu 195, Glu 203, and Glu 267. Strikingly, the amino group of GAR that is formylated during the reaction lies at 2.8 A from one of the gamma-phosphoryl oxygens of the AMPPNP.
Перевод аннотации:
В организме Escherichia coli белок PurT трансформилаза (или глицинамидрибонуклеотидтрансформилаза, закодированная в гене PurT) катализирует альтернативную реакцию переноса остатка муравьиной кислоты на глицинамидрибонуклеотид (GAR) в процессе биосинтеза пуринов de novo (из простых предшественников, а не из сложных метаболитов). На основании анализа аминокислотной последовательности известно, что PurT трансформилаза принадлежит к АТФ-связывающему суперсемейству белков. Белки принадлежащие к этому суперсемейсиву катализируют реакции, идущии с затратом АТФ и образованием, в качестве промежуточного интермедиата, ацетилфосфата. Ферменты, принадлежащие к АТФ-связывающему суперсемейству содержат 3 характерных участка аминокислотной последовательности: A-, B-, и C-домены, АТФ закрепляется между B- и C-доменами. В статье обсуждаются структуры высокого разрешения PurT трансформилазы, полученные методом рентгеноструктурного анализа: структура белка в комплексе с негидролизуемым аналогом ATP - AMPPNP и структура в комплексе с AMPPNP и GAR. Последняя особенно важна, поскольку это одна из первых структур, определенных для белка АТФ-связывающего суперсемейства, задействованного в биосинтезе пуринов, в результате дающая новую информацию об участке рибонуклеотидного связывания. GAR субстрат специфически связывается с PurT трансформилазой через взаимодействие с Glu 82, Asp 286, Lys 355, Arg 362, и Arg 363. Среди аминокислотных остатков ключевых для связывания AMPPNP - Arg 114, Lys 155, Glu 195, Glu 203, и Glu 267. Удивительно, что аминогруппа GAR, формилируемая в ходе реакции, расположена на расстояние 2.8 Å от одного из кислородов AMPPNP, связанных с фосфором.
Проверим действительно ли аминокислотные остатки, о которых упомянуто в аннотации, связываются с AMPPNP и GAR???
| Аминокислотные остатки Arg 114, Lys 155, Glu 195, Glu 203, и Glu 267, контактирующие с AMPPNP (1EYZ в RasMol) (аминокислотные остатки - в шарнирной модели, AMPPNP - в проволочной) | 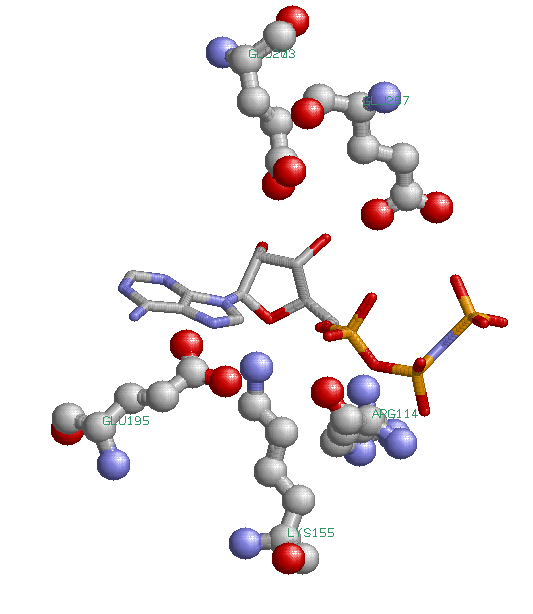
|
| Контакт GAR c Glu 82, Asp 286, Lys 355, Arg 362, и Arg 363 (GAR - в проволочной модели, аминокислотные остатки - в шарнирной модели) (1EZ1 в RasMol) | 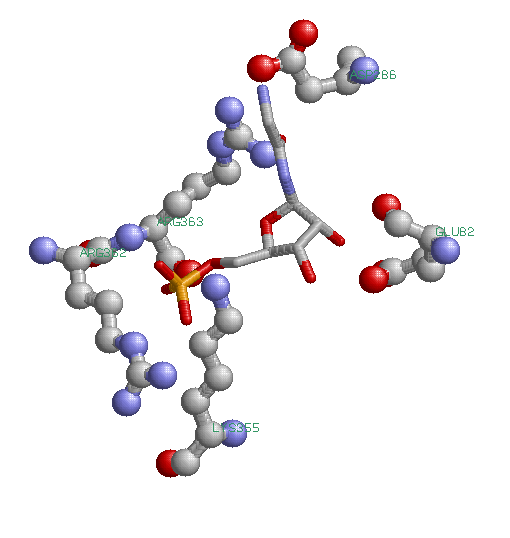
|
| И действительно, аминогруппа GAR, формилируемая в ходе реакции, расположена на расстояние даже меньше, чем 2.8 Å от одного из кислородов AMPPNP, связанных с фосфором. | 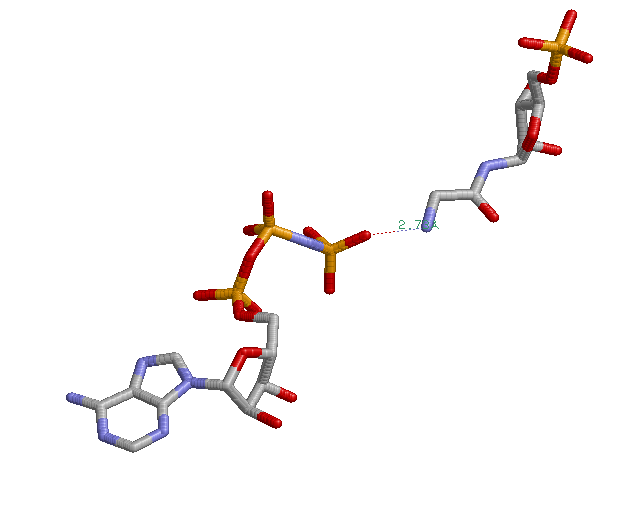
|
Всё как в аннотации, хотя может быть это потому, что файлы 1EYZ и 1EZ1 прилагаются к статье)
Третичная структура белка (1EYZ в RasMol): Бета-листы показаны синими, альфа-спирали - зелеными.

Идентификаторы белка в различных базах данных:
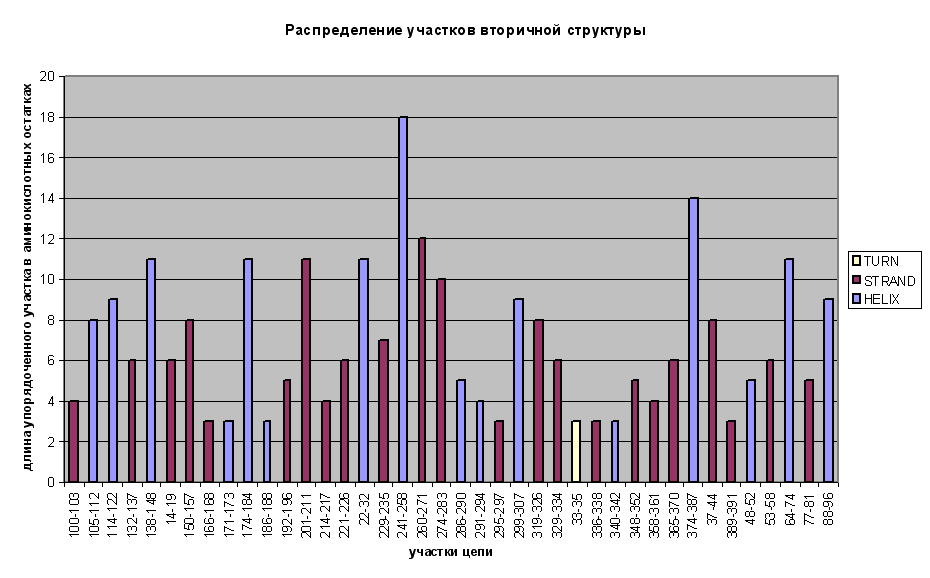
Распределение участков вторичной структуры в последовательности белка (turn - бета-поворот, strand - бета-тяж, helix - альфа-спираль):
(на основании записи о белке в UniProt )
Больше о структуре белка Purt организма E.Coli (код в базе данных PDB - 1EYZ ):
Элементы вторичной структуры: