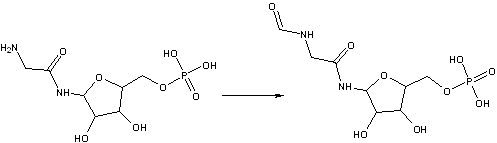
Схема реакции:
Код фермента
В качестве примера фермента рассмотрим белок PURT_ECOLI ( p33221 ):
PURT_ECOLI - фосфорибозилглицинамидформилтрансфераза 2. Код фермента (EnzymeCode): EC=2.1.2.- означает, что:
2 - фермент относится к классу трансфераз (transferases)
2.1 - трансфераза, переносящая одноуглеродные группы (transferring one-carbon groups)
2.1.2 - гидроксиметил- или формилзависимые трансфераза (Hydroxymethyl-, Formyl- and Related Transferases)
Фремент фосфорибозилглицинамидформилтрансфераза 2 катализирует реккцию: АТФ + формат + N(1)-(5-фосфо-D-рибозил)глицинамид = N(2)-формил-N(1)-(5-фосфо-D-рибозил)глицинамид + АДФ + дифосфат
Схема реакции:

Для PURT_ECOLI, на сайте International Union of Biochemistry and Molecular Biology не представлено описания и полного код фермента.
К данному ферменту наиболее близка фосфорибозилглицинамидформилтрансфераза (например PUR3_ECOLI ) (EC 2.1.2.2 phosphoribosylglycinamide formyltransferase). PUR3_ECOLI также катализирует реакцию образования N(2)-формил-N(1)-(5-фосфо-D-рибозил)глицинамида из N(1)-(5-фосфо-D-рибозил)глицинамидa, однако в качестве субстрата не использует
АТФ, а вместо формата использует 10-формилтетрагидрофолат:
10-формилтетрагидрофолат + N(1)-(5-фосфо-D-рибозил)глицинамид = N(2)-формил-N(1)-(5-фосфо-D-рибозил)глицинамид + тетрагидрофолат
С работой PURT_ECOLI связана побочная реакция: получения ацетилфосфата и АДФ из уксусной кислоты и АТФ. PUR3_ECOLI не катализирует такую реакцию, поскольку вообще не использует АТФ в качестве субстрата.
Поиск метаболического пути, в котором участвует PURT_ECOLI:
Имя локуса гена белка PURT_ECOLI:b1849
В базе данных KEGG были найдены метаболические пути, ассоциированные с геном b1849:
| идентификатор KEGG | русское название | английское название |
| eco00230 ( карта пути ) | Пуриновый метаболизм | Purine metabolism |
| eco00670 | Перенос одноуглеродных групп фолатом | One carbon pool by folate |
| eco01100 | Метаболические пути | Metabolic pathways |
Поиск структурных формул химических соединений в KEGG:
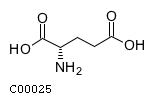
1) L-глутамат (L-глутаминовая кислота) - русское название
L-Glutamate (L-Glutamic acid, L-Glutaminic acid) - английское название
C00025 - идентификатор KEGG
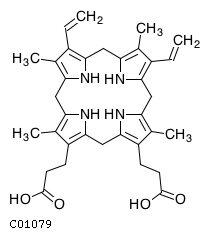
2) протогем - русское название
protoporphyrin - английское название
C01079 - идентификатор KEGG
Метаболический путь от L-глутамата к протогему:
Выбранная цепочка ферментативных реакций:
путь: метаболизм порфирина и хлорофилла ( ko00860 )
цепочка: L-глутамат → протогем, реакцияя протекают только в одном направлении
Промежуточные соединения (идентификаторы KEGG): C02987, C03741, C00430, C00931, C01024, C01051, C03263.
На карте промежуточные соединения окрашены желтым, L-глутамат - красным, протогем - зеленым: карта .
Возможен ли этот метаболический путь в разных организмах?
| Организм | Возможна ли цепочка реакций | Обоснование |
| Escherichia coli K-12 MG1655 | да ( карта пути ) | присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Archaeoglobus fulgidus | неизвестно | неизвестены гены ферментов, катализирующих 7 (EC 4.1.1.37) и 8 (EC 1.3.3.3 или EC 1.3.99.22) стадии |
| Arabidopsis thaliana | неизвестно | неизвестены гены ферментов, катализирующих 6 (EC 4.2.1.75) и 7 (EC 4.1.1.37) стадию |
| Homo sapiens | неизвестно | неизвестны гены ферментов, катализирующих 2 (EC 1.2.1.70) и 3 (EC 5.4.3.8) стадию |
Сравнение ферментов из далеких организмов.
1) Найдем в UniProt ферменты с кодом EC 2.7.7.4 (сульфатаденилилтрансферазы) у человека и археи Archaeoglobus fulgidus , для этого обратимся к SRS с запросом: ([uniprot-ECNumber:2.7.7.4] & ([uniprot-ID:*_Human] | [uniprot-ID:*_ARCFU]))
При поиске необходимо снять опцию "Use wildcards" для того, чтобы поиск проводился по ферментам EC 2.7.7.4, а не EC 2.7.7.4*, иначе будут также найдены ферменты, EC которых начинается на 2.7.7.4 (например 2.7.7.40).
По запросу найдено 4 разных фермента, 3 из которых относятся к человеку (PAPS1_HUMAN, PAPS2_HUMAN, Q9UIR2_HUMAN) и 1 к Archaeoglobus fulgidus (SAT_ARCFU).
2) Сравнение доменной организации (согласно Pfam) найденных белков:
51-209 а.о. 295-618 а.о.
82-424 а.о.
Как видно из рисунка, PAPS1_HUMAN и SAT_ARCFU содержат домен ATP-sulfurylase (PF01747), соответствующий сульфатаденилилтрансферазам (EC 2.7.7.4), однако человеческий белок
содержит также APS_kinase (PF01583) домен, соответствующий ферментам, присоединяющим фосфат к 3'-гидроксилу аденилилсульфата.
Объясняется это тем, что у людей синтез фосфоаденозин-фосфосульфата осуществляется бифункциональным ферментом: на первой стадии образуется аденозин 5'-фосфосульфат (APS), 3'-гидроксил которого затем фосфорилируется. У бактерий же, грибов и растений - в этом процессе участвуют 2 белка - фермента (EC 2.7.7.4 и EC 2.7.1.25).
Доменная структура PAPS2_HUMAN схожа с PAPS1_HUMAN. Q9UIR2_HUMAN содержит только один домен (ATP-sulfurylase), вызвано это тем, что его аминокислотная последовательность определена не полностью (Sequence status - Fragment ).
3) Определим процент идентичности последовательностей доменов ATP-sulfurylase белка PAPS1_HUMAN и SAT_ARCFU: для этого сделаем выравнивание соответствующих фрагментов белков с помощью protein BLAST
идентичность: 23%
схожие а.о.: 40%
Процент идентичности не очень высок, однако у выравнивания низкое E-value: 2e-15.
4) Для гена PAPS1_HUMAN лучший ортолог среди генов архей - cysC из Aeropyrum pernix (ген аденилилсульфаткиназы EC 2.7.1.25). Идентичность: 44,4%, длина выравнивания: 183 а.о.
Для гена SAT_ARCFU лучший ортолог среди генов эукариот - ген EHI_197160 из амебы Entamoeba histolytica (ген сульфатаденилилтрансферазы EC 2.7.7.4). Идентичность: 30,3%, длина выравнивания: 423 а.о.
Схожие по функциям ферменты из далеких организмов не всегда обладают в высокой степени схожими аминокислотными последовательностями. В процессе эволюции, ферменты могут также приобретать дополнительные домены, усложняя свою функцию. Так PAPS1_HUMAN приобрел домен APS_kinase, что позволило синтезировать фосфоаденозин-фосфосульфат с использованием 1 бифункционального фермента.