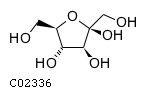
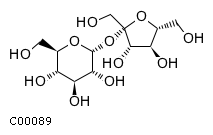
В качестве белка прототипа задан белок B5Y088_KLEP3 из бактерии Klebsiella pneumoniae (strain 342).
B5Y088_KLEP3 - репрессор сахарозного оперона, на странице UniProt нет информации об эффекторе белка.
Однако, согласно онтологии GO, B5Y088_KLEP3 изменяет свое состояние в присутсвии фруктозы (GO:0009750),
белок связывается с ДНК (GO:003677) и участвует в регуляции транскрипции (GO:0045449).
Поскольку белок - репрессор сахарозного оперона, в качестве эффектора должна выступать сахароза.

| 
|
| фруктоза | сахароза |
Создать хорошее множественное выравнивание доменов заданной группы белков.
Необходимо получить множественное выравнивание ДНК-связывающих доменов белков группы спецефичности scrr .
Определим доменную организацию белка-прототипа B5Y088_KLEP3, обладающего этой спецефичностью. Для этого обратимся к базе данных InterPro :
| Pfam домен PF00356 , HTH (спирал-поворот-спираль) ДНК-связывающий мотив | 
| координаты в аминокислотной последовательности: 7 - 55 а.о. |
| Smart домен SM00354 , HTH (спирал-поворот-спираль) ДНК-связывающий мотив | 
| координаты в аминокислотной последовательности: 5 - 78 а.о. |
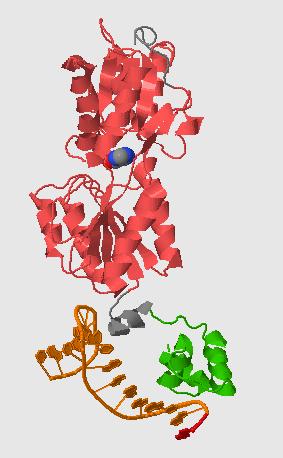
Домен SM00354 занимает большое количество аминокислотных остатков, чем PF00356. Как видно из пространственной структуры белка PURR_ECOLI (PDB ID: 1pnr), содержащего домен PF00356 (отмечен зеленым цветом на рисунке), вблизи ДНК располагается также участок белка, не включенный в домен PF00356 (отмечен серым цветом), поэтому для получения выравнивания ДНК-связывающих доменов воспользуемся базой данных Smart:
Представительское выравнивание доменов HTH_LACI из БД Smart: SMART.fasta
Последовательности всех бактериальных доменов этого семейства: SMART_bact.fasta
Далее из них были выбраны последовательности доменов белков, обладающих спецефичностью scrr ( scrr ): dna_scrr.fasta
С помощью программы ClustalW2 построено множественное выравнивание этих последовательностей по профилю SMART.fasta: dna_scrr_aln.fasta
Создать единое множественное выравнивание заданных доменов всех групп специфичности.
Множественное выравнивание эффекторсвязывающих доменов всех групп специфичности: eff_bind.msf
Изображение выравнивания: eff_bind.gif
Каждая группа отмечена своим цветом, а.о. остатки консервативные для группы окрашены цветом группы (Groups -> Shade Group Conservd), последовательности заданной группы scrr расположены сверху.
На позициях 52, 58, 73, 83, 90, 107, 108, 160, 169, 210, 212, 225, 253, 257, 262, 263 и 265 выравнивания расположены а.о., консервативные для группы scrr и не совпадающие с консервативными а.о. в других группах на тех же позициях.
На позиции 146 выравнивания распологаются гидрофобные а.о. валин, лейцин, изолейцин, метионин, консервативные для всех групп (отмечены черным).
Множественное выравнивание ДНК-связывающих доменов всех групп специфичности: dna_bind.msf
Изображение выравнивания: dna_bind.gif
Каждая группа отмечена своим цветом, а.о. остатки консервативные для группы окрашены цветом группы (Groups -> Shade Group Conservd), последовательности заданной группы scrr расположены сверху.
На позициях 25, 49, 69 и 86 выравнивания расположены а.о., консервативные для группы scrr и не совпадающие с консервативными а.о. в других группах на тех же позициях.
Создайте лого-изображения полного выравнивания заданных доменов и выравнивания доменов заданной группы специфичности.
Logo диаграмма для полных выравниваний: eff_logo.gif - эффекторсвязывающего домена, dna_logo.gif - ДНК-связывающего домена.
Logo диаграмма для выравниваний доменов группы scrr: eff_scrr_logo.gif - эффекторсвязывающего, dna_scrr_logo.gif - ДНК-связывающего.
Высота букв на рисунке logo соответсвует частотам встречаемости соответсвующих а.о. в выравнивании.
Как и следовало ожидать на logo группы scrr большое количество однозначно встречаемых а.о., в то время как на тех же позициях общего logo отражены различные варианты, представленные в различных группах спецефичности.
Создание профиля для поиска на основании доменов белков группы scrr.
Воспользуемя программами пакета PFTOOLs для добавления веса в выравнивания dna_scrrgr.fasta , eff_scrrgr.fasta , затем для построения и нормирования относительно
случайной базы профиля:
pfw -m dna_scrrgr.fasta > dna_scrrgr.weighted.fasta
pfmake -m dna_scrrgr.weighted.fasta /usr/share/pftools23/blosum45.cmp > dna_profile.prf
autoscale -m dna_profile.prf > dna_profile.scaled.prf
В итоге получим 2 профиля dna_profile.scaled.prf и (ан-но) eff_profile.scaled.prf .
Поиск по протеому Staphylococcus xylosus.
Найдем последовательности белков из протеома Staphylococcus xylosus в БД TrEMBL через SRS запрос Staphylococcus xylosus. В TrEMBL найдено всего 215 белков Staphylococcus xylosus, странно, но в UniProt найдено больше, чем в TrEMBL 259, возпользуемся последовательностями UniProt. Файл с находками.
Теперь проведем поиск по протеому:
pfsearch -C 20.0 -f dna_profile.scaled.prf staxy.fasta > dnas_satxy.search
pfsearch -C 20.0 -f eff_profile.scaled.prf staxy.fasta > eff_staxy.search
При пороге 20 не найдено никаких находок, однако при пороге 10, в случае ДНК-связывающего домена найдено 3 белка ( dnas_satxy.search ), а в случае эффекторсвязывающего домена - 2 ( eff_staxy.search ).
Хорошо, что 2 находки совпадают: CCPA_STAXY - предполагамый регулятор катаболизма, SCRR_STAXY - репрессор сахарозного оперона.
3-ая находка по ДНК-связывающему домену - MALR_STAXY - репрессор мальтозного оперона, содержащий, как и белок прототип B5Y088_KLEP3, ДНК-связывающий мотив HTH (спираль-поворот-спираль), однако распознающий в качестве эффектора не сахарозу, а мальтозу, поэтому понятно, почему этот белок найден при поиске по ДНК-связывающему домену, но не найден при поиске по эффекторсвязывающему домену.
Определим, совпали ли на выравнивании консервативные для ДНК-связывающего домена а.о., для этого построим на множественное выравнивание ДНК-связывающих доменов scrr и последовательностей находок, полученное программой muscle (файлс с выравниванием: dna_staxy_aln.fasta ):

При сравнении с logo диаграммой ДНК-связывающих доменов scrr находим, что почти все консервативные позиции совпадают (A 15 (координаты для выравнивания), S 21, T 24, S 26, N 30, T 42, Y 55, A 61, L 64, G 73), некоторые не консервативны для белка MALR_STAXY N 30, R 68, N 86. Однако в целом выравнивание хорошее, с высокой достоверностью можно полагать, что найденные белки обладают схожим ДНК-связывающим доменом.
К тому же из предположения о том, что находки - репрессоры сахарозного оперона, не следует, что они должны иметь идентичный ДНК-связывающий домен, ведь в разных бактериях последовательность ДНК сахарозного опреона может несколько отличаться.
Теперь посмторим на аналогичное выравнивание эффекторсвязывающего домена ( eff_staxy_aln.fasta ):
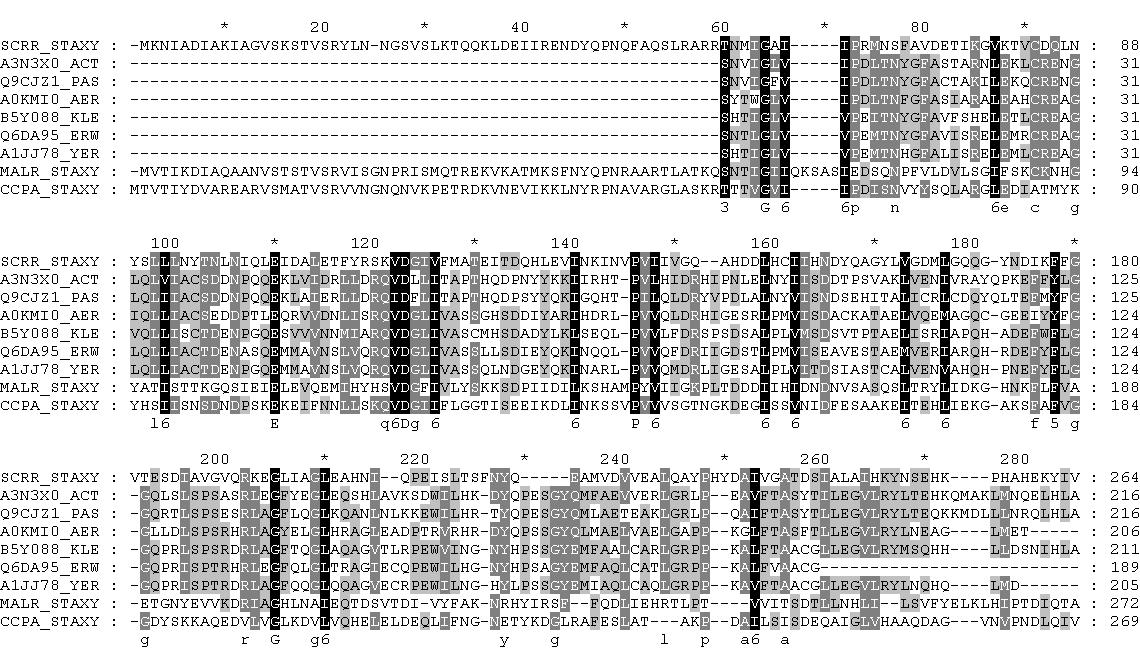
Многие консервативные а.о. для эфекторсвязывающего домена scrr также консервативны и для найденных белков: G 64 (координаты выравнивания), G 205, L 210, P 146, D 123, E 110, L 118... В некоторых случаях в найденных белках происходит замена на аминокислоту с похожими свойствами (V 66 на I, I/V 72...). На некоторых позициях предполагаемый регулятор ктаболизма CCPA_STAXY имеет а.о. более близкие по свойствам к а.о. scrr, чем репрессор сахарозного оперона SCRR_STAXY: T 76, у CCPA_STAXY - S, а у SCRR_STAXY - N; N 77, у CPPA_STAXY - N, а у SCRR_STAXY - S; F 80, у CCPA_STAXY ароматическая Y, а у SCRR_STAXY - V; Q 121 у CCPA_STAXY - Q, а у SCRR_STAXY - K; L 118 у CPPA_STAXY - L, а у SCRR_STAXY - Y.
Есть отличии между последовательностью MALR_SATXY и последовательностями остальных белков: на позиции 73 выравнивания у всех белков, кроме MALR_STAXY, пролин - довольно редкая аминокислота, обеспечивающая поворот белковой цепи. 147 V/I соответсвует Y MALR_STAXY...
Для белков группы специфичности scrr не представлено ни одной пространственной структуры, выводы о значимости замен а.о. приходится делать только на основании выравнивания.
Вывод:
В протеоме Staphylococcus xylosus найден белок SCRR_STAXY обладающий scrr специфичностью, также, возможно, этой специфичностью обладает регулятор катаболизма CCPA_STAXY. В протеоме найден белок MALR_STAXY по сходству с ДНК-связывающим доменом грппы scrr. Из БД UniProt удалось получить последовательности только 259 белков Staphylococcus xylosus, среди остальных белков данного организма возможно могут находиться еще несколько представителей, обладающих scrr специфичностью.