Укоренение дерева в стеднюю точку с помощью программы retree пакета Phylip:
Укореним дерево, полученное методом neighbor-joining, в среднюю точку:
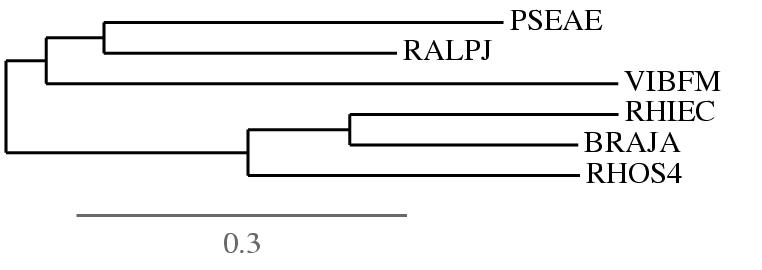
Филогенетическое дерево, построенное для бактерий методом neighbor-joining, на основании множественного выравнивания последовательностей белка семейства PROZ (омега-субъединица ДНК-зависимой РНК-полимеразы):
Файд со скобочной формулой
+SALTY
!
! +--VIBFM
! !
1--2 +-------RALPJ
! ! +-5
! ! ! +----------PSEAE
! +------------6
! ! +--------RHOS4
! +-------4
! ! +-----BRAJA
! +--3
! +------RHIEC
!
+ECOLI
Получееное дерево:

На этом дереве укоренение произошло в нетривиальную ветвь {PSEA, RALPJ, VIBFM} против {RHIEC, BRAJA, RHOS4}, которой нет на дереве neighbor-joining.
На дереве остутсвуют SALTY и ECOLI, возможно потому, что оно строилось с учетом эволюционного расстояни (корень - точка равноудаленная от всех листов), а согласно файлу программу fneighbor (алгоритм Neighbor-Joining), расстояние до ECOLI и SALTY = 0 (они слились между собой и с корнем в одну точку):
SALTY:0.00000,(VIBFM:0.10448,((RALPJ:0.26606,PSEAE:0.36292):0.05241,
(RHOS4:0.30141,(BRAJA:0.20746,RHIEC:0.24388):0.09238):0.25605):0.41507):0.09782,ECOLI:0.00000);
Полученное дерево отличается от правильного дерева также наличием ветви {PSEAE, RALPJ} против остальных бактерий:
Использование аутгруппы:
В качестве аутгруппы используем белок семейства RPOZ (омега-субъединица ДНК-зависимой РНК-полимеразы) из сенной палочки (Bacillus subtilis, BACSU) ( fasta-файл с последовательностями белков)
Fasta-файл с выровненными последовательностями (программой muscle)
Результат работы программы fprotpars с выровненными последовательностями: файл со скобочной структурой, файл с деревом. BACSU образут тривиальную ветвь против всех остальных бактерий.
После обработки программой retree (в качестве аутогруппы выбрана BACSU), укорененное дерево: ( файл со скобочной формулой)

В полученном дереве, по сравнению с правильным , есть ветвь {PSEAE, VIBFM, SALTY, ECOLI, RHIEC, BRAJA, RHOS4} против {RALPJ} (если рассматривать поддерево без BACSU), вто время как в правильном дереве есть ветвь {VIBFM, SALTY, ECOLI, PSEAE, RALPJ} против {RHOS4, RHIEC, BRAJA} (на правильном дереве RALPJ (бетапротеобактерия) ближе к гаммапротеобактериям (ECOLI, SALTY, VIBFM, PSEAE))
Добавленный BACSU относится к типу Firmicutes, остальные же - к типу протеобактерий. Такая отдаленность согласутся с деревом.
Бутстрэп:
Проведем бутстрэп-анализ филогении на основе множественного выравнивания :
+------ECOLI
+100.0-|
+100.0-| +------SALTY
| |
+100.0-| +-------------VIBFM
| |
| | +------PSEAE
+100.0-| +--------76.5-|
| | +------RALPJ
+------| |
| | +---------------------------RHOS4
| |
| +----------------------------------BRAJA
|
+-----------------------------------------RHIEC
Тривиальный ветви всегда имеют 100% поддержки (так как всегда представлены на деревьях) (ветви {RHOS4} против остальных, {BRAJA} против остальных, {RHIEC} против отсальных)
Полученное дерево совпадает с результатом fprotpars на исходном выравнивании. Отличие от правильного дерева - наличие ветви {PSEA, RALPJ} против {RHIEC, BRAJA, RHOS4, VIBFM, SALTY, ECOLI}.
Ветви, не включенные в консенсунсное дерево:
{ECOLI, SALTY, VIBFM, RALPJ} против {PSEAE, RHOS4, BRAJA, RHIRC} (23%) - такой ветви нет в правильном дереве
малую поддержку (0,5%) получила ветвь {ECOLI, SALTY, VIBFM, PSEAE} против {RALPJ, RHOS4, BRAJA, RHIEC} - она есть на правильном дереве, но отсутсвует на всех построенных деревьях.