Фиолгенетическое дерево отобранных бактерий:
| Название | Мнемоника |
| Bradyrhizobium japonicum | BRAJA |
| Rhizobium etli | RHIEC |
| Ralstonia pickettii | RALPJ |
| Escherichia coli | ECOLI |
| Salmonella typhimurium | SALTY |
| Rhodobacter sphaeroides | RHOS4 |
| Pseudomonas aeruginosa | PSEAE |
| Vibrio fischeri | VIBFM |
Скобочная формула дерева:
(((BRAJA, RHIEC), RHOS4), (RALPJ, (PSEAE, (SALTY, ECOLI), (VIBFM))))
Изображение дерева:
Ветви дерева:
Дерево содержит нетривиальные ветви:
1) {BRAJA, RHIEC} против {RHOS4, RALPJ, PSEA, ECOLI, SALTY, VIBFM}
2) {BRAJA, RHIEC, RHOS4} против {RALPJ, PSEA, ECOLI, SALTY, VIBFM}
3) {BRAJA, RHIEC, RHOS4, RALPJ} против {PSEA, ECOLI, SALTY, VIBFM}
4) {BRAJA, RHIEC, RHOS4, RALPJ, PSEA} против {ECOLI, SALTY, VIBFM}
5) {BRAJA, RHIEC, RHOS4, RALPJ, PSEA, VIBFM} против {ECOLI, SALTY}
Таксоны:
Отобранные бактерии относятся к 3 классам: Альфапротеобактериям (BRAJA, RHIEC, RHOS4), Бетапротеобактериям (RALPJ) и Гаммапротеобактериям (ECOLI, SALTY, PSEAE, VIBFM). Дерево соответсвует такому разделению бактерий на таксоны. Кроме того, бактерии BRAJA и RHIEC входят в один порядок Rhizobiales, а ECOLI и SALTY - в Enterobacteriales, что также отражено на дереве.
Для реконструкции выбрано семейство белков RPOZ (омега-субъединица ДНК-зависимой РНК-полимеразы). По файлу с последовательностями программой muscle сделано множественное выравнивание . Файл GeneDoc:
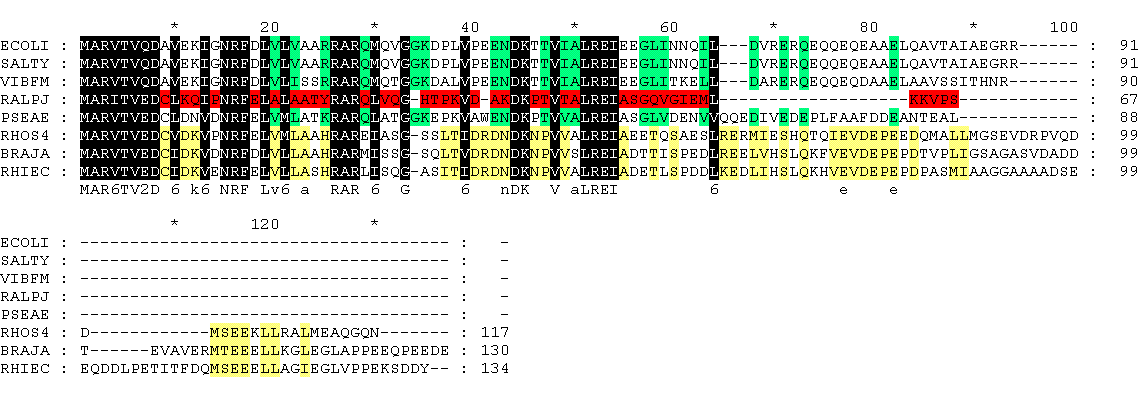
Зеленым цветом омечены консервативные позиции белков гаммапротеобактерий, желтым - альфапротеобактерий, красным - бетапротебактерии. Первые 64 позиции выравнивания богаты консервативными позициями, характерными для всех выбранных таксонов. Как видно из выранивания, альфапротеобактерии обладают более длинными омега-субъединицами ДНК-зависимой РНК-полимеразы, на их концах также располагаются консервативные участки.
На выравнивании видно также большее сходство у представителей одого порядка Rhizobiales (BRAJA и RHIEC): E на 118 позиции выравнивания, 122 - G, 124 - E, 125 - G, 126 - A, 128 - P, 129 - P, 130 - E, 93 - G, 94 - A - также являюься консервативными позициями для них.
Программа fprotpars:
Программа fprotpars выдала одно дерево ( файл ).
+--RHIEC
+--7
+-----6 +--BRAJA
! !
+--5 +-----RHOS4
! !
! ! +--PSEAE
+--3 +--------4
! ! +--RALPJ
+--2 !
! ! +--------------VIBFM
1 !
! +-----------------SALTY
!
+--------------------ECOLI
Скобочная формула дерева: ((((((RHIEC,BRAJA),RHOS4),(PSEAE,RALPJ)),VIBFM),SALTY),ECOLI)
Полученное дерево отличается от правильного: у полученного дерева есть ветвь {PSEA, RALPJ} против {RHIEC, BRAJA, RHOS4, VIBFM, SALTY, ECOLI}. В оба дерева входит поддерево (RHOS4, (BRAJA, RHIEC)), объеденяющае альфапротеобактерий и демонстрирующае большое родство между BRAJA и RHIEC, относящимся к одному порядку Rhizobiales. Из-за меньшего числа консервативных позиций в белке RPOZ, представители гаммапротеобактерий (ECOLI, SALTY, PSEAE, VIBFM) разобщены сильнее на полученном дереве (отсутсвует ветвь {PSEAE, VIBFM, SALTY, ECOLI} против {RALPJ, RHOS4, RHIEC, BRAJA}).
Программа fprotdist:
Матрица расстояний ( файл ):
8 ECOLI SALTY VIBFM RALPJ RSEAE RHOS4 BRAJA RHIEC
ECOLI 0.000000 0.000010 0.202306 0.824727 0.901105 1.133481 1.032552 1.068326
SALTY 0.000010 0.000000 0.202306 0.824727 0.901105 1.133481 1.032552 1.068326
VIBFM 0.202306 0.202306 0.000000 0.854818 0.951874 1.101444 1.031764 1.053574
RALPJ 0.824727 0.824727 0.854818 0.000000 0.628981 0.854974 0.887133 0.919653
PSEAE 0.901105 0.901105 0.951874 0.628981 0.000000 0.949981 0.976538 1.068248
RHOS4 1.133481 1.133481 1.101444 0.854974 0.949981 0.000000 0.623121 0.615785
BRAJA 1.032552 1.032552 1.031764 0.887133 0.976538 0.623121 0.000000 0.451340
RHIEC 1.068326 1.068326 1.053574 0.919653 1.068248 0.615785 0.451340 0.000000
Пример ультраметричности: d(RALPJ, ECOLI)=d(RALPJ, SALTY)>d(ECOLI,SALTY)
Пример отклонения от ультраметричности: d(BRAJA, RHOS4) < d(RALPJ, RHOS4) < d(RALPJ, BRAJA)
Для 4 последовательностей: ECOLI, SALTY, RHOS4, RHIEC выполняется свойство аддитивности: d(ECOLI, SALTY)+d(RHOS4, RHIEC)=0,615795; d(ECOLI, RHOS4)+d(SALTY, RHIEC)=d(ECOLI, RHIEC)+d(SALTY, RHOS4)=1,133481 + 1,068326
Вообще, можно заметить, что расстояние от ECOLI до любой другой бактерии, кроме SALTY, равно расстоянию от SALTY до той же бактерии. Расстояние же от ECOLI до SALTY - минимальное среди всех остальных расстояний, следовательно свойство ультраметричности выполняется. Однако оно выполняется только для троек бактерий, включающих ECOLI и SALTY. Для остальных троек наблюдается отклонение от ультраметричности.
Программа fneighbor, алгоритм Neighbor-Joining:
Полученный файл . Файл со скобочной структурой Дерево:
+SALTY
!
! +--VIBFM
! !
1--2 +-------RALPJ
! ! +-5
! ! ! +----------PSEAE
! +------------6
! ! +--------RHOS4
! +-------4
! ! +-----BRAJA
! +--3
! +------RHIEC
!
+ECOLI
На полученном дереве, как и на правильном, дереве программы fprotpars отражено близкое родство альфапротеобактерий: поддерево (RHOS4, (BRAJA, RHIEC)).
Полученное дерево совпадает с деревом fprotpars.
Программа fneighbor, алгоритм UPGMA:
Полученный файл . Файл со скобочной структурой. Укорененное дерево:
+ECOLI
+-----1
+--------------------2 +SALTY
! !
+---6 +-----VIBFM
! !
! ! +------------------RALPJ
! +-------5
--7 +------------------PSEAE
!
! +------------------RHOS4
+-----------4
! +-------------BRAJA
+----3
+-------------RHIEC
Во всех построенных деревьях и в правильном дереве есть поддеревья ((RHOS4, (BRAJA, RHIEC)) и ((ECOLI, SALTY), VIBFM).
Все построенные деревья совпадают между собой и отличаются от правильного наличаем ветви {PSEA, RALPJ} против {RHIEC, BRAJA, RHOS4, VIBFM, SALTY, ECOLI}.
Возможно для реконструкции филогенетического дерева не хватает сравнения только по одному белку, поэтому построенные деревья отличаются от правильного.