Краткое описание структуры в файле 1VAS.pdb (скачать файл ):
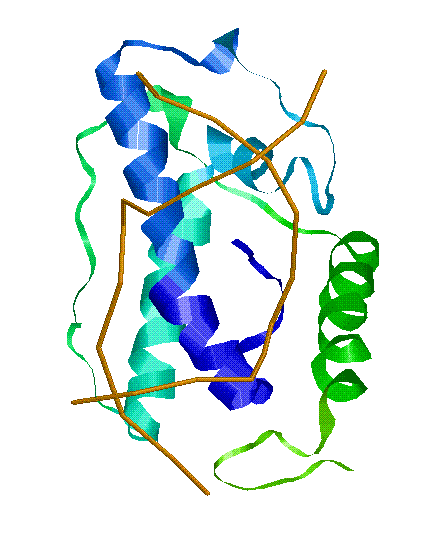
В файле приведены координаты атомов 2 молекул ДНК, 1 молекулы белка T4 эндонуклеазы V.
Организм: энтеробактериофаг T4 (вирус энтеробактерий), белок эксперссировался в Escherichia coli
Для исследования выбраны цепь белка A и цепи B, C ДНК со следующей последовательностью:
цепь B [201] 5'- a t c g c g t t g c g c t - 3' [213]
| | | | | | | | | | | |
цепь C [226] 3'- a g c g c a a c g c g a t - 5' [214]
Функции белка T4 эндонуклеазы V:
Идентификатор белка T4 эндонуклеазы V в базе данных UniProt: P04418
T4 эндонуклеаза V - фермент гликозидаза, удаляющая димеры пиримидиновых оснований из ДНК. Такие димеры образуются в ДНК под действием ультрафиолетового излучения, таким образом фермент участвует в репарации ДНК.
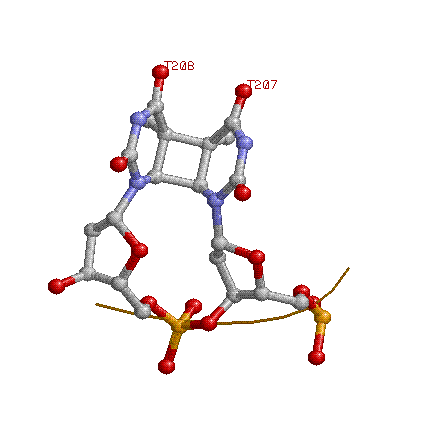
В цепи B ДНК как раз содержится димер тимина (на 207-208 позициях):

Исследование структуры ДНК:
С помощью программ remediator, find_pair, analyze из файла 1VAS.pdb получен файл 1VAS_old.out , содержащий информацию о структуре ДНК.
Из таблицы этого файда видно, что ДНК - в B-форме.
В этом же файле находится таблица с торсионными углам нуклеотидов:
Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I
base alpha beta gamma delta epsilon zeta chi
1 T --- 178.3 49.9 134.6 -160.8 -105.9 -125.4
2 C -72.3 -178.8 49.8 132.6 -175.0 -115.1 -110.6
3 G -53.6 167.3 54.0 139.5 -167.3 -96.7 -116.3
4 C -72.6 -173.5 40.0 122.6 176.7 -109.4 -95.0
5 G -54.0 -173.2 44.0 132.8 --- --- -96.5
6 T --- 166.8 43.8 114.2 174.7 -137.9 -119.0
7 G -47.2 154.8 64.1 140.3 -167.2 -103.2 -112.1
8 C -55.0 169.2 51.4 132.2 -161.1 -134.4 -106.4
9 G -61.3 160.9 55.7 137.4 -164.5 -104.6 -115.6
10 C -54.8 169.1 43.3 127.3 -179.5 -114.8 -106.4
11 T -54.0 167.2 57.2 134.4 --- --- -122.0
Strand II
base alpha beta gamma delta epsilon zeta chi
1 A -71.8 -169.9 61.4 135.6 --- --- -101.8
2 G -76.4 165.8 62.6 135.7 173.6 -104.6 -107.4
3 C -53.3 170.8 56.8 132.6 -158.9 -107.4 -125.5
4 G -76.7 166.9 70.4 127.1 176.6 -116.2 -103.5
5 C --- -170.4 102.2 136.8 -179.3 -106.7 -93.7
6 A -70.7 177.8 45.2 129.2 --- --- -72.5
7 C -51.7 161.4 58.5 139.1 -155.6 -98.8 -119.3
8 G -69.5 168.1 69.8 132.3 176.0 -127.1 -90.5
9 C -51.3 174.7 47.7 132.8 -161.0 -107.9 -109.1
10 G -51.6 170.2 47.3 130.5 179.0 -107.0 -103.9
11 A --- 175.4 47.1 132.1 -171.5 -108.5 -104.5
Для каждого торсионного угла я определил средний угол среди внутренних нуклеотидов, получив "средний нуклеотид", наешл сумму его углов и сравнил с такой же суммой углов каждого внутреннего нуклеотида. Там, где эта разница велика - "кривой" нуклеотид, сумма его торсионных углов значительно отличается от суммы углов "среднего" нуклеотида. Формулы и вычисления в таблице . Если считать разницу больше 200 по модулю значительной, получаем "кривые нуклеотиды": 203, 206, 207, 216, 218, 219, 223, 225. Причем интересно, что "кривые" 203 и 225 комплементарны. ДНК довольно сильно деформировано, особенно в области димерного тимина, в комплексе с белком, нити ДНК в этом месте раздвинуты (на рисунке ниже), этому месту соответсвуют "кривые" нуклеотиды 206, 207, 219, 218.
В деформацию ДНК внесло вклад не только взаимодействие с белком, но и содержание в последовательности димера тимина.
Исследование природы ДНК-белковых контактов:
Будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 5 | 46 | 51 |
| остатками фосфорной кислоты | 14 | 17 | 31 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 20 | 22 |
| остатками азотистых оснований со стороны малой бороздки | 6 | 11 | 17 |
Данные получены с помощью скрипта my_dna.def в RasMol.
Как видно из таблицы, во взаимодействии белка с ДНК преобладают гидрофобные контакты, особенно их много с атомами 2'-дезоксирибозы. Это согласуется с тем, что изучаемая ДНК находится в B-форме, для которой характерно размещение гидрофобных участков внутри двойной спирали, а поскольку белок и "лезет" внутрь, в его взаимодействии с ДНК преобладают гидрофобные контакты.
Получение популярной схемы ДНК-белковых контактов с помощью nucplot:
С пмощью программы nucplot получаем схему взаимодействия белка и ДНК:
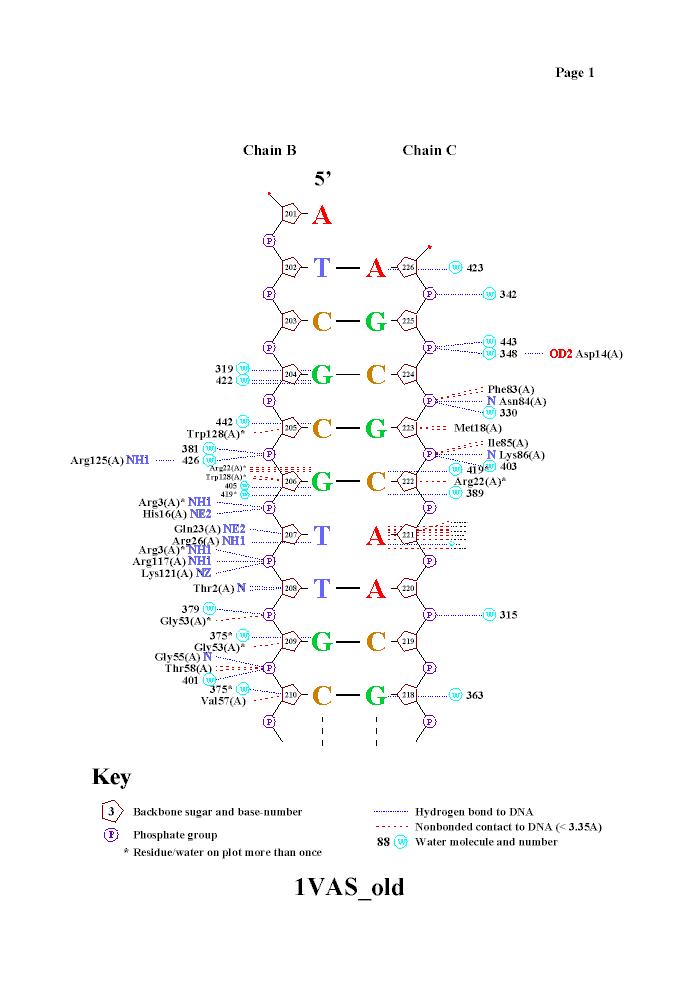
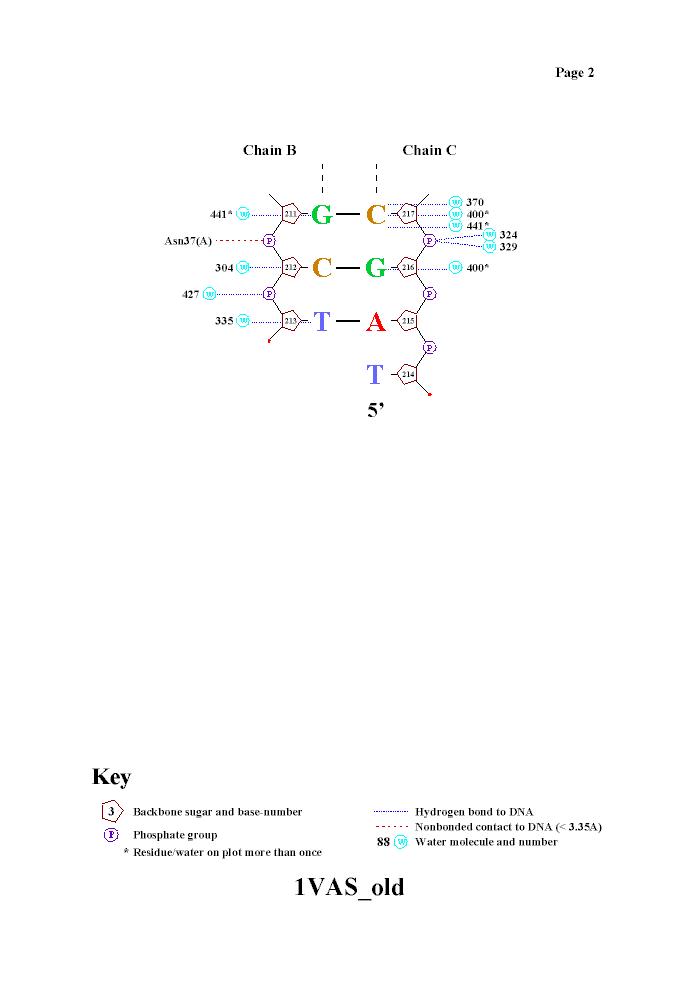
Из схемы видно, что с ДНК взаимодействует еще и много молекул воды. Заметим также, что nucplot выдал мощное гидрофобное взаимодействие с 221 нуклеотидом, большое число аминокислот взаимодействует с ДНК в области димера тимина (207, 208 нуклеотиды). Отсюда можно сделать предположение о распознающих контактах.
Возможные распознающие контакты:
Поскольку димер тимина - необычная структура, не очень часто встречающаяся в ДНК, 207 и 208 нуклеотиды, на мой взгляд, хорошие кандидаты на роль распознающего контакта. К тому же интересна ориентция 221 нуклеотида (напротив димера тимина) - его азотистое основание выставлено к наружу от двойной спирали:
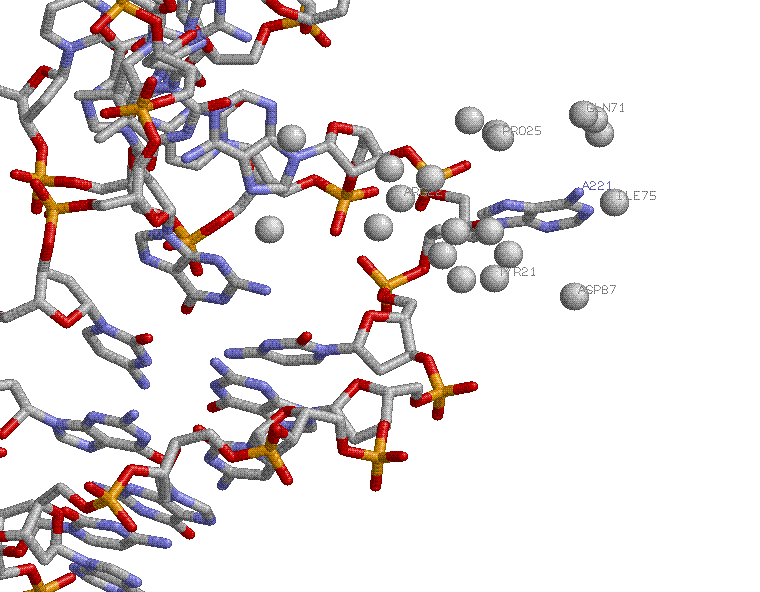
| рисунок получен с помощью скрипта my_dna.def , на нем изображена молекула ДНК и атомы белка, гидрофобно взаимодействующих с атомами оснований большой бороздки. Довольно много атомов белка взаимодействуют с 221 нуклеотидом, повернутым от спирали ДНК. |
Характеристика ДНК-связывающего домена P04418 :
С помошью инструментов Pfam определим доменную структуру T4 эндонуклеазы V (P04418):

Белок содержит всего 1 домен, котрый занимает весь белок целиком (с 1 по 138 аминокислоту (в pdb файле 1VAS.pdb в белке 137 аминокислот: 1-ая метионин отсутсвует))
Описание домена в InterPro: IPR004260 Pyrimidine dimer DNA glycosylase Пиримидин димер ДНК гликозидаза участвует в репарации днк, вырезая димерный нуклеотид.