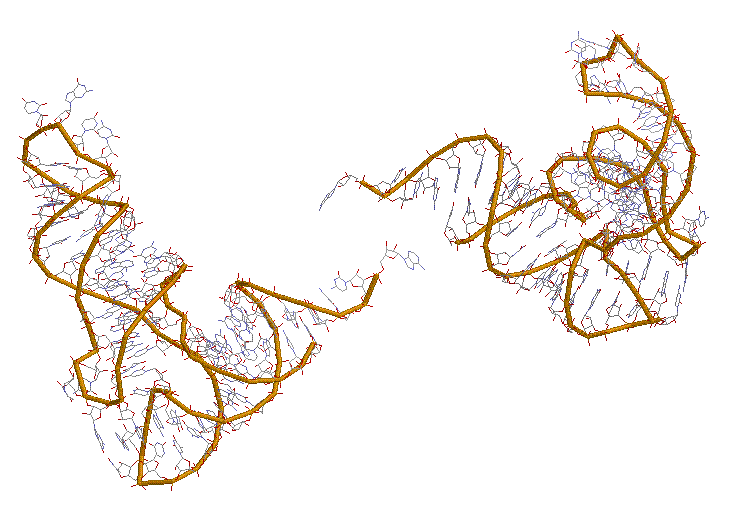
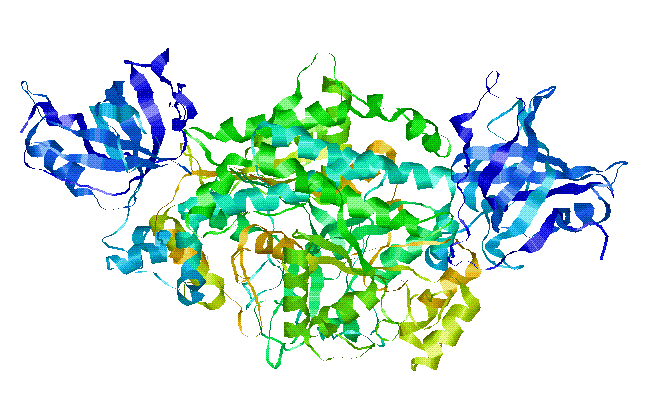
Краткое описание структуры в файле 1asz.pdb
Молекулы выделены из организма дрожжей Saccharomyces cerevisiae
В файле приведены координаты атомов комплекса аспартил-тРНК-синтетазы и ее субстрата - тРНК:
| транспортаня РНК (75-MER), 75 азотистых оснований, цепи не содержат разрывов, цепи R, S | 
|
| аспартил-тРНК-синтетаза, 490 аминокислотных остатков, цепи A, B | 
|
В структуре также содержится лиганд - 2 молекулы АТФ. АТФ участвует в реакции ацетилирования, в кристалле расположены около 3'-концов тРНК.
| Идентификатор | Название | Позиция в молекулах РНК |
| PSU | Псевдоуридин-5'-монофосфат | 613, 632, 655 |
| H2U | 5,6-дигидроуридин-5'-монофосфат | 616, 619 |
| 1MG | 1N-метилгуанозин-5'-монофосфат | 637 |
| 5MC | 5-метилцитозин-5'-монофосфат | 649 |
| 5MU | 5-метилуридин-5'-монофосфат | 654 |
Для исследования выбрана цепь R, представляющая тРНК со следующей последовательностью:
[601] 5'- U C C G U G A U A G U U PSU
A A H2U G G H2U C A G A A U G
G G C G C PSU U G U C 1MG C G
U G C C A G A U 5MC G G G G
5MU PSU C A A U U C C C C G U
C G C G G A G C C A -3' [676]
В последовательности на 3'-конце есть триплет CCA, к которому присоединяется аспарагиновая аминокислота, в файле приведены координаты атомов триплета.
Исследование вторичной структуры
С помощью программ find_pair и analyze пакета 3DNA определены возможные водородные связи между азотистыми основаниями: файл 1ASZ_old.out
Рассмотри участок этого файла:
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
1 (0.014) R:.601_:[..U]U-----A[..A]:.672_:R (0.011) |
2 (0.014) R:.602_:[..C]C-----G[..G]:.671_:R (0.012) |
3 (0.015) R:.603_:[..C]C-----G[..G]:.670_:R (0.014) |
4 (0.011) R:.604_:[..G]G-----C[..C]:.669_:R (0.016) |
5 (0.016) R:.605_:[..U]U-*---G[..G]:.668_:R (0.011) |
6 (0.013) R:.606_:[..G]G-----C[..C]:.667_:R (0.014) |
7 (0.012) R:.607_:[..A]Ax----U[..U]:.666_:R (0.018) |
8 (0.012) R:.649_:[5MC]c-----G[..G]:.665_:R (0.014) |
9 (0.010) R:.650_:[..G]G-----C[..C]:.664_:R (0.009) |
10 (0.011) R:.651_:[..G]G-----C[..C]:.663_:R (0.014) |
11 (0.014) R:.652_:[..G]G-----C[..C]:.662_:R (0.014) |
12 (0.016) R:.653_:[..G]G----xC[..C]:.661_:R (0.014) |
13 (0.008) R:.654_:[5MU]u-**-xA[..A]:.658_:R (0.011) |
14 (0.011) R:.655_:[PSU]Px**+xG[..G]:.617_:R (0.013) x
15 (0.013) R:.639_:[..G]G-----C[..C]:.631_:R (0.012) |
16 (0.022) R:.640_:[..U]U-*---G[..G]:.630_:R (0.013) |
17 (0.013) R:.641_:[..G]G-----C[..C]:.629_:R (0.014) |
18 (0.016) R:.642_:[..C]C-----G[..G]:.628_:R (0.013) |
19 (0.016) R:.643_:[..C]C-----G[..G]:.627_:R (0.012) |
20 (0.010) R:.644_:[..A]Ax*---G[..G]:.626_:R (0.020) |
21 (0.014) R:.610_:[..G]G-*---U[..U]:.625_:R (0.019) |
22 (0.013) R:.611_:[..U]U-----A[..A]:.624_:R (0.014) |
23 (0.015) R:.612_:[..U]U-----A[..A]:.623_:R (0.013) |
24 (0.014) R:.613_:[PSU]P-*--xG[..G]:.622_:R (0.012) |
25 (0.014) R:.614_:[..A]A-**-xU[..U]:.608_:R (0.018) |
26 (0.008) R:.615_:[..A]Ax**+xU[..U]:.648_:R (0.014) x
27 (0.009) R:.618_:[..G]Gx---xC[..C]:.656_:R (0.017) +
Strand I Strand II Helix
Note: This structure contains 20[9] non-Watson-Crick base-pairs.
| На основании статьи сделаны выводы, что:
строки таблицы 1..7 соответсвуют акцепторному стеблю , 21...24 - D-стеблю , 15...19 - антикодоновому стеблю , 8...11 - T-стеблю . |
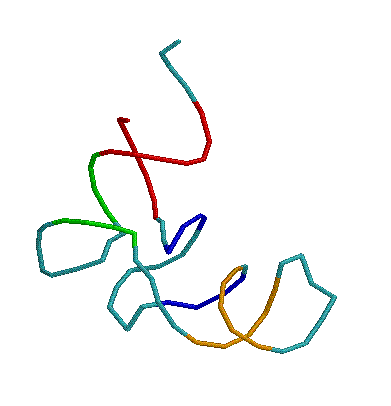
|
Текст скрипта:
restrict none
background white
restrict *R
backbone 100
color [55, 203, 206]
define ac_st *R and (601-607, 666-672)
define D_st *R and (610-613, 622-625)
define anti_st *R and (639-643, 627-631)
define T_st *R and (649-652, 662-665)
select ac_st
color red
select T_st
color green
select D_st
color blue
select anti_st
color [255, 165, 0]
echo red ac_st - is acceptor stem, green T_st - is T-stem,
blue D_st - is D-stem, orange anti_st - is anticodon stem
|
Структуру стеблевых дуплексов поддерживают 16 канонических и 4 неканоничексих пар оснваний, вот одна из них (U-*---G):

В последовательности РНК нет тимидина, нет его и в Т-петле.
В D-петеле есть дигидроуридины (на позициях 616, 619).
Вариабельная петля может находится на 644-648 нуклеотидах, думаю такое количество нуклеотидов недостаточно для образования петли.
* Изучаемая тРНК связывается с аспарагиновой кислотой, кодонами которой являются GAU, GAC. Антикодоновая петля состоит из 7 нуклеотидов
[32] 5'- PSU U G U C 1MG C -3' [38].
Исследование третичной структуры
С помощью программы analyze была выявлена возможность стекинг-взаимодействия между основаниями тРНК: stacking.pdb
В файле 1ASZ_old.out представлены данные о площади перекрывания 2-х последовательных пар азотистых оснований: фрагмент файла 1ASZ_old.out
Исследуем возможность стекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля: 665 U - конец T-стебля, 666 G - конец акцепторного стебля, исходя из записи 1ASZ_old.out :
7 (0.012) R:.607_:[..A]Ax----U[..U]:.666_:R (0.018) |
8 (0.012) R:.649_:[5MC]c-----G[..G]:.665_:R (0.014) |
взаимодествуют Ac/GU с нормерами 607,649/665,666, в файле stacking.pdb взаимодействующие нкулеотиды соответсвуют Section #0007 Ac/GU
Наложение оснований:
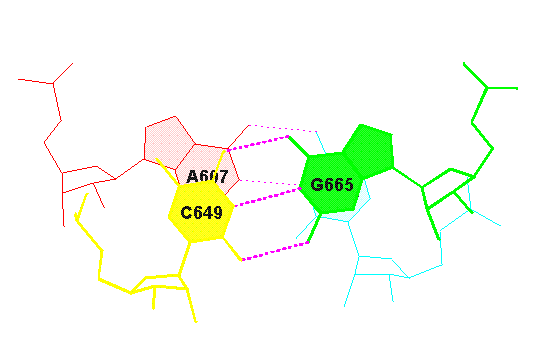
Инфрмацию о площади перекрывания представленных пар азотистых оснваний найдем в файле 1ASZ_old.out (step 7):
step i1-i2 i1-j2 j1-i2 j1-j2 sum
7 Ac/GU 4.44( 1.20) 0.00( 0.00) 0.00( 0.00) 6.48( 3.76) 10.92( 4.95)
Как видно из рисунка, нуклеотиды перекрываются хорошо, велика площадь перекрытия 10.92 ( при максимальной 13.82 и минимальной 0.00).
Те же нуклеотдиты в RasMol:
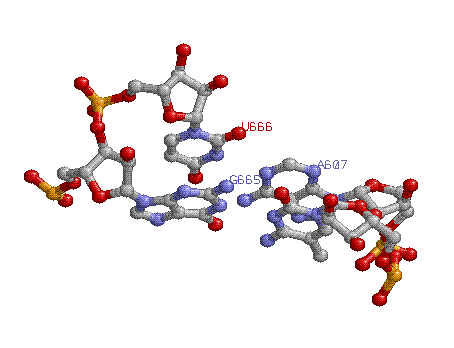
Есть ли дополнительные водородные связи между основаниями D- и Т-петель? Есть. D-петля (614-621 основания) и T-петля (653-664 основания), как видно из файла 1ASZ_old.out , соединяются водородными связями взаимодействия 1 канонической пары (G618 - C656):
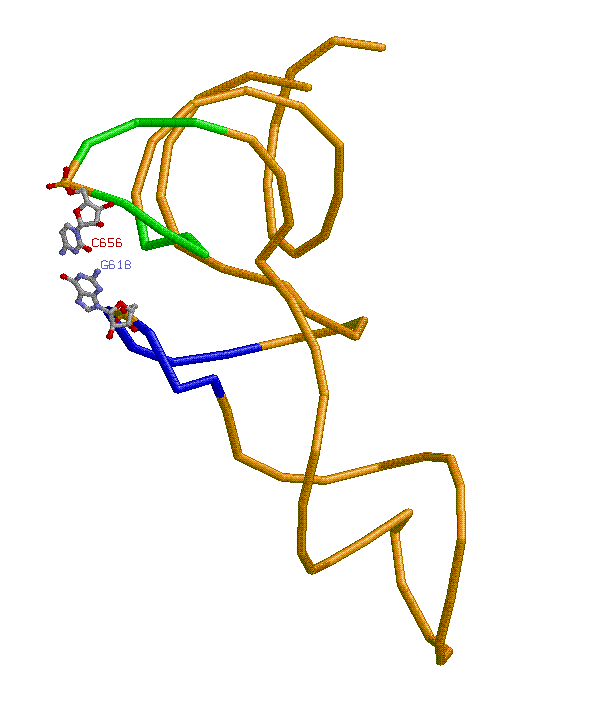
| зеленым отмечена T-петля, синим D-петля |
Предсказание вторичной структуры тРНК
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'- 601-607 - 3' 5'- 666-672 - 3' всего 7 пар нуклеотидов | 0 | 5'- 601-608 -3' 5'- 665-672 -3' всего 8 пар |
| D-стебель | 5'- 610-613 -3' 5'- 622-625 -3' всего 4 пары | 0 | 5'- 610-613 -3' 5'- 622-625 -3' всего 4 пары |
| T-стебель | 5'- 649-652 -3' 5'- 662-665 -3' всего 4 пары | 5'- 645-652 -3' 5'- 660-666 -3' всего 6 пар | 5'- 649-652 -3' 5'- 659-662 -3' всего 4 пары |
| Антикодоновый стебель | 5'- 627-631 -3' 5'- 639-643 -3' всего 5 пары | 0 | 5'- 627-631 -3' 5'- 639-643 -3' всего 5 пар |
| Общее число канонических пар нуклеотидов | 16 | 6 | 15 |
При работе с einverted , программа спросила назачаемый штраф за геп (gap penalty): снизил стандартный 12 до 6, минимальный вес выравнивания: стандартный 50. А также match score и mismatch score, чьи параметры я не менял ([3], [-4]). Если минимальный вес оставить стандартным = 50 - выравнивание вовсе не получается, при снижении до 30, 20 - тоже ничего не выходит. Первое выранивание появилось при минимльном весе 15, результатом его был T-стебель, но немного расширенный: 645_652 и 660_666
: Score 15: 7/7 (100%) matches, 1 gaps
45 gatcgggg 52
|| |||||
66 ct-gcccc 60
К тому же такой стебель содержит 1 геп (ему соответсвует выпетливание длиной в 1 нуклеотид).
Результаты программы mfold :
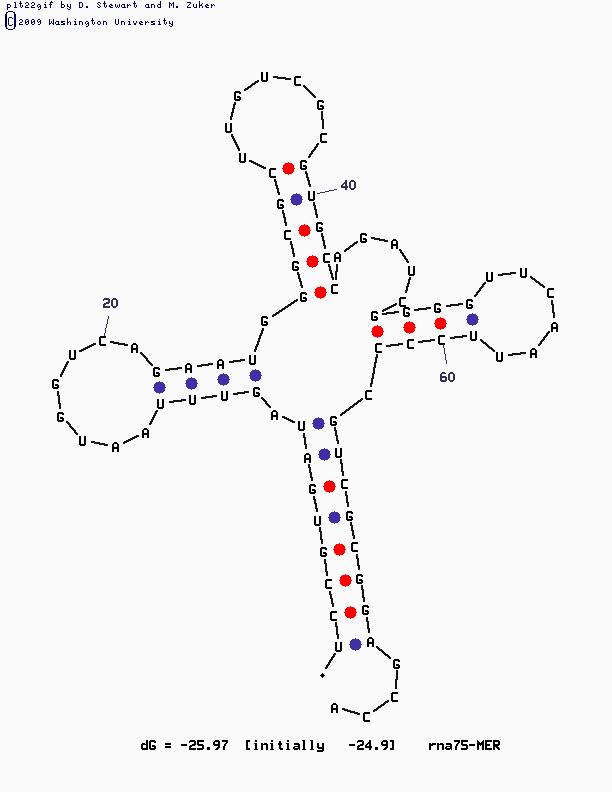
Это предсказание наиболее близко к реальной структуре (получено при значении параметра P=10)
Предсказанная структура схожа с реальной, но между ними есть различия: предсказанный акцепторный стебель длинее на 1 нкклеотид; в реальной T-петле содержится 9 нуклеотидов, в предсказаной же 6, причем в обоих случаях T-петля начинается с 661 нуклеотида.
Из рисунка видно расположение вариабельной петли на предсказанной структуре: 5'- 644-648 -3'.