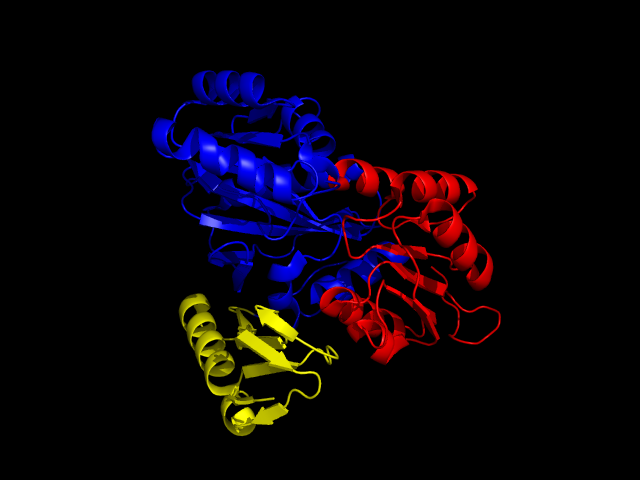
1) Классификация доменов записи 1EYZ PDB согласно SCOP (scop 1.75):
С помощью SCOP было найдено 3 домена в записи 1EYZ:
1) N-концевой домен белка PURT E.coli:
2) Домен 2 белка PURT E.coli:
3) C-концеовой домен белка PURT E.coli:
На рисунке изображена пространственная структура белка, красным покрашен N-концевой домен, синим - 2 домен, желтым - С-концевой домен:

Действительно, N-концевой домен содержит параллельный бета-лист, а 2-ой домен - 3 антипараллельных бета-листа, что соответсвует их классам.
2) Классификация доменов записи 1EYZ PDB согласно CATH :
С помощью CATH также было найдено 3 домена, также определенных одинаково для обеих цепей белка.
1) 1-ый домен:
1) 2-ой домен:
1) 3-ый домен:
На рисунке изображена пространственная структура белка, красным покрашен 1 домен, синим - 2 домен, желтым - 3 домен:

3) Различия между CATH и SCOP в определении доменов PURT E.col:
CATH и SCOP сильно отличаются в определении координат 2 и 3 доменов белка. По SCOP 2 домен является АТФ-связывающим, по CATH - оба домена связывают АТФ.
Как видно из записи 1EYZ, АТФ связывается со 2-ым доменом по SCOP, или же со 2-ым и 3-им доменом по CATH, находясь между ними
(на рисунке домены окрашены по CATH, AMPPNP - негидролизуемый аналог АТФ - изображен в проволочной модели и окрашен в зеленый цвет):
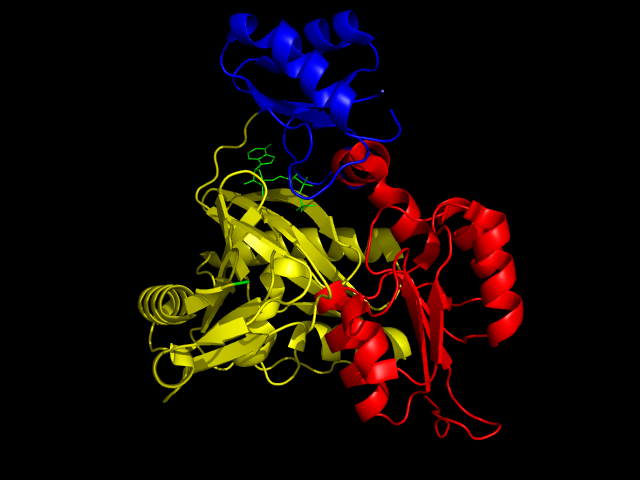
Таким образом, SCOP и CATH определили 2 и 3 домены одинаково с функциональной точки зрения, но по-разному со структурной.
SCOP и CATH примерно одинаково определили координаты 1-го (N-концевого) домен белка. При этом, согласно SCOP, домен имеет укладку PreATP-grasp domain, состоящую
из 3 слоев: a/b/a, возможно - укладка Россмана в рудиментарной форме.
Согласно CATH, домен имеет топологию Rossmann fold, имеющую схожую структуру с укладкой PreATP-grasp SCOP.
Функционально Rossmann fold связывает мононуклеотиды, но в данном случае AMPPNP связывается в другом месте структуры.
Таким образом, SCOP и CATH одинаково с структурной точки зрения определили 1-ый домен, но SCOP оказался точнее функционально, ведь 1-ый домен действительно распологается перед АТФ-связывающим доменом.
3) Пространственное выравнивание 2 доменов с одной укладкой по SCOP, но из разных суперсемейств:
Возьмем белок глутатионсинтетазу человека (запись PDB 2HGS) и альфа-субъединицу фермента, кэпирующего мРНК дрожжей (запись PDB 1P16).
Оба белка имеют укладку ATP-grasp по SCOP, но 1-ый относится к суперсемейству Glutathione synthetase ATP-binding domain-like (семейство Eukaryotic glutathione synthetase ATP-binding domain),
а 2-ой к - DNA ligase/mRNA capping enzyme, catalytic domain (семейство mRNA capping enzyme).
Интересующие нас домены занимают а.о. цепи A 1-го белка с 3 по 201 (2HGS PDB); цепи A 2-го белка с 1 по 245 (1P16 PDB).
С помощью PyMol, были созданы pdb файлы с этими остатками (h.pdb - фрагмент человеческого белка; y.pdb - фрагмент белка дрожжей).
На сервере PDBe Fold построено пространственное выравнивание фрагментов, отвечающих доменам (выравнивание последовательностей, полученное по структурному выравниванию, h_y.seq).
По выравниванию.seq, с помощью сервиса Geometrical core найдено геометрическое ядро с порогом 2 Å :
| Pos. | 1P16_A | 2HGS_A |
| 21 | ASP18 | ASP11 |
| 22 | GLU19 | LYS12 |
| 23 | GLU20 | GLN13 |
| 24 | GLU21 | GLN14 |
| 25 | THR22 | LEU15 |
| 26 | LYS23 | GLU16 |
| 27 | GLU24 | GLU17 |
| 28 | LEU25 | LEU18 |
| 29 | ARG26 | ALA19 |
| 30 | LEU27 | ARG20 |
| 32 | VAL29 | ALA22 |
| 33 | ALA30 | VAL23 |
| 34 | GLU31 | ASP24 |
| 35 | LEU32 | ARG25 |
| 36 | LEU33 | ALA26 |
| 37 | GLY34 | LEU27 |
| 133 | ASP61 | LEU123 |
| 134 | TYR62 | ASN124 |
| 135 | PHE63 | ARG125 |
| 136 | VAL64 | SER126 |
| 139 | LYS67 | MET129 |
| 290 | ALA218 | PRO138 |
| 292 | ASP220 | LEU140 |
| 293 | GLY221 | LYS141 |
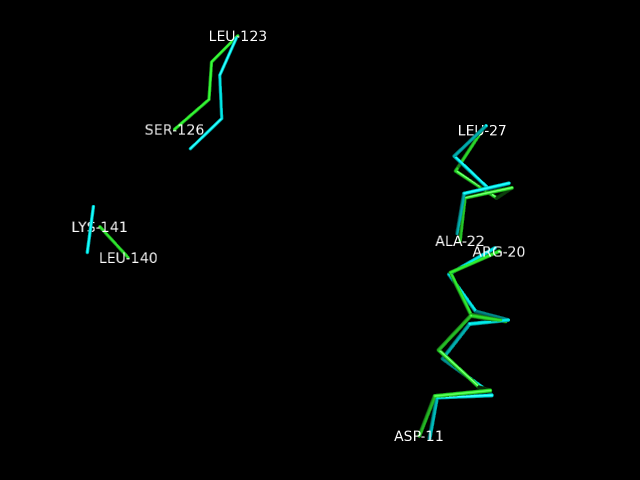
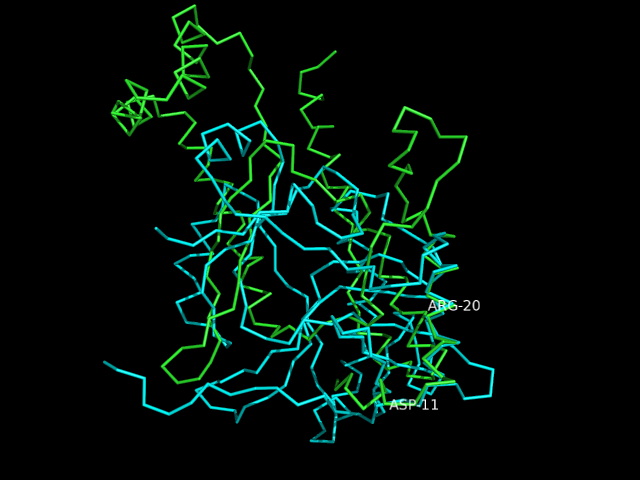
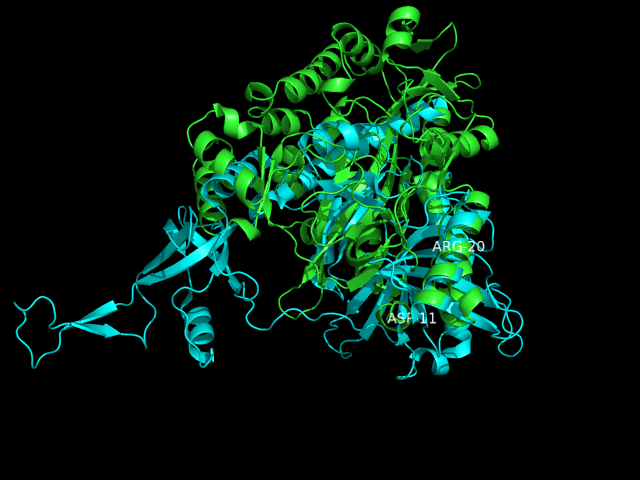
RMSD совмещения CA-атомов ядра не большое (1.629 Å), что говорит о том, что они хорошо совмещаются. Однако, мы видим, что домены совмещаются значительно хуже, значит домены, относящиеся к разным суперсемействам, хоть и с одной укладкой, сильно отличаются (по крайней мере на данном примере). Еще хуже совмещаются полные структуры цепей A, что неудивительно, ведь цепи A больше входящих в них доменов.