Определим вторичнцю структуру белка, представленного в записи 1EYZ PDB с помощью программы DSSP (выдача программы - файл 1EYZ.dssp ; Excel файл ).
Поскольку в записи 1EYZ представлена пространственная структура 2 цепей (A и B) белка PURT_ECOLI, последовательности которых на 100% идентичны, можно ожидать, что и вторичная структура этих цепей будет одинакова, однако программа DSSP определила ее несколько по-разному для A и B (см Excel файл ). Всего для цепи A определено 22 бета-тяжа и 11 альфа-спиралей, для цепи B - 21 бета-тяж и 12 альфа-спиралей.
Согласно записи 1EYZ PDB, авторы опредилили вторичную структуру белка отлично от выдачи DSSP (см Exel файл сравнения описания вторичной структуры ).
Можно заметить, что все альфа-спирали, определенные DSSP также определены автормаи записи, однако каждая такая альфа-спираль DSSP короче не 2 аминокислоты по сравнению с соответсвующей альфа-спиралью по мнению авторов (например, если согласно DSSP альфа-спираль занимает положение с 22 по 32 аминокислотный остаток, то согласно PDB - с 21 по 33). По данным PDB обе цепи белка содержат 15 альфа-спиралей.
Бета-тяжи в PDB и согласно DSSP определены одинаково.
Согласно выдаче DSSP, положительное значение торсионного угла φ имеют остатки лейцин 8 цепи A, глицин 19 цепи A, глицин 35 цепи A, аспарагиновая кислота 46 цепи A, лизин 75 цепи A, глицин 98 цепи A, глутамин 126 цепи A, глицин 149 цепи А, серин 161 цепи A, глицин 162 цепи A, глицин 164 цепи A, глицин 185 цепи A, аланин 187 цепи A, глицин 214 цепи A, аспарагиновая кислота 227 цепи A, глицин 228 цепи A, глицин 259 цепи A, глицин 260 цепи A, глицин 272 цепи A, глицин 309 цепи A, глицин 313 цепи A, глицин 319 цепи A, аспарагин 339 цепи A, глутаминовая кислота 347 цепи A, глицн 354 цепи A, глицин 360 цепи A и такие же остатки цепи B. Как видно особенно часто среди таких остатков встречается глицин.

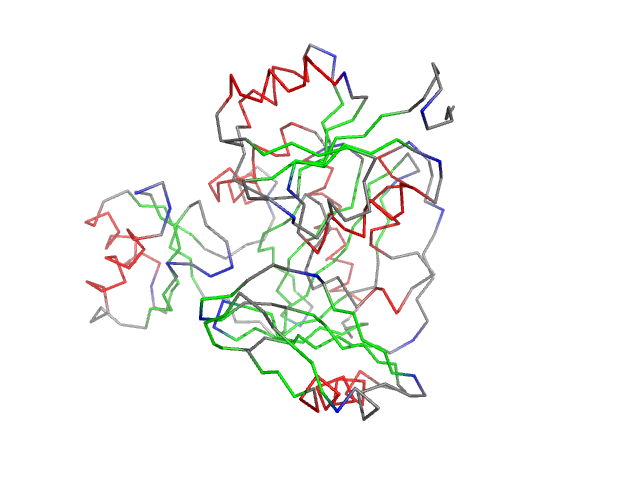
На рисунке изображена остовная модель цепи A белка PURT_ECOLI; красным отмечены альфа-спирали, зеленым - бета-тяжи, синим - остатки с положительным торсионным углом φ (согласно DSSP).
Остатки с положительным значением φ часто встречаются там, где полипептидная цепь делает поворот.
С помощью программы HBPlus не удалось определить водородные связи в записи 1EYZ.pdb, поскольку в ней содержится слишком много атомов. Тогда с помощью RasMol была выделена цепь A белка, а также все небелковые атомы, распологающиеся к ней ближе, чем на 5 Å, и сохранены в отделный PDB файл. После его обработки программой HBPlus, был получен файл с водородными связями.
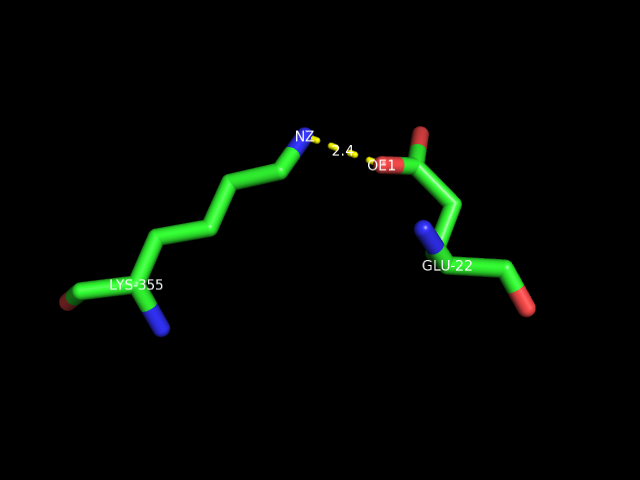
Пример водородной связи между боковыми цепями аминокислотных остатков
Водородная связь между атомом Nz лизина 355 цепи A и атомом OE1 глутаминовоя кислоты 22 цепи A:
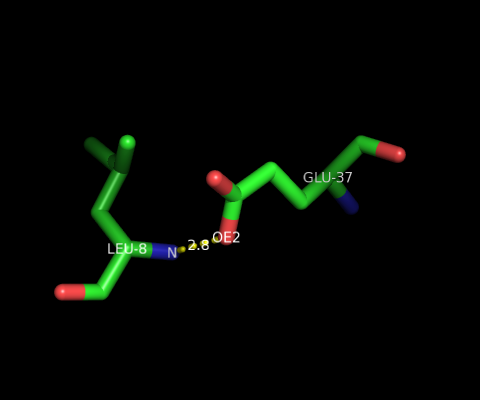
Пример водородной связи между боковой цепью одного остатка и остовным атомом другого:
Водородная связь между остовным атомом N лейцина 8 цепи A и атомом боковой цепи OE2 остатка глутаминовой кислоты 37 цепи A:
Пример водородной связи между белком и лигандом:
Водородная связь между атомом NH1 боковой группы аргинина 114 цепи A и атомом O2B лиганда ANP (в поле HETATM эта молекула лиганда имеет номер 400):

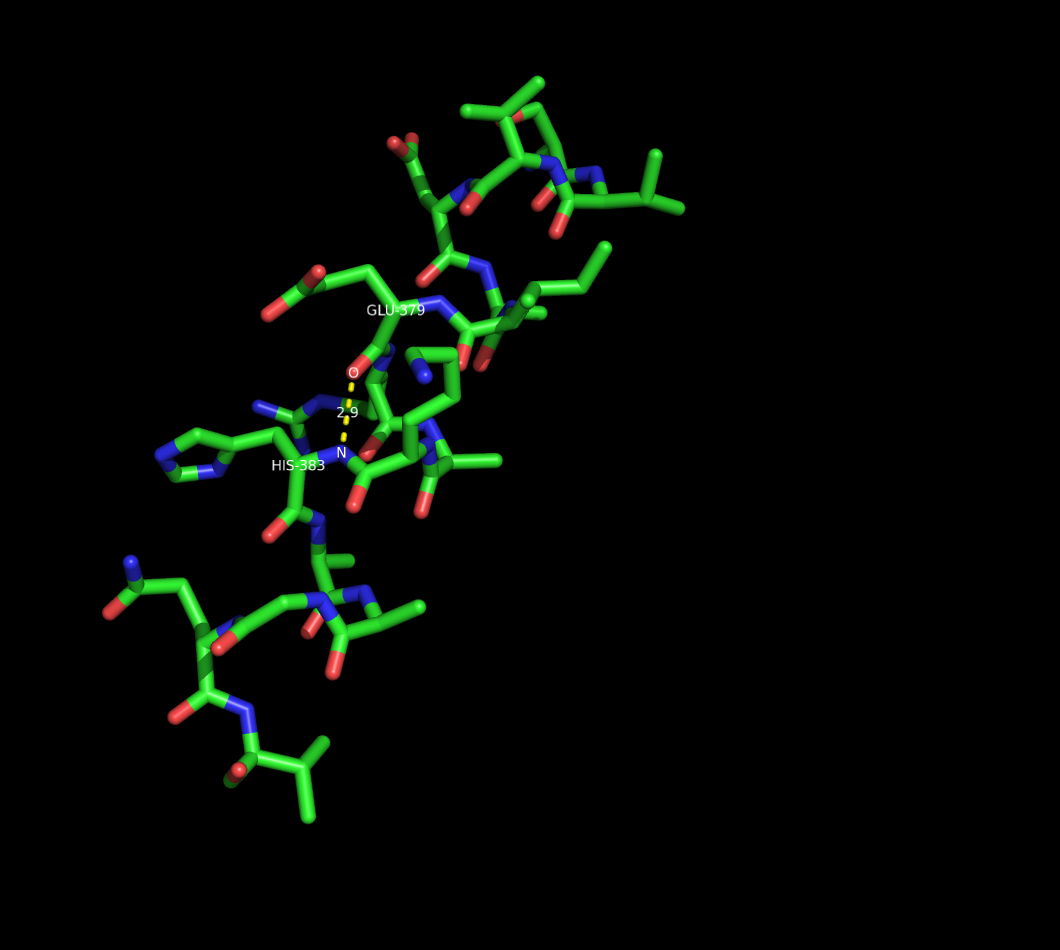
Пример водородной связи, участвующей в поддержании вторичной структуры:
Водородная связь между остовными атомами O и N остатков глутаминовой кислоты 379 и гистидина 383 цепи A участвует в поддержании альфа-спирали 373-388, на рисунке изображена альфа-спираль и водородная связь между HIS-388 и GLU-379:
Пример водяного мостика
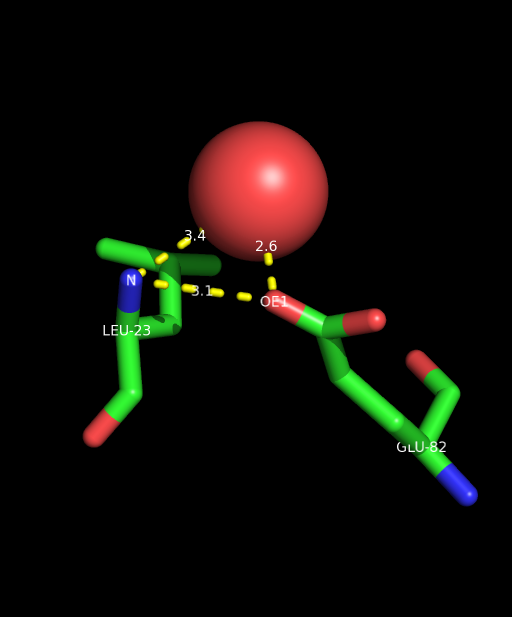
На рисунке изображены водородные связи между атомом N лейцина 23 цепи A и молекулой воды (номер A-1298) и между атомом OE1 глутаминовой кислоты 82 цепи A и той же молекулой воды. Хотя атомы N и OE1 располагаются и так близко друг к другу (3,1 Å, между ними формируется водородная связь) и угол (N, HOH, OE1) острый, тем не менее, на мой взгляд, молекула воды дополнительно укрепляет связь между N и OE1.
Для примеров использовались данные HBPlus (в том же порядке, как примеры):
<---DONOR---> <-ACCEPTOR--> atom ^
c i cat <-CA-CA-> ^ H-A-AA ^ H-
h n atom resd res DA || num DHA H-A angle D-A-AA Bond
n s type num typ dist DA aas dist angle dist angle num
A0355-LYS NZ A0022-GLU OE1 2.44 SS -1 8.43 131.8 1.65 116.3 122.3 60
A0008-LEU N A0037-GLU OE2 2.83 MS 29 6.48 161.2 1.86 128.4 124.4 20
A0114-ARG NH1 A0400-ANP O2B 3.23 SH -2 -1.00 140.9 2.39 -1.0 -1.0 257
A0383-HIS N A0379-GLU O 2.88 MM 4 5.83 161.5 1.91 142.9 148.9 866
A0023-LEU N A1298-HOH O 3.43 MH -2 -1.00 165.3 2.45 -1.0 -1.0 64
A1298-HOH O A0082-GLU OE1 2.62 HS -2 -1.00 -1.0 -1.00 -1.0 120.4 189