1) Программа PROCHECK
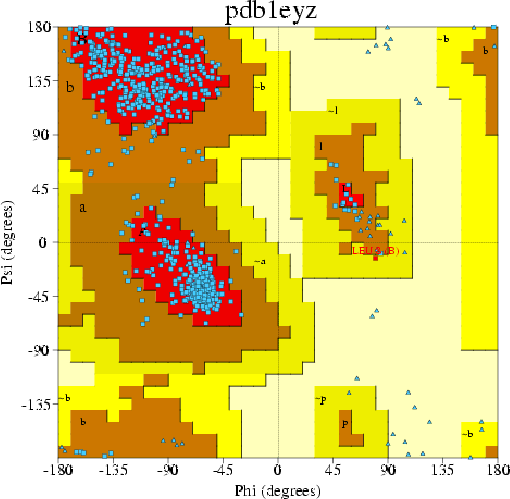
Выдача программы PROCHECK (карта Рамачандрана) для записи 1EYZ PDB:

остатков (отличных от глицина и пролина), попавших в предпочтительные области: 90.6%
попавших в допустимые области: 9.2%
в нежелательные области: 0.1%
в недопустимые области: 0.0%
Судя по карте Рамачандрана, модели, прелставленной в записи 1EYZ, можно доверять, поскольку большинство остатков имеют предпочтительные торсионные углы.
2) Значения RSR остатков структуры 2YWR
На сервере EDS нет информации для структуры 1EYZ, поэтому для выполнения задания использовалась запись 2YWR PDB - структура схожего фермента бактерии Aquifex aeolicus.
Согласно EDS, 10 остатков (5%) имеют большой Z-score по RSR. Один из этих остатков - аргинин 71 (RSR=0.242; коэффициента корреляции для электронной плотности = 0.668; Z-score = 2.150662). Изображение модели остатка и электронной плотности вокруг него (поверхность уровня 1 изображена желтыми линиями, уровня 0.5 - белыми):
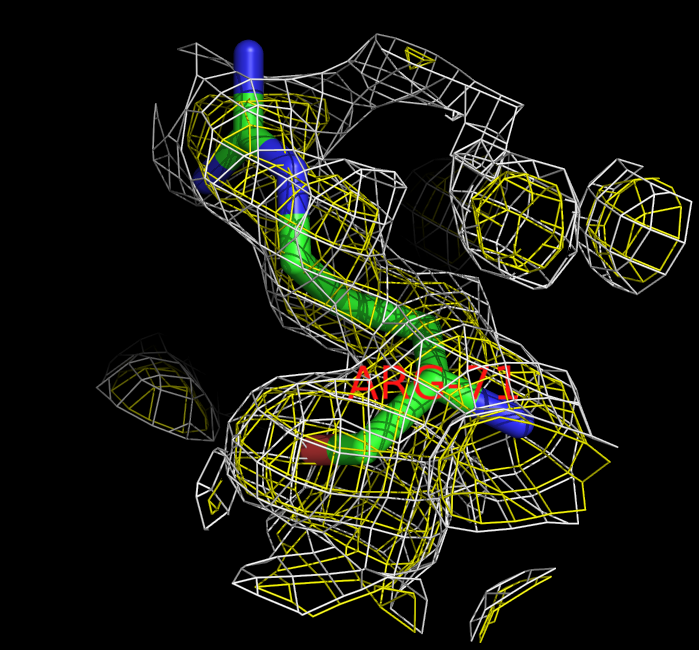
Как видно из рисунка атомы этого остатка не очень хорошо вписаны в электронную плотность, так центры атомов O, NH1 и NH2 вылезли за пределы поверхности уровня 1, а центры атомов NE и CD - на поверхности уровня 1. Вероятно поэтому этот остаток имеет слишком высокое значение RSR (>0,1), низкое значение коэффициента корреляции между экспериментальной электронной плотностью и плотностью модели (<0,9), высокое значение Z-score (>2).
3) Показатели оптимизированной структуры 2YWR согласно EDS
| Из записи PDB | Вычисленные значения | После оптимизации | После полной оптимизации | |
| R | 0.2040 | 0.2052 | 0.1703 | 0.1628 |
| R-free | 0.2190 | 0.2185 | 0.2165 | 0.2121 |
| σR-free | 0.0036 | 0.0035 | 0.0035 | |
| R-free Z-score | 9.36 | -2.06 | -3.43 |
После полной оптимизации значение R уменьшилось (улучшилось), так же незначительно уменьшился R-free (улучшился), однако разница между R и R-free возросла, что настораживает в отношении переоптимизации модели. О переоптимизации модели так же говорит отрицательный R-free Z-score.
| Original PDB entry | Conservatively optimised | Fully optimised | |
| 1st generation packing quality1 | 0.101 | 0.211 | 0.182 |
| 2nd generation packing quality1 | -0.855 | -0.543 | -0.402 |
| Ramachandran plot appearance1 | 0.273 | 0.547 | 0.553 |
| Chi-1/Chi-2 rotamer normality1 | 0.239 | 0.610 | 1.067 |
| Backbone conformation1 | -0.512 | -0.623 | -0.621 |
| Bond length RMS Z-score2 | 0.292 | 0.649 | 0.634 |
| Bond angle RMS Z-score2 | 0.750 | 0.764 | 0.748 |
| Total number of bumps3 | 41 | 29 | 22 |
| Unsatisfied H-bond donors/acceptors3 | 8 | 10 | 11 |
После полной оптимизации улучшились показатели 1st generation packing quality и 2nd generation packing quality. Так же улучшились значения торсионных углов на карте Рамачандрана, значения торсионных углов ротамеров. Несколько ухудшилась конформация остова, показатели длины связей и валентных углов ухудшились, но остались в пределах нормы. Уменьшилось число слишком близко расположенных атомов. Увеличилось число неудовлетворенных доноров/акцепторов водородных связей (параметр ухудшился).
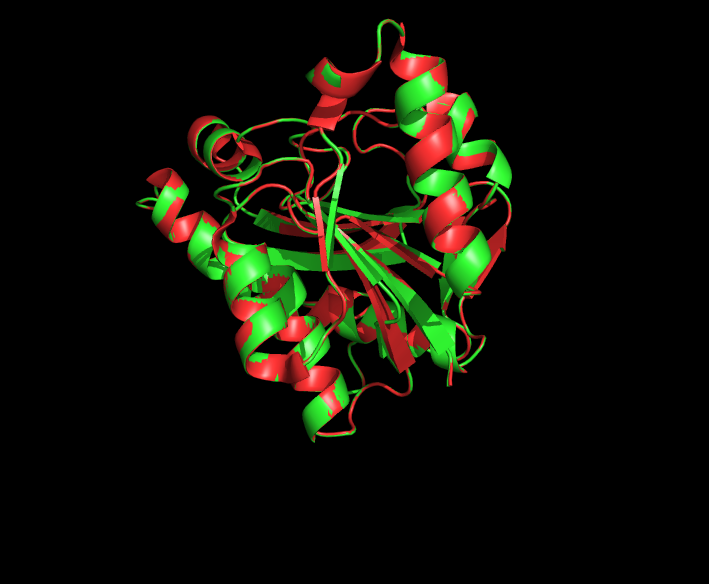
4) Полностью оптимизированная структура 2YWR
Визуально оптимизированная структура не сильно отличается от начальной. На рисунке - наложенные друг на друга начальная структура (красная) и полностью оптимизированная (зеленная), на переднем плане заметны различия в ориентации бета-тяжей.
Посмотрим как вписывается в электронную плотность остаток 71 оптимизированной модели:

На рисунке толстыми линиями изображен аргинин 71 оптимизированной модели, тонкими - аргинин 71 начальной структуры. Остаток оптимизированной структуры вписывается в электронную плотность лучше, так центр атома O, NE и CD располагается внутри поверхности уровня 1. Однако атомы NH1 и NH2 по-прежнему вылезают не только за пределы поверхности уровня 1, но так же и за пределы поверхности уровня 0.5.
Выдача WHAT_CHECK для структуры 2YWR PDB
В выдаче WHAT_CHEK для 2YWR содержится информация о молекулярном весе полимерных цепей, объеме элементраной ячейки и количестве молекул в ней; информацию о различных административных проблемах структуры (аминокислотные остатки в лигандах, ковалентные связи между различными полимерными цепями, остатки без боковой группы, перекрывающиеся группы); карта Рамачандрана; информация о вторичной структуре белка; неправильные валентные углы, связи, геометрия молекул; информация о B-факторе.....
WHAT IF позволяет отследить ошибки в структуре и предотвратить использование недостоверных данных в исследовании.