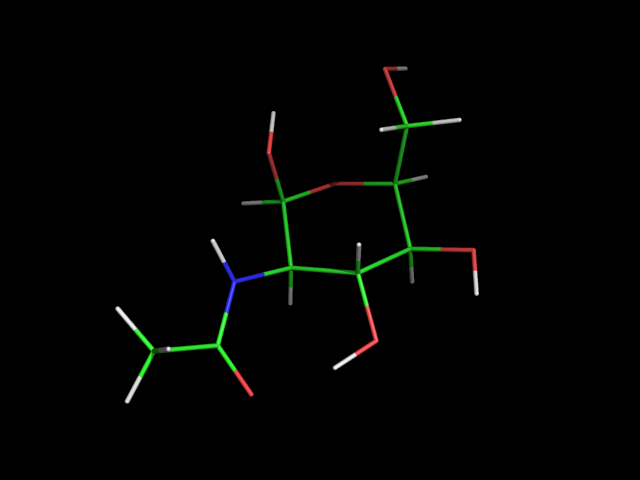
1,2. С помощью obgen построим 3D структуру сахара NAG (N-acetyl-d-glucosamine):
obgen nag.smi > nag.mol babel -imol nag.mol -opdb nag.pdb

3. С помощью скрипта prepare_ligand4.py из пакета Autodock tools был создан pdbqt-файл лиганда NAG nag.pdbqt :
prepare_ligand4.py -l nag.pdb
4. Также создадим pdbqt-файл для структуры лизоцима человека (используем 5-ую модель из предыдущего практикума) model5.pdbqt :
prepare_ligand4.py -l model5.pdb
5. Для докинга необходимо указать область структуры белка, в которой будет происходить поиск места для связывания. Удобно его задать как куб (с длиной ребра - 25 ангстрем) с неким центором. Координаты центра определим из модели комплекса, построенной на прошлом занятии. Выберем атом сахара, находящийся в центре сайта связывания (например, атом C1B), и извлечемиз текста pdb-файла model5.pdb его координаты (42.970 42.880 27.195). Создадим по этим данным файл vina.cfg .
Изображение лиганда и белка, атом C1B лиганда подписан:

6. Проведем первый докинг:
vina --config vina.cfg --receptor model5.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
В результате докинга были получены файлы nag_prot.pdbqt и nag_prot.log .
7. По данным nag_prot.log такова энергия 3-ёх лучших расположений лиганда и геометрическая разница между ними:
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -5.9 | 0 |
| 2 | -5.6 | 1.564 |
| 3 | -5.4 | 3.058 |
Файлы nag_prot.pdbqt и model5.pdbqt были загружены в PyMOL. Все состояния на одной картинке изображены ниже:
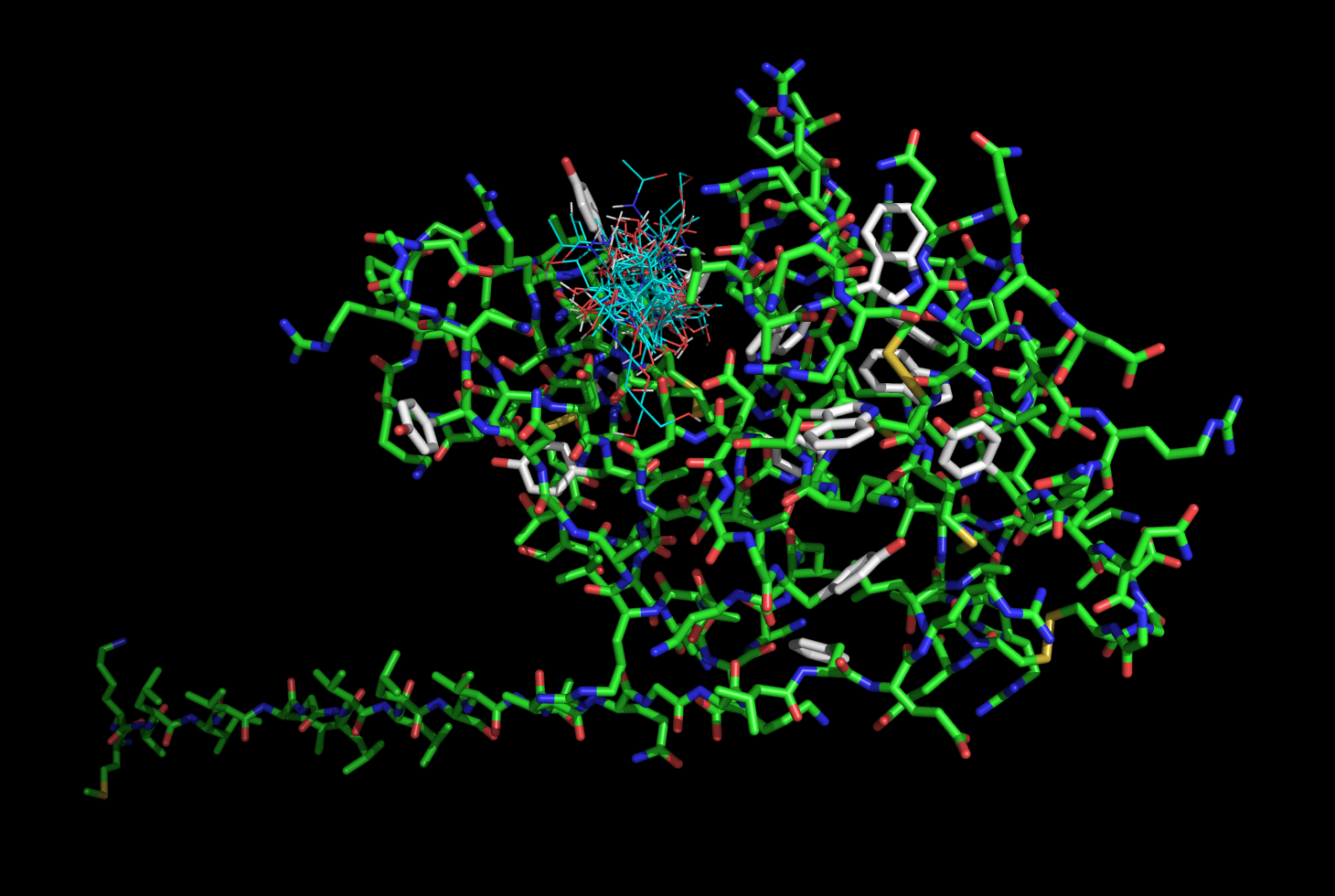
В некоторых состояниях молекула лиганда довольно сильно выходит из места связывания.
8. Теперь проведем докинг, рассматривая подвижность некоторых боковых радикалов белка. Сначала разобьем белок на две части: подвижную и неподвижную. Для подвижной части выберем 3 аминокислоты, которые мы использовали в прошлом задании для позиционирования лиганда (Trp-82, Asp-120 и Glu-53). Для создания pdbqt-файла воспользуемся скриптом prepare_flexreceptor4.py:
prepare_flexreceptor4.py -r model5.pdbqt -s GLU1_ASN5_ASP13В результате были получены файлы model5_flex.pdbqt и model5_rigid.pdbqt . Теперь проведем докинг:
vina --config vina.cfg --receptor model5_rigid.pdbqt --flex model5_flex.pdbqt --ligand nag.pdbqt --out vina_prot_flex.pdbqt --log vina_prot_flex.logВ результате докинга были получены файлы vina_prot_flex.pdbqt и vina_prot_flex.log .
В этот раз докинг проходил дольше.
9,10. Просмотрим файл vina_prot_flex.log . Энергии трех лучших расположений и геометрическая разница между ними представлена в таблице:
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -5.1 | 0 |
| 2 | -4.9 | 1.152 |
| 3 | -4.8 | 1.541 |
Файлы model5_rigid.pdbqt и vina_prot_flex.pdbqt были загружены в PyMOL. Все состояния на одной картинке изображены ниже:
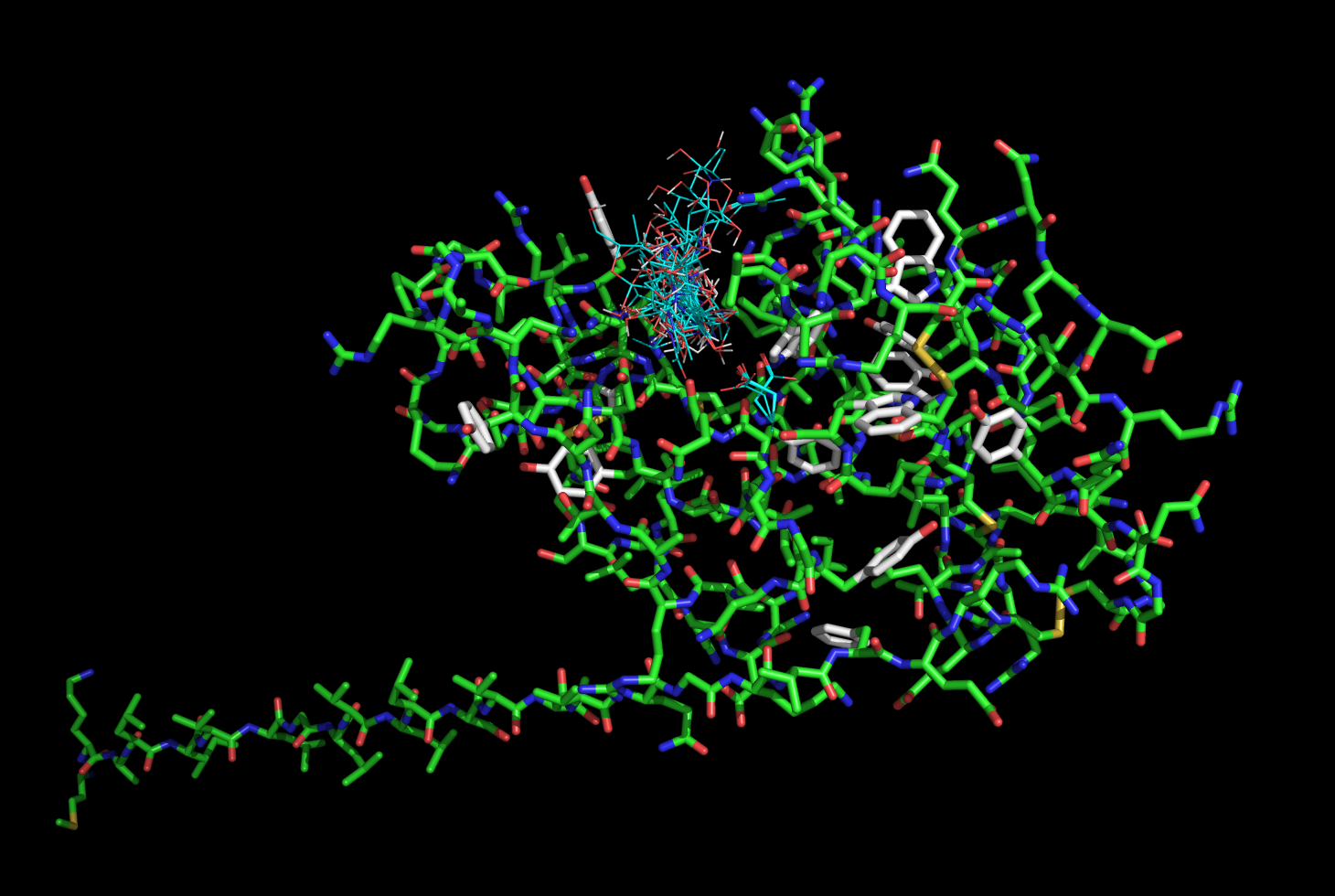
В этом случа подвижными стали также 3 выделенные а.о. Движения лиганда изменились, теперь он стал выходить в другую сторону из места связывания.
Ни в том, ни в другом случае не удалось расположить лиганд как в полученной модели №5.
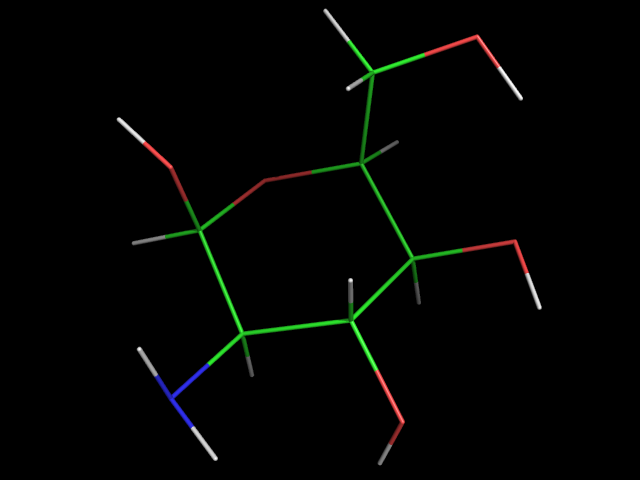
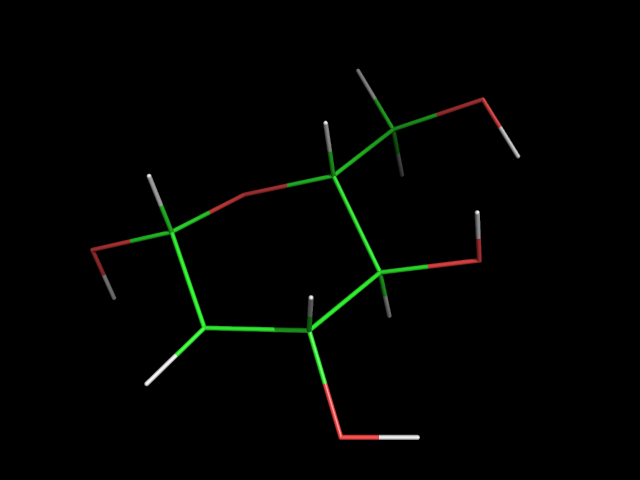
11. NAG содержит в себе СH3C(=O)NH группу. Создим 3 лиганда где метильный радикал этой группы будет заменён на OH ( nag2.smi , nag2.pdb ), NH2 ( nag3.smi , nag3.pdb ), H ( nag4.smi , nag4.pdb ).
| nag2 | nag3 | nag4 |
| 
| 
|
В результате соответствующих моделирований были получены файлы: nag_prot_2.pdbqt и nag_prot_2.log ; nag_prot_3.pdbqt и nag_prot_3.log ; nag_prot_4.pdbqt и nag_prot_4.log .
Таблица трех лучших расположений для первого лиганда:
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -5.0 | 0 |
| 2 | -5.0 | 1.992 |
| 3 | -4.9 | 2.095 |
Таблица трех лучших расположений для второго лиганда:
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -5.0 | 0 |
| 2 | -5.0 | 3.060 |
| 3 | -4.8 | 1.970 |
Таблица трех лучших расположений для третьего лиганда:
| Расположение | Энергия (ккал/моль) | Геометрическая разница с лучшей моделью (rmsd l.b.) |
| 1 | -4.8 | 0 |
| 2 | -4.8 | 2.308 |
| 3 | -4.6 | 2.244 |
Как видно из энергий, модифицированные лиганды связываются хуже, чем NAG. Из модифицированных лигандов хуже остальных связывается nag4.