1. Подготовим файл координат и файл топологии, используя файл box_38.gro с 38 молекулами этана (плотность как в жидкой фазе).
make_ndx -f box_38.gro -o 1.ndx
Создадим gro файл с одной молекулой и зададим ячейку
editconf -f box_38.gro -o et1.gro -n 1.ndx editconf -f et1.gro -o et.gro -d 2 -c
В результате получим файл et.gro . Исправим файл et.top из прошлого задания, сократив число молекул до 1 ( et.top ).
2. 5 файлов с разными параметрами контроля температуры:
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
3. Сначала построим входные файлы для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd список mdp файлов
4. Для каждого из 5 tpr файлов запустим mdrun:
mdrun -deffnm et_${i} -v -nt 1
5. Теперь переходим к анализу результатов. Начнем с визуального анализа. Для каждой из 5 систем проведем конвертацию в pdb и просмотрим в PyMol:
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
6. Сравним потенциальную энергию связи и кинетическую энергию для каждой из 5 систем:
g_energy -f et_${i}.edr -o et_${i}_en.xvg
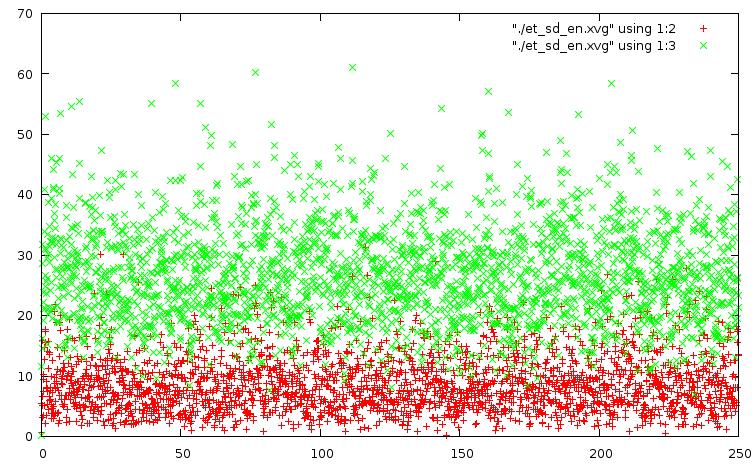
Построим графики изменения энергий. Для этого воспользуемся Gnuplot:
set datafile commentschars "#@&" plot "./et_be_en.xvg" using 1:2, "./et_be_en.xvg" using 1:3 .... plot "./et_sd_en.xvg" using 1:2, "./et_sd_en.xvg" using 1:3
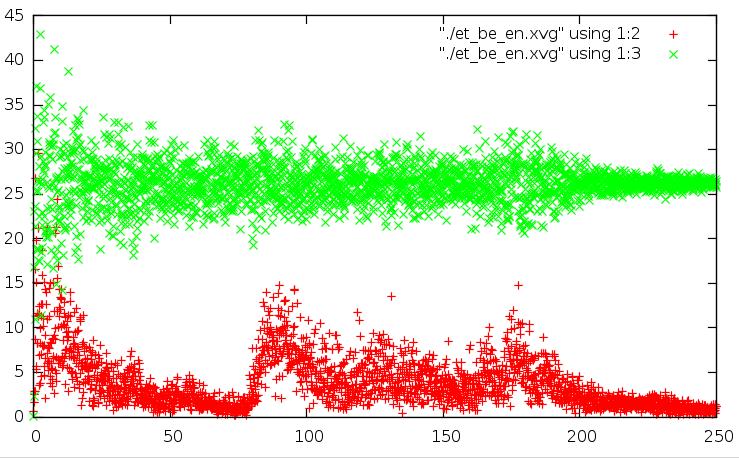
На графиках зеленым показана кинетическая энергия, красным - потенциальная. По оси x - время в пико-секундах.
График для метода Берендсена:

График для метода "Velocity rescale":
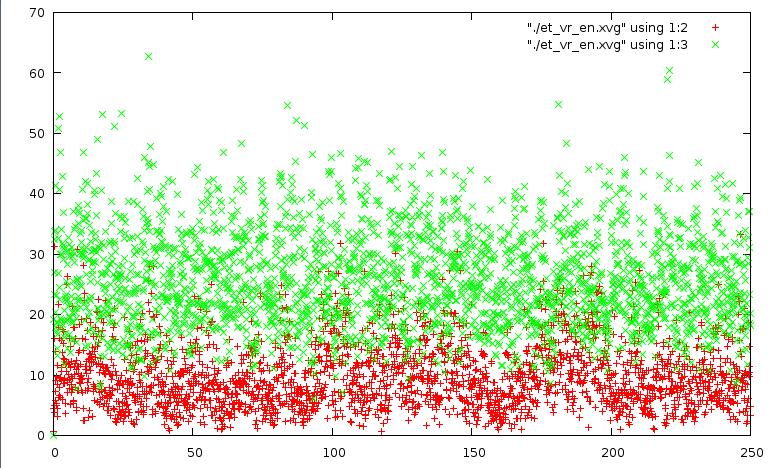
График для метода Нуза-Хувера:
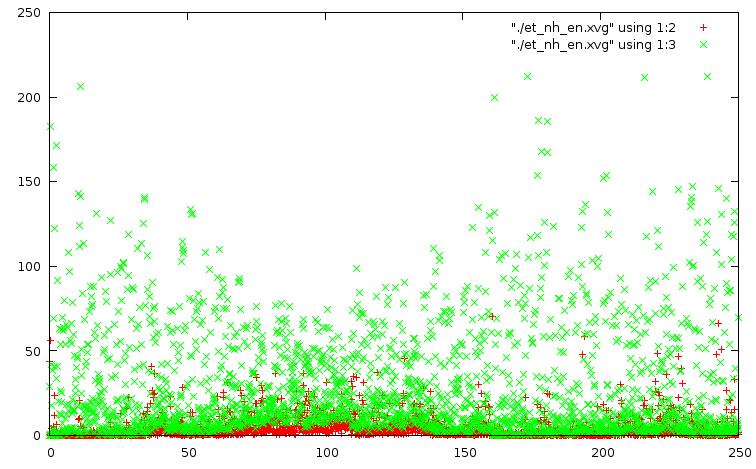
График для метода Андерсена:
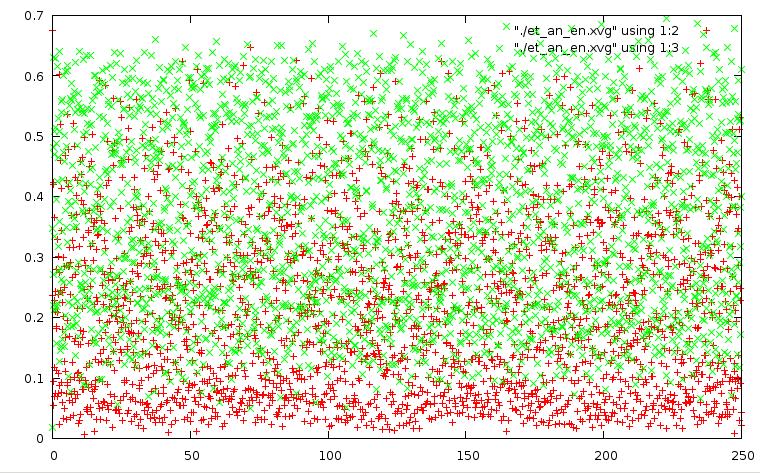
График для метода стохастической молекулярной динамики:
7. Рассмотрим распределение длинны связи С-С за время моделирования. Сначала создадим индекс файл с одной связью. В текстовом редакторе создайте файл b.ndx со следующим содержимым:
[ b ] 1 2
И запустим утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Графики изменений C-C связей за время моделирования:
График для метода Берендсена:
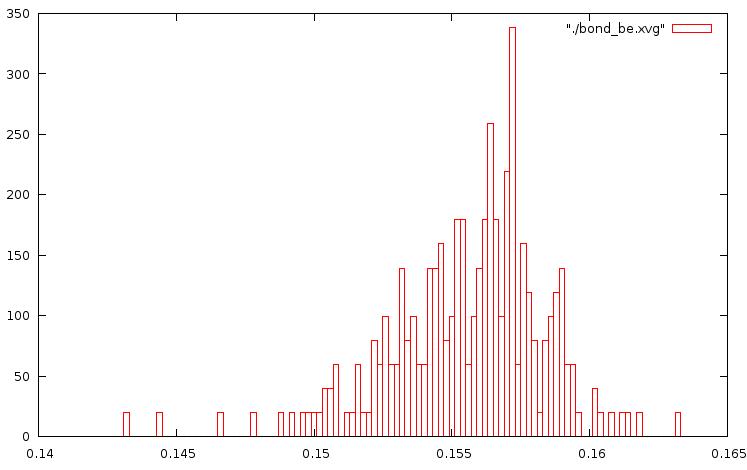
График для метода "Velocity rescale":
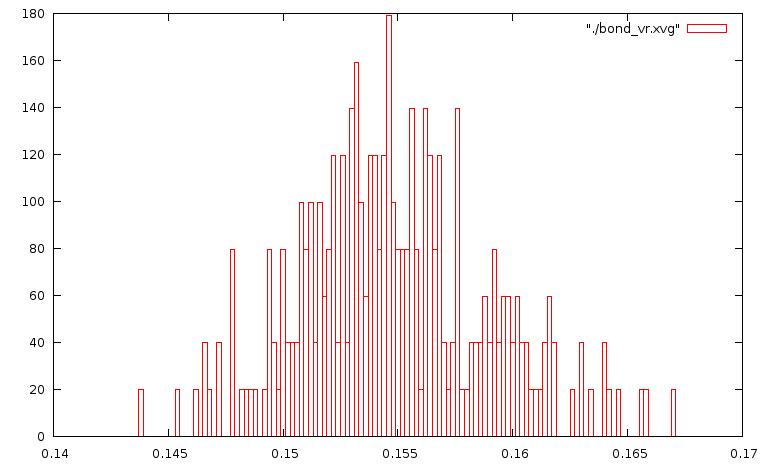
График для метода Нуза-Хувера:
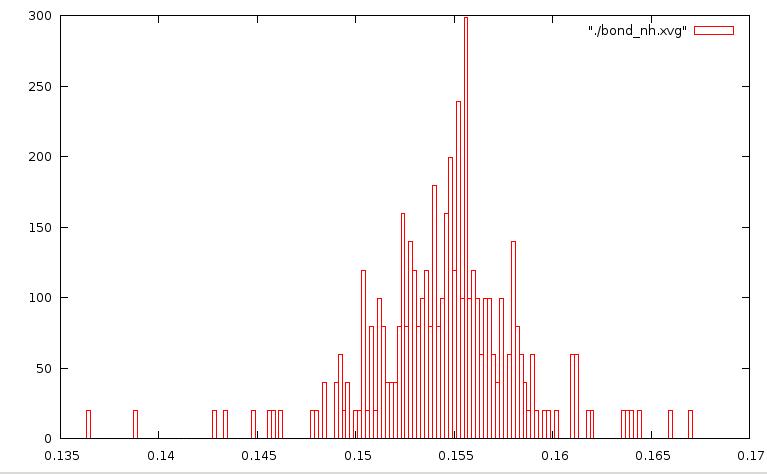
График для метода Андерсена:
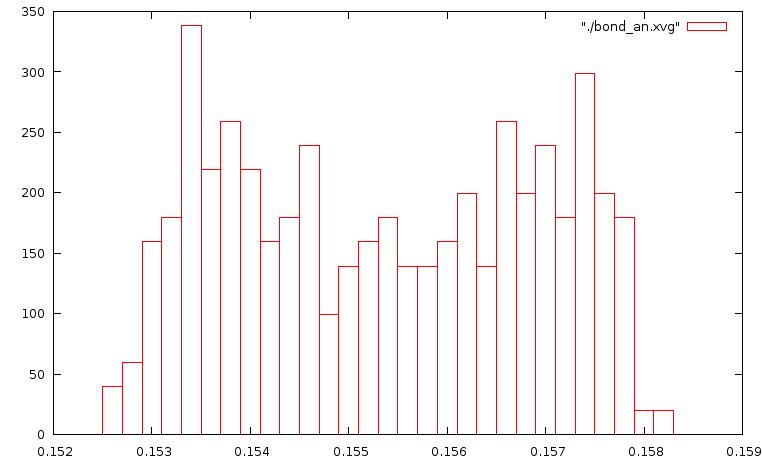
График для метода стохастической молекулярной динамики:
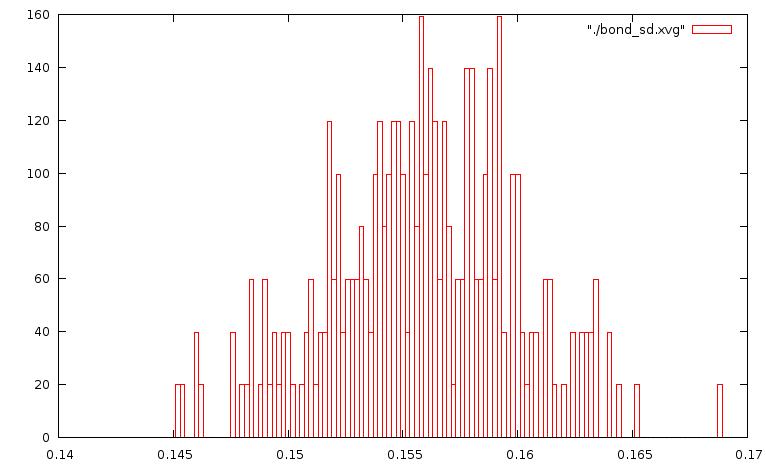
8. Сравним распределение длин связей с распределением Больцмана:
Распределение Больцмана:
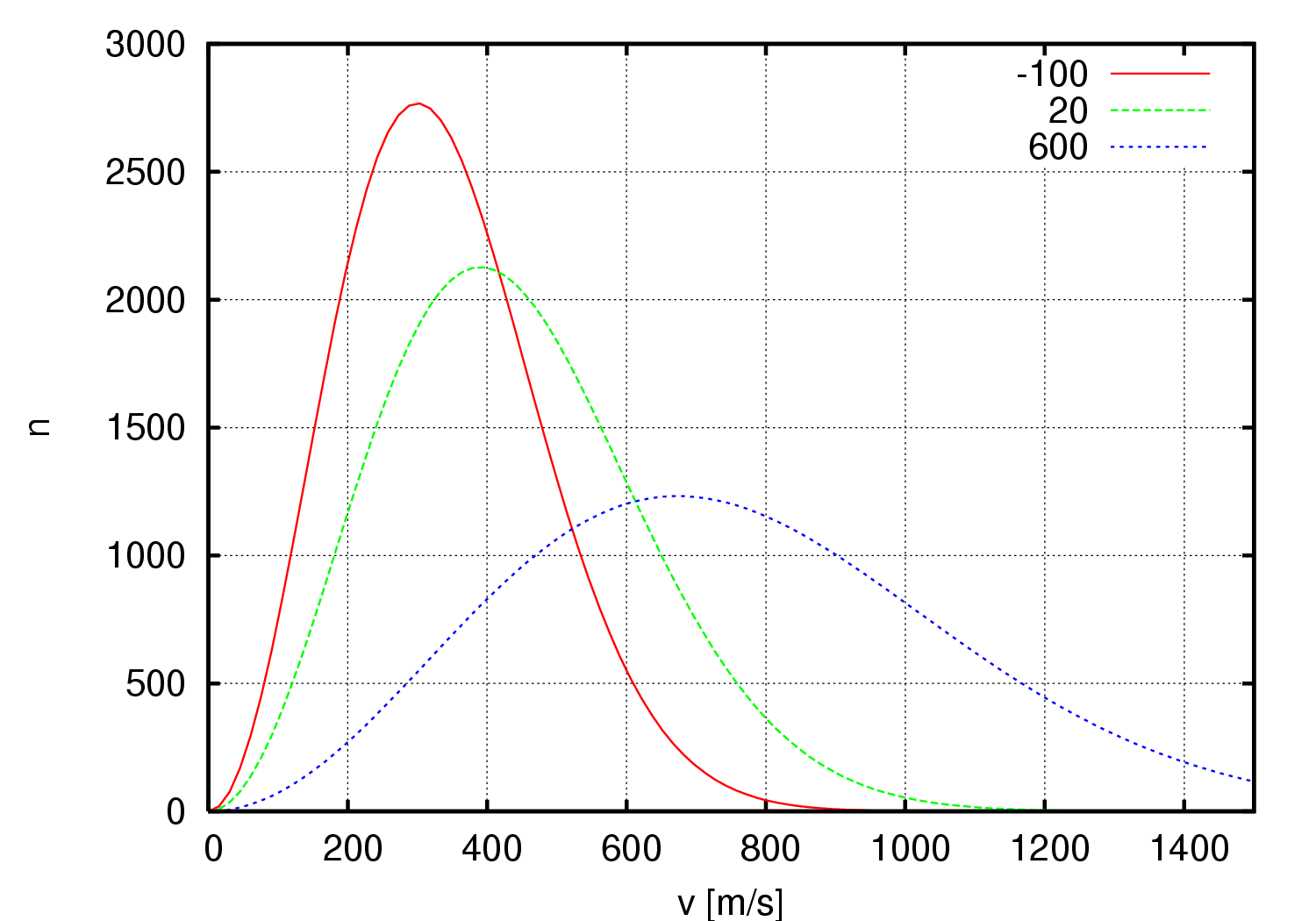
Можелирование проводилось при температуре 300 K, значит распределение длин связей должно быть близко к зеленой пунктрирной линии.
С этой точки зрения метод Андерсона хуже остальных методов. Похожее распределение имеют методы Velocity rescale и стохастической молекулярной динамики, однако учитывая странное поведение молеклы при моделировании методом стохастической молекулярной динамики, метод Velocity rescale вероятно лучший.