Цель работы построить модель комплекса лизоцима человека с лигандом на основании структуры 1LMP.pdb лизоцима форели с лигандом.
1. Построим выравнивание последовательностей белка рыбы ( 1lmp.fasta взята с сайта pdbsum) и человека ( P61626.fasta взята из uniprot) с помощью Clastal . Полученное выравнивание сохраним в формате pir ( aln.pir ).
2. Модифицируем файл выравнивания: последовательность лизоцима человека назовем ">P1;seq", последовательность лизоцима форели - ">P1;1lmp", после имени последовательности моделируемого белка добавим строчку "sequence:ХХХХХ::::::: 0.00: 0.00", после имени последовательности белка-образца - "structureX:1lmp_now.ent:1 :A: 132 :A:undefined:undefined:-1.00:-1.00", в конце каждой последовательности добавим символы "/.". После каждой послежовательности оставим также строчку с "*".
3. Модификация файла со структурой: удалим всю воду из структуры (в текстовом редакторе) всем атомам лиганда присвойте один и тот же номер "остатка" (MODELLER считает, что один лиганд = один остаток) и модифицируйте имена атомов каждого остатка, добавив в конец буквы A, B, C.
Сохраним файл в 1lmp_now.ent .
4,5. Используем управляющий скрипт lysc_human.py .
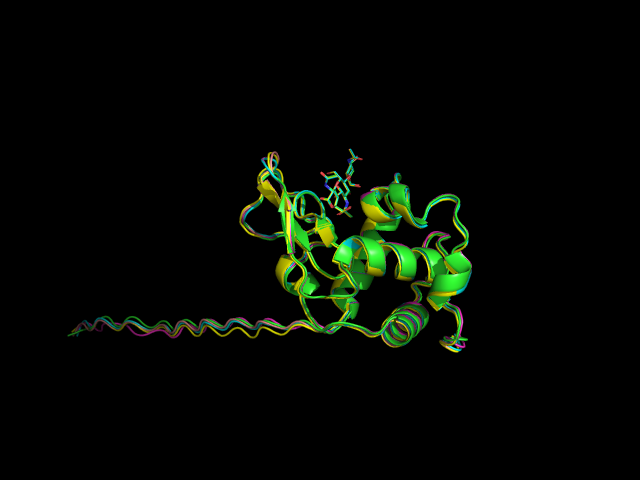
6. В результате получено 5 моделей: model1.pdb , model2.pdb , model3.pdb , model4.pdb , model5.pdb .
Изображение наложенных структур моделей:

Неструктурированный N-конец полученных моделей соответствует гэпам в выравнивании с белком-образцом. Все модели очень похожи.
7. Для проверки качества моделей воспользуемся WHATIF :
Судя по результатам Anomalous bond lengths и Anomalous bond angles 5-ая модель лучшая.