Обзор генома и протеома бактерии Streptomyces albidoflavus
Автор: Васильев Артем Михайлович, студент факультета биоинженерии и биоинформатики
РЕЗЮМЕ
В данном обзоре приведено описание и частичный анализ генома и протеома бактерии Streptomyces albidoflavus (S. albidoflavus). В работе обсуждаются общие характеристики генома, встречаемость различных типов генов и распределение 6-меров в нём, местоположение точек начала и терминации репликации геномной ДНК, а также случайность распределения кодирующих белки генов по её прямой и обратной цепи.
Ключевые слова: Streptomyces albidoflavus, геном, протеом, k-меры, микросателлиты, GC-skew, Excel.
ВВЕДЕНИЕ
Streptomyces albidoflavus – аэробная грамположительная бактерия. Популяции S. albidoflavus обнаружены в различных средах: в почве, солёных водоёмах, других организмах, в частности, в насекомых (1). Геном S. albidoflavus представлен единственной кольцевой хромосомой. Медианный размер генома оценивается в 7.05313 мегабаз (мегабаза = 106 пар нуклеотидов (п.н.)). Геном примечателен высоким GC-составом: медианный процент GC-пар составляет 73,4% (2), (3). Как и другие представители рода Streptomyces, S. albidoflavus обладает развитым вторичным метаболизмом, а именно, синтезирует фунгициды, эффективные против ряда видов плесеней и дрожжей (4).
В настоящей работе проведено исследование генома и протеома S. albidoflavus.
МАТЕРИАЛЫ И МЕТОДЫ
Обзор написан на основе данных о геноме и протеоме S. albidoflavus из базы данных genomes портала NCBI (5).
Для их обработки использовались возможности электронных таблиц (ЭТ) Microsoft Excel 2016. Все результаты, если не указано иначе, получены следующими методами ЭТ:
- выделение ячеек мышью и клавиатурой (в том числе с использованием Shift, Ctrl, Ctrl+A);
- переход на другую ячейку с помощью Enter, Tab, Shift+Enter и Shift+Tab;
- копирование и вставка (в том числе специальная) с помощью горячих клавиш Ctrl+C и Ctrl+V соответственно;
- импорт данных из файлов форматов txt и xlsx;
- сортировка данных по значениям;
- формулы: арифметические операции и функции SUM, PRODUCT, AVERAGE, COUNT, SUBSTITUTE, COLUMN, IFS, VLOOKUP, GETPIVOTDATA, CHISQ.INV.RT;
- фиксация формул с помощью символа $;
- распространение формул мышью и с помощью горячих клавиш Ctrl+R и Ctrl+D;
- фильтр и сортировка строк и столбцов;
- поиск и замена значений ячеек с помощью горячих клавиш Ctrl+F и Ctrl+H;
- создание и форматирование плоских таблиц;
- создание и форматирование сводных таблиц;
- создание и форматирование графиков.
Для вычисления количества 6-меров и нуклеотидов каждого типа в геноме использовалась программа wordcount пакета программного обеспечения emboss.
Для определения точек начала и терминации репликации геномной ДНК был использован сервис GenSkew (6).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Общие сведения о геноме
Размер генома S. albidoflavus составляет 7,170,504 пар нуклеотидов, в его состав входят только канонические 4 нуклеотида (A, T, G и C), при этом GC-состав генома составляет 73.34% (данные приведены на листе genome_size в сопроводительных материалах). Таким образом, полученные значения приблизительно равны медианным. Высокий GC-состав скорее всего нужен для увеличения устойчивости структуры двуспиральной ДНК при высоких температурах (например, в нагретой солнцем почве), так как в ДНК с высоким GC-составом больше водородных связей на пару нуклеотидов (в паре GC три, а в AT – две), что увеличивает устойчивость структуры двойной спирали.
Типы генов
Геном S. albidoflavus представлен семью типами генов, причём гены белков составляют подавляющее большинство – 96.52% от общего числа генов (данные приведены на листе genes_per_type в сопроводительных материалах). 3 типа РНК представлены в геноме всего одним геном на каждый. Вероятно они выполняют специфические функции в регуляции экспрессии генов.
Распределение 6-меров
На гистограмме (рис. 1) видно, что число наименее представленных 6-меров (первый столбец гистограммы) намного больше, чем число наиболее представленных (последний столбец).
На диаграмме (рис. 2) выявлено, что наиболее представленными 6-мерами являются GAAGAA, AAGAAG и комплементарные им 6-меры (диаграмма, на которой видно, какой 6-мер принадлежит каждому столбцу, приведена на листе 6-mer_bar_chart в сопроводительных материалах). Так как они перекрываются по четырём позициям, то их высокую представленность можно объяснить тем, что в геноме есть участки, состоящие из многократно повторяющихся последовательностей (в данном случае AAG) – микросателлиты.
Более того, на диаграмме выделяется 6 групп k-меров с близкими значениями cb и с близким количеством встреч в геномной ДНК, поэтому можно предположить наличие нескольких типов обильно представленных сателлитов в геноме S. albidoflavus (расчёт cb k-меров приведён на листе 6-mer_frequency в сопроводительных материалах).
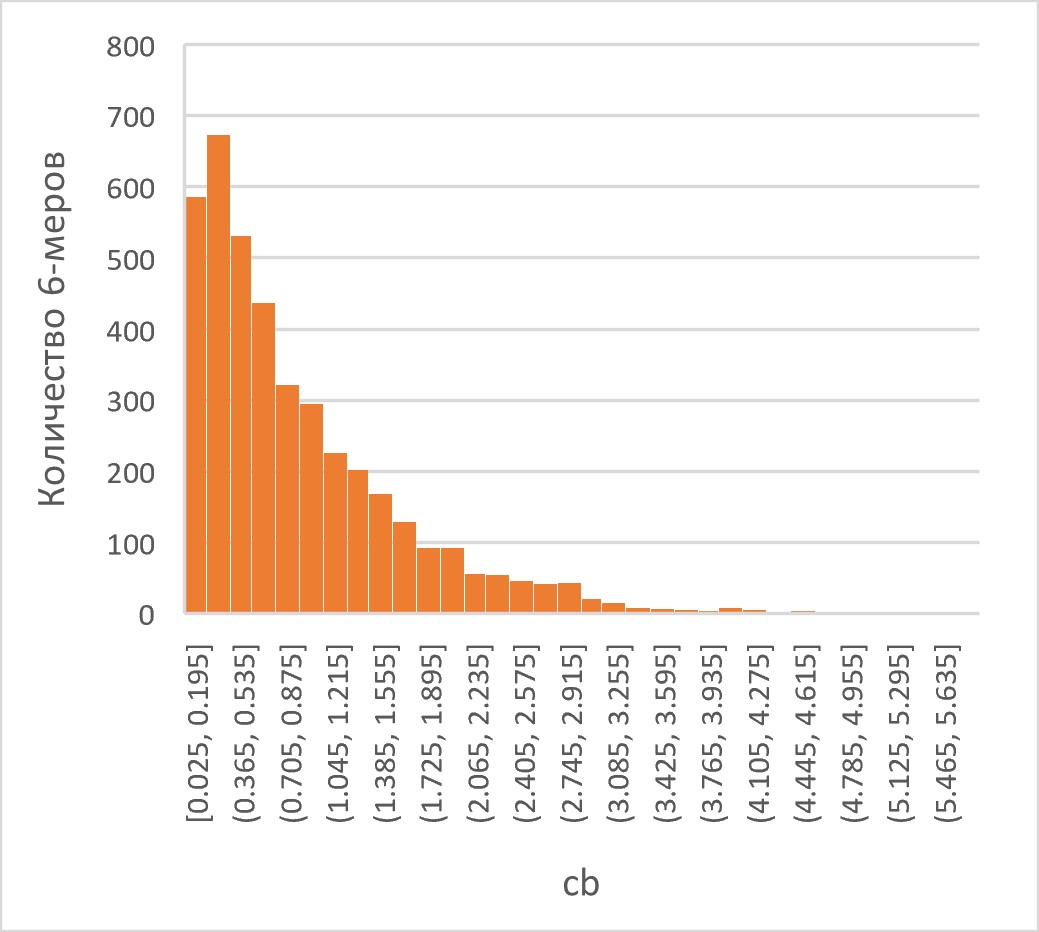
Рисунок 1. Гистограмма распределения cb 6-меров. 6-мер – подпоследовательность длины 6 в последовательности ДНК. cb – отношение реальной частоты встреч 6-мера в геноме к его предполагаемой частоте встреч.
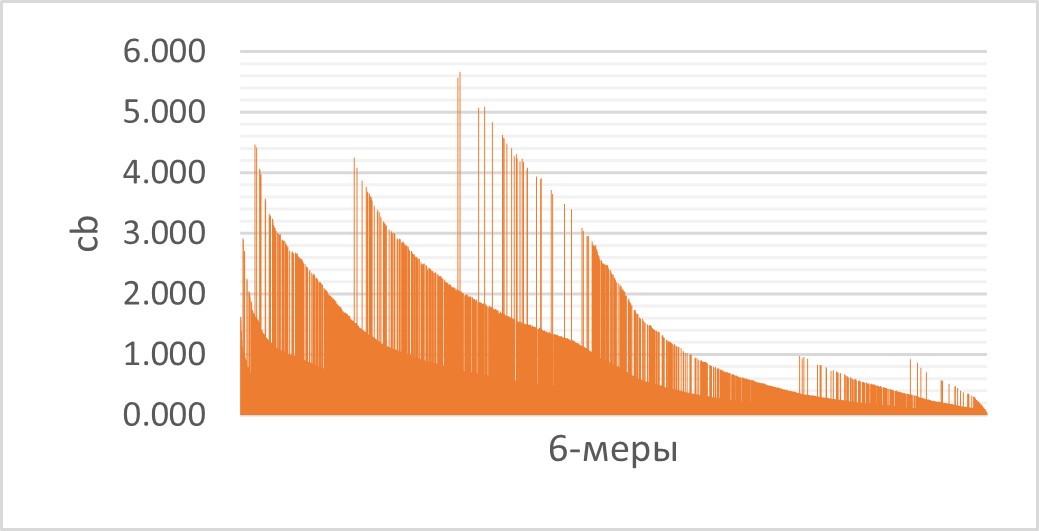
Рисунок 2. Диаграмма, отражающая cb каждого 6-мера. Каждый столбец соответствует одному 6-меру. 6-меры располжены по убыванию абсолютного числа встреч в геноме.
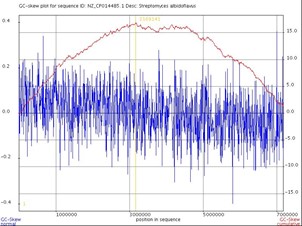
Места начала и терминации репликации
Известно, что в большинстве бактериальных геномов с одной стороны от точки начала репликации (oriC) число G на 1000 нуклеотидов меньше числа C на 1000 нуклеотидов (в среднем). А с другой стороны от oriC – наоборот. oriC соответствует минимуму GC-skew cumulative, а ter (терминатор) – максимуму (7). Соотношение G и C в данной точке определяется по формуле:
GC-skew normal = (G − C) / (G + C), где G – число G на 1000 нуклеотидов, а C – число C на 1000 нуклеотидов.
GC-skew cumulative равен сумме GC-skew normal во всех точках до данной включительно. Так как GC-skew normal на участке от oriC до ter в направлении лидирующей цепи положителен, а на участке от oriC отрицателен, то при движении от oriC к ter этом направлении GC-skew cumulative будет постоянно увеличиваться, а при движении от ter к oriC будет постоянно уменьшаться, поэтому oriC и ter будут соответствовать минимуму и максимуму GC-skew cumulative соответственно.
Определено, что координаты oriC находятся в окрестности нуля (рис. 3, в высоком разрешении приведён на листе origin в сопроводительных материалах). Это означает, что ислледуемая последовательность геномной ДНК начинается с oriC. Максимум GC-skew cumulative достигается в точке 3,169,141 п.н., однако на участке примерно от 3,0 до 3,8 мегабаз GC-skew cumulative находится на плато, то есть частота G равна частоте C, поэтому терминатор может находиться в окрестности любой точки на этом участке.
Подобная неточность может возникать из-за того, что по одной из гипотез неоднородность соотношения G и C по разные стороны от oriC объясняется тем, что у большинства бактерий большая часть белков кодируется на лидирующей цепи ДНК (обоснование гипотезы по ссылке) (8), (9). Однако у S. albidoflavus на лидирующей и отстающей цепи закодировано примерно одинаковое число белков (см. ниже), поэтому неоднородность соотношения G и C менее выражена. Поэтому результаты поиска ter оказались менее однозначными, чем ожидалось.
Распределение генов, кодирующих белки, по прямой и обратной цепи ДНК
У S. albidoflavus распределение генов белков по двум цепям геномной ДНК (табл. 1) случайно (подтверждение с помощью критерия хи-квадрат приведено на листе prtn_genes_distrib_analysis сопроводительных материалов). Данный результат противоречит приведённому выше утверждению о преимущественном расположении генов на прямой цепи ДНК. Следовательно, в геноме S. albidoflavus нерепарируемые мутации возникают чаще. Поэтому эта бактерия с меньшей вероятностью имеет сложные биохимические механизмы, легко повреждающиеся при возникновении мутаций. Эта гипотеза противоречит наличию у S. albidoflavus развитого вторичного метаболизму, но подтверждает её низкую специфичность по отношению к среде обитания.
| Count | Strand | |
|---|---|---|
| Gene_type | + | − |
| protein_coding | 3040 | 2971 |
| pseudogene | 60 | 65 |
| tRNA | 35 | 33 |
| rRNA | 9 | 12 |
| SRP_RNA | 1 | 0 |
| RNase_P_RNA | 1 | 0 |
| tmRNA | 1 | 0 |
Таблица 1. Распределение генов по прямой (+) и обратной (-) цепи геномной ДНК.
ЗАКЛЮЧЕНИЕ
В результате частичного анализа генома и протеома S. albidoflavus получены схожие данные с данными предыдущих исследований этой бактерии, а также выявлены интересные особенности генома, на основе которых была выдвинута гипотеза о существовании в геноме множества микросателлитов нескольких типов, а также гипотезы о наличии связи между случайным распределением генов белков по двум цепям ДНК и а) равными частотами G и C на её участке; б) низкой специфичностью бактерии по отношению к среде обитания. Для окончательного подверждения данных гипотез необходимо в дальнейшем найти для них экспериментальные или дополнительные косвенные подтверждения.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Таблица Vasiliev_supple_fin с информацией о геноме и протеоме S. albidoflavus:
- Лист genome_size с размером генома, его GC-составом и частотой нуклеотидов в нём;
- Лист genes_per_type с количеством генов различного типа в геноме;
- Лист 6-mer_frequency с частотой 6-меров в геноме;
- Лист 6-mer_bar_chart с диаграммой cb каждого 6-мера;
- Лист origin с графиком GC-skew;
- Лист prtn_genes_distrib_analysis с оценкой случайности распределения генов по + и − цепям ДНК.
БЛАГОДАРНОСТИ
Автор благодарит учебную часть факультета биоинженерии и биоинформатики за создание условий для качественного дистанционного обучения, способствовавшего развитию необходимых для написания этого обзора умений, Залевского А.О., а также преподавателей факультета биоинженерии и биоинформатики за формирование у автора навыков оформления научных работ.
СПИСОК ЛИТЕРАТУРЫ
- Cheng K, Rong X, Pinto-Tomás AA, Fernández-Villalobos M, Murillo-Cruz C., Huang Y. Genetic Analysis of Streptomyces albidoflavus Reveals Habitat Barriers to Homologous Recombination in the Diversification of Streptomycetes. Appl Environ Microbiol. 2015 Feb; 81(3):966975. doi: 10.1128/AEM.02925-14.
- Pylro VS, Dias ACF, Andreote FD, Varani AM, Andreote CCF, Ribeiro IAF, Kitano IT, Bernardo ERA. Draft Genomic Sequences of Streptomyces misionensis ACT66 and Streptomyces albidoflavus ACT77, Bacteria with Potential Application for Phytopathogen Biocontrol. Microbiol Resour Announc. 2019 Sep 5;8(36):e00949-19. doi: 10.1128/MRA.00949-19.
- Страница Streptomyces albidoflavus в базе данных genome портала NCBI.
- Augustine SK; Bhavsar SP; Kapadnis BP. A non-polyene antifungal antibiotic from Streptomyces albidoflavus PU 23. Journal of Biosciences 01 Mar 2005, 30(2):201-21. doi: 10.1007/bf02703700.
- Источник с данными о геноме и протеоме Streptomyces albidoflavus.
- Сервис, определяющий неоднородность соотношения нуклеотидов двух типов на одной цепочке ДНК.
- Ссылка на страницу с информацией о неоднородности соотношения G и C в геномной ДНК.
- Страница Википедии с гипотезами возникновения неоднородности соотношения G и C в геномной ДНК.
- Chen X, Zhang J. Why are genes encoded on the lagging strand of the bacterial genome? Genome Biol Evol. 2013;5(12):2436-9. doi: 10.1093/gbe/evt193.